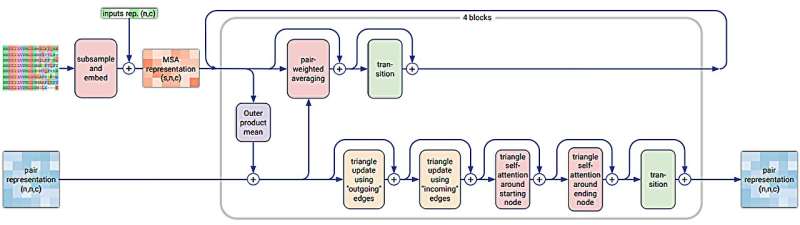
Un equipo combinado de investigadores médicos y especialistas en sistemas de inteligencia artificial del proyecto Deep Mind de Google e Isomorphic Labs, ambos en Londres, ha realizado lo que el grupo describe como mejoras sustanciales en AlphaFold 2 que hacen posible que la aplicación prediga la estructura de una amplia variedad. de sistemas biomoleculares de forma más amplia y precisa. La nueva iteración se llama AlphaFold 3.
En su estudio publicado en la revista Nature , el grupo utilizó técnicas de difusión para realizar mejoras en el modelo arquitectónico subyacente de la aplicación para permitir hacer predicciones más generales.
La primera versión del sistema de inteligencia artificial AlphaFold basado en aprendizaje profundo se lanzó hace apenas cuatro años y fue aclamada por su capacidad para hacer predicciones precisas sobre la estructura de las proteínas utilizando secuencias de aminoácidos. También ha ayudado a los investigadores a comprender mejor cómo funcionan las proteínas. AlphaFold 2 se basó en dichas capacidades, ampliando los complejos que podían predecirse.
En esta nueva iteración, el equipo de investigación ha dotado a la aplicación de la capacidad de predecir sistemas biomoleculares más allá de las proteínas. Puede predecir ligandos, por ejemplo, o estructuras de ARN o ADN. Señalan que incluso puede hacer predicciones sobre la estructura de iones, ácidos nucleicos, otras proteínas e interacciones entre anticuerpos y antígenos.
Estas capacidades, señalan los investigadores, lo convierten en una herramienta útil para el descubrimiento de nuevos fármacos. Una empresa de descubrimiento de fármacos (y una filial de DeepMind) ya está utilizando el nuevo sistema para hacer precisamente eso.
Además de hacer predicciones sobre otras estructuras biomoleculares, el equipo de investigación afirma que AlphaFold 3 también es mucho más preciso que sus iteraciones anteriores y sus competidores. Pero también reconocen que hay margen para crecer:AlphaFold 3 tiene una tasa de error de quiralidad del 4,4%, por ejemplo. A veces también produce alucinaciones, lo que reduce la apariencia de las cintas.
Señalan que el trabajo continuará con el sistema AlphaFold mientras el equipo busca mejorar la precisión y agregar más tipos de sistemas a los que se pueda aplicar. También planean introducir una estructura de clasificación para ayudar a los usuarios a emitir juicios sobre los resultados proporcionados por el sistema.
Más información: Josh Abramson et al, Predicción estructural precisa de interacciones biomoleculares con AlphaFold 3, Naturaleza (2024). DOI:10.1038/s41586-024-07487-w
Información de la revista: Naturaleza
© 2024 Red Ciencia X