El núcleo de cada una de tus células contiene toda la información genética (el genoma) necesaria para construir cada tipo de célula y proteína en todo tu cuerpo. Como una biblioteca compleja en un espacio diminuto 50 veces más pequeño que el ancho de un cabello humano, los genes están organizados en regiones precisas en tres dimensiones.
Además del genoma, el núcleo contiene estructuras llamadas cuerpos nucleares que contienen altas concentraciones de proteínas y ácidos nucleicos específicos. Sin embargo, el papel de los cuerpos nucleares ha sido un misterio durante casi un siglo. Un nuevo estudio del laboratorio de Mitch Guttman, profesor de biología de Caltech, muestra que estos cuerpos nucleares pueden actuar como fábricas en miniatura para permitir la producción eficiente de ARNm.
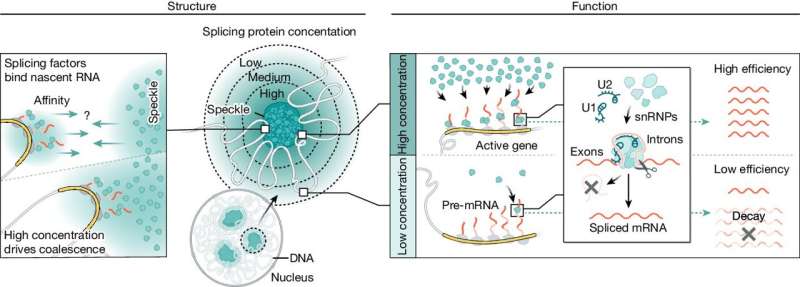
El empalme del ARNm es una parte fundamental del proceso que se produce cuando las instrucciones codificadas del ADN se convierten en una proteína funcional. Después de que un gen se transcribe de ADN a ARN, se deben eliminar secciones extrañas (llamadas intrones), un proceso facilitado por enzimas de empalme.
En el nuevo estudio, los investigadores de Caltech examinaron un tipo particular de cuerpo nuclear, llamado mota nuclear, que contiene altas concentraciones de enzimas de empalme. El equipo de Guttman descubrió que el genoma se desplaza físicamente de modo que los genes altamente transcritos estén muy cerca de las motas, lo que permite un empalme más eficiente.
Los hallazgos tienen implicaciones tanto para la comprensión científica fundamental de la relación entre la estructura nuclear y la función celular como, en última instancia, para el desarrollo futuro de terapias para enfermedades en las que el empalme del ARNm se ve afectado, incluidas las enfermedades neurodegenerativas como la ELA.
El artículo que describe los hallazgos fue publicado en la revista Nature. el 8 de mayo. Se titula "La organización del genoma alrededor de las motas nucleares impulsa la eficiencia del empalme del ARNm".
Durante una década, los investigadores del laboratorio Guttman han estudiado cómo está organizado espacialmente el núcleo; en otras palabras, la distribución de la biblioteca. Las estructuras tridimensionales en las que se ordena el ADN hacen que ciertos genes sean más o menos accesibles a la maquinaria que convierte el ADN en ARNm, y el nuevo estudio muestra que la estructura física del genoma vincula la transcripción con el proceso de empalme.
Una célula muscular, por ejemplo, cambiará su genoma de modo que los genes altamente transcritos para la actividad muscular estén físicamente cerca de las motas nucleares, donde las altas concentraciones de enzimas de empalme hacen que el empalme del ARN sea particularmente eficiente. Una célula neuronal, por otro lado, reorientará su genoma en el espacio de modo que los genes necesarios para producir células especializadas para la función neurológica estén más cerca de las motas.
"Sabemos que el empalme depende de las enzimas, y cualquier mutación que altere la concentración de enzimas tiene efectos dramáticos", dice Prashant Bhat, investigador postdoctoral asociado en biología e ingeniería biológica y primer autor del estudio.
"Anteriormente, tendíamos a pensar en el empalme como una constante:se crea un pre-ARNm; se lo empalma. Ahora sabemos que la organización física del núcleo marca una gran diferencia en la eficiencia del empalme y, por extensión, en otros procesos. ."
Muchas enfermedades, incluidos los trastornos neurodegenerativos y el cáncer, tienen una característica común:el empalme disfuncional del ARN. Si bien no se sabe si el empalme inadecuado es la causa principal de la enfermedad o un efecto, en última instancia, el proceso de empalme es un objetivo prometedor para la terapéutica.
Por ejemplo, las enfermedades en las que se produce una cantidad inadecuada de proteínas (conocidas como enfermedades por haploinsuficiencia) podrían tratarse reposicionando la copia normal del gen de esa proteína cerca de las motas nucleares para estimular el empalme y restaurar niveles saludables de proteínas.
Más información: Prashant Bhat et al, La organización del genoma alrededor de motas nucleares impulsa la eficiencia del empalme de ARNm, Naturaleza (2024). DOI:10.1038/s41586-024-07429-6
Información de la revista: Naturaleza
Proporcionado por el Instituto de Tecnología de California