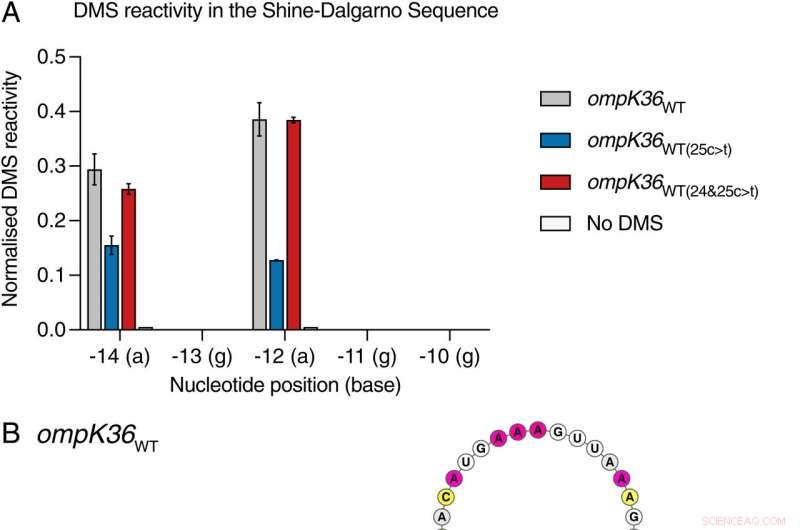
Posición 25 en el ompK36WT(25c> t) El ARN induce un tallo que involucra al SDS. (A) Señal de DMS normalizada lado a lado por nucleótido en el SDS de ompK36WT transcrito in vitro y replegado de longitud completa , ompK36WT(25c> t) , ompk36WT(24&25c> t) y ompK36WT sin tratar con DMS . Los valores más altos corresponden a una mayor accesibilidad base. Se muestra la señal DMS (± SD) de 2 repeticiones biológicas para los nucleótidos -14a a -10g. (B–D). Modelos de estructura restringida por DMS del extremo 5 'de ompK36WT (B), ompK36WT(24&25c> t) (C) y ompK36WT(25c> t) (D). Los nucleótidos están coloreados por la señal DMS normalizada. Los SDS en las estructuras de ARN están resaltados en gris. Las flechas indican la posición −14 y la posición 25c> t en ompK36WT(25c> t) . Crédito:Actas de la Academia Nacional de Ciencias (2022). DOI:10.1073/pnas.2203593119
Los investigadores han descubierto una nueva forma en que las infecciones adquiridas en hospitales resisten a los antibióticos, a través de una mutación genética "silenciosa".
Las bacterias pueden adquirir resistencia a los antibióticos a través de mutaciones aleatorias en su ADN que les proporcionan una ventaja que les ayuda a sobrevivir. Encontrar mutaciones genéticas y descubrir cómo ayudan a las bacterias a sobrevivir al ataque de los antibióticos es clave para ayudarnos a luchar con nuevos medicamentos.
Los investigadores ahora han descubierto una mutación "silenciosa" en el código genético que conduce a la resistencia a los antibióticos. Por lo general, las mutaciones de este tipo se pasarían por alto y es posible que ya estén presentes en otras bacterias infecciosas.
El equipo, dirigido por investigadores del Imperial College London y que incluye a colaboradores internacionales, publicó hoy sus resultados en la revista Proceedings of the National Academy of Sciences. .
Resistencia creciente
Los investigadores observaron la bacteria Klebsiella pneumoniae, que causa infecciones en los pulmones, la sangre y las heridas de las personas en los hospitales, y los pacientes que tienen sistemas inmunitarios comprometidos, como los que se encuentran en unidades de cuidados intensivos, son especialmente vulnerables.
Al igual que muchas bacterias, K. pneumoniae se está volviendo cada vez más resistente a los antibióticos, en particular a una familia de medicamentos llamados carbapenémicos. Estos importantes medicamentos de último recurso se usan en hospitales cuando otros antibióticos ya han fallado.
Dado que el aumento de la resistencia a los carbapenémicos podría afectar drásticamente nuestra capacidad para tratar infecciones, la K. pneumoniae resistente a los carbapenémicos se clasifica como organismo "crítico" de Prioridad 1 de la Organización Mundial de la Salud.
Para ser efectivos, los antibióticos deben penetrar en las bacterias, y en K. pneumoniae esto sucede a través de un canal en la membrana externa de la bacteria, formado por una proteína llamada OmpK36. El equipo descubrió una mutación genética que hace que las bacterias produzcan menos proteína, cerrando efectivamente algunos de estos canales y evitando que entren los antibióticos carbapenémicos.
Mutaciones 'silenciosas'
Sin embargo, esta mutación funciona de manera diferente a las mutaciones estándar que resultan en resistencia a los antibióticos. Por lo general, las mutaciones cambian el código genético de modo que cuando los ribosomas lo "leen" y lo convierten en una proteína, produce una cadena diferente de aminoácidos con funciones diferentes.
Esta mutación todavía produce la misma cadena de aminoácidos, pero altera la estructura de un intermediario de ARNm importante, evitando que los ribosomas lean el código y produzcan proteínas a partir de él.
Cuando se buscan mutaciones, las técnicas genómicas suelen buscar cambios en la secuencia de aminoácidos. Sin embargo, dado que esta mutación altera una estructura, en lugar de la secuencia en sí, podría considerarse una mutación "silenciosa".
El primer autor, el Dr. Joshua Wong, del Departamento de Ciencias de la Vida de Imperial, dijo:"En la era de los grandes datos y la genómica, las mutaciones como las que hemos descubierto pueden considerarse 'silenciosas', ya que el código genético da como resultado la misma secuencia de proteínas .
"Este descubrimiento debería cambiar la forma en que vemos el código genético de las bacterias y potencialmente indica que nosotros, en la comunidad científica, hemos pasado por alto otras mutaciones similares que pueden tener efectos importantes. Nuestro trabajo se centra en una sola mutación, pero cambia fundamentalmente la forma en que interpretamos las mutaciones, especialmente aquellas que se pensaba que eran silenciosos".
Impulsado por el uso de antibióticos
El equipo de Imperial, que caracterizó la mutación, trabajó con equipos de la Universidad de Oxford, la Universidad de Florencia y la Universidad de Harvard para identificar la distribución de la mutación a nivel mundial, evaluar los niveles de resistencia y determinar cómo afectó la mutación al ARNm intermedio. estructura.
Usando datos de muestras de bacterias resistentes recolectadas en todo el mundo, el equipo mostró que la mutación había surgido varias veces de forma independiente. Esto sugiere que no es aleatorio, sino que se debe a la necesidad de la bacteria de defenderse de los antibióticos.
El investigador principal, el profesor Gad Frankel, del Departamento de Ciencias de la Vida de Imperial, dijo:"La mutación evolucionó en varias ocasiones de forma independiente, y esto nos dice que este mecanismo novedoso no es una casualidad única, sino impulsado por el consumo de antibióticos. Esto sugiere que la mutación ocurre bajo la presión de los antibióticos y destaca los efectos secundarios del uso excesivo de antibióticos en hospitales y otros entornos".
El equipo ahora espera que su hallazgo se incorpore a herramientas bioinformáticas que analizan secuencias genéticas para identificar la presencia de la mutación, como se hizo con un mecanismo anterior que descubrió el equipo.
También seguirán trabajando con sus colaboradores para buscar otras mutaciones importantes en este patógeno clave. La superbacteria común resistente a los medicamentos desarrolla una rápida resistencia al antibiótico de "último recurso"