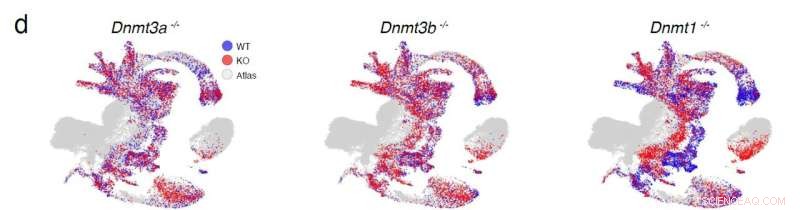
Mapeo de células de las líneas de ratones knock-out de DNMT al atlas de referencia (gris) con las células de tipo salvaje indicadas en azul y las células knock-out indicadas en rojo. Crédito:Instituto Babraham
La investigación que aplicó técnicas de análisis de células individuales y comparaciones con un recurso de atlas de células permitió a los investigadores del Instituto Babraham conectar los defectos de desarrollo observados causados por procesos de metilación del ADN interrumpidos con una comprensión de los tipos de células afectadas. Este trabajo, publicado en Genome Biology , se basa en trabajos previos realizados con colaboradores para establecer un atlas celular detallado que traza el destino celular a través del desarrollo temprano. Este trabajo aporta nuevos conocimientos importantes para comprender el papel de la metilación del ADN durante la embriogénesis, ayudando a descifrar las reglas que rigen cómo surgen los diferentes tipos de células. La comprensión de estas reglas será esencial para que los investigadores puedan dirigir con precisión y seguridad el destino de las células para producir tipos de células clínicamente relevantes para la medicina regenerativa.
La investigación del Reik labat del Instituto Babraham ha mejorado nuestra comprensión del papel de la metilación del ADN durante las primeras etapas de desarrollo. Los avances tecnológicos en este campo, que brindan la capacidad de recopilar tipos de datos paralelos de una sola célula y la existencia de referencias de atlas de células y conjuntos de datos completos, están revolucionando lo que sabemos sobre los procesos que determinan el destino de las células. Esclarecer las reglas de cómo se forman los diferentes tipos de células tiene aplicaciones en la medicina regenerativa, así como en la comprensión de enfermedades y trastornos del desarrollo.
Se sabe que el borrado y la reintroducción de la metilación del ADN son vitales para establecer la identidad celular a medida que se forman los tejidos y órganos del embrión. La eliminación de enzimas de metilación clave en ratones provoca graves defectos de desarrollo y, en algunos casos, la letalidad del embrión. A pesar de la importancia de la metilación del ADN en el desarrollo, los mecanismos subyacentes de cómo se logra esto son poco conocidos. Esto se debe a las limitaciones en la información que los investigadores podían recopilar previamente para comprender los efectos de los cambios en los procesos habituales de metilación del ADN en el desarrollo, que se restringía al análisis de defectos de desarrollo, imágenes de muestras y análisis limitados de todo el genoma utilizando muestras a granel. Estos métodos no fueron suficientes para resolver los efectos a nivel de diferentes tipos de células.
Usando ratones en los que se eliminaron las enzimas de metilación clave, los investigadores del laboratorio Reik en el programa de Epigenética del Instituto realizaron un análisis de expresión génica de una sola célula al comienzo del desarrollo del órgano, que ocurre el día 8.5 después de la fertilización. Utilizando el poder de los enfoques de una sola célula, los investigadores pudieron seguir qué tipos de células se vieron afectadas, en términos de no poder formarse en el embrión de ratón, lo que sugiere los mecanismos detrás de los efectos observados a escala de todo el organismo.
La investigación utilizó ratones modificados genéticamente en los que se eliminaron dos grupos clave de enzimas de metilación:líneas de ratones knock-out en las que se eliminaron individualmente las metiltransferasas de ADN (DNMT 1, 3a y 3b) que introducen y mantienen la metilación del ADN, y un sistema para investigar los efectos de una eliminación combinada de las tres enzimas TET (translocación diez-once (TET) metilcitosina dioxigenasas) 1/2/3), que causan la desmetilación.
El Dr. Stephen Clark, investigador sénior en el laboratorio de Reik cuando se llevó a cabo esta investigación, dijo:"El uso de enfoques unicelulares realmente proporciona la resolución que necesitamos para estudiar la mecánica de la metilación del ADN durante el desarrollo. La imagen que pudimos para acumular confirma el papel represivo de la metilación del ADN en este punto de desarrollo, en primer lugar, que se requiere mantener la metilación correcta del ADN para suprimir las identidades de tipos de células pasadas y alternativas, y en segundo lugar, que la metilación del ADN debe eliminarse de partes del genoma para permitir que ciertas células tipos a formar."
Se utilizó una técnica llamada secuenciación de ARN de una sola célula para medir la expresión génica en todo el genoma en cada línea de ratón. La comparación de estos perfiles de expresión con un conjunto de datos de referencia permitió identificar todos los tipos de células del embrión. Después de ese paso, el efecto de las perturbaciones de la metilación en el destino celular podría evaluarse comparando la composición de los embriones knock-out (donde se eliminaron las enzimas de metilación) con los embriones de tipo salvaje en la misma etapa de desarrollo para resaltar las diferencias en el tipo de célula. proporciones.
Los investigadores pudieron correlacionar los efectos sobre la formación de tipos de células en el día 8.5 de desarrollo que coincidían con los fenotipos observados y analizar los cambios específicos de tipos de células en la expresión génica que podrían estar relacionados con defectos en el compromiso del destino celular.
El Dr. Ricard Argelaguet, exinvestigador postdoctoral en el laboratorio Reik del Instituto y coautor del artículo, dijo:"La capacidad de tener la perspectiva de todo el organismo y la granularidad de observar cambios en los tipos de células y la expresión génica nos ha brindado la capacidad de separar el papel de la metilación y desmetilación del ADN en el embrión en desarrollo en este punto de tiempo particular para crear nuevos conocimientos. Será igualmente interesante aplicar este enfoque a puntos de tiempo posteriores para comprender más sobre el papel de la metilación del ADN. a medida que avanza el desarrollo".
La investigación ha creado una plataforma de datos interactivos que proporciona lecturas de expresión génica a nivel de una sola célula de embriones de ratones mutantes Dnmt y Tet.
El profesor Wolf Reik, director del Instituto de Ciencias Altos Cambridge, quien dirigió la investigación mientras era líder de grupo en el programa de Epigenética en el Instituto Babraham, dijo:"Esta investigación proporciona un recurso valioso para investigar la conexión entre la metilación del ADN y el establecimiento de destino celular Esta investigación se benefició de conjuntos de datos publicados y atlas de referencia y esperamos que, a su vez, nuestro trabajo sea útil para otros investigadores tanto en el campo del desarrollo como en el de la epigenética". Nuevo método potencia el estudio de la regulación de la actividad génica