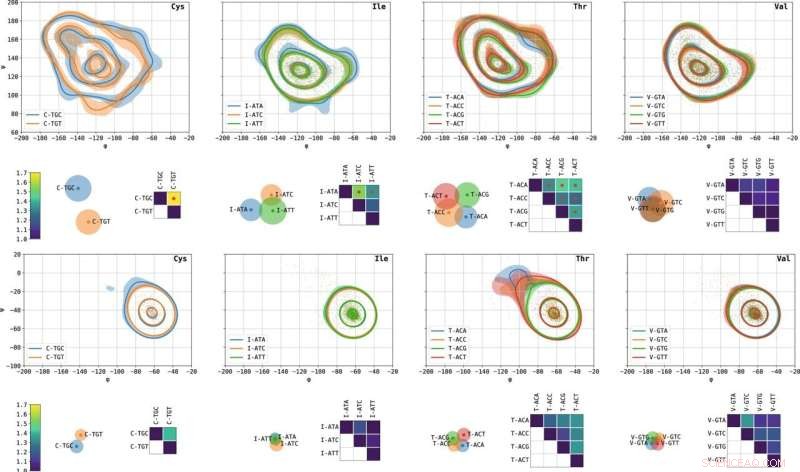
Gráficos de Ramachandran específicos de codón de aminoácidos seleccionados y distancias entre ellos. De izquierda a derecha se muestran cisteína, isoleucina, treonina y valina. Los gráficos de contorno representan las líneas de nivel que contienen el 10, 50 y 90% de la masa de probabilidad. Las regiones sombreadas representan intervalos de confianza del 10 % al 90 % calculados en 1000 arranques aleatorios. Se representan los modos β- (superior) y α- (inferior). Las matrices muestran L1 distancias entre pares de gráficos de Ramachandran específicos de codón, normalizados para que la distancia propia sea 1. Los puntos rojos indican pares con distribuciones de ángulo diedro significativamente diferentes en función de su valor p. Los gráficos de dispersión que visualizan las matrices de distancia se obtuvieron mediante una variante de escalamiento multidimensional (MDS). Cada punto representa un codón; Las distancias euclidianas por pares entre los puntos se aproximan a L1 distancia entre los codones correspondientes. Los círculos aproximan los radios de incertidumbre. Cuanto más se superponen dos círculos, menos distinguibles son los gráficos de Ramachandran específicos de codones correspondientes. Crédito:Comunicaciones de la naturaleza (2022). DOI:10.1038/s41467-022-30390-9
Un estudio que integra ideas biológicas y nuevas herramientas informáticas ha descubierto asociaciones novedosas entre la codificación genética y la estructura de las proteínas, lo que podría cambiar potencialmente la forma en que pensamos sobre la producción de proteínas en el ribosoma, la "línea de ensamblaje de proteínas" de la célula. La investigación, encabezada por el profesor Alex Bronstein, la Dra. Ailie Marx y Ph.D. estudiante Aviv Rosenberg, fue publicado en Nature Communications .
Las proteínas, las moléculas complejas que desempeñan funciones críticas en prácticamente todos los mecanismos biológicos, son producidas por los ribosomas en un proceso llamado traducción. El ribosoma decodifica las "instrucciones genéticas" entrantes para sintetizar cadenas de aminoácidos, los componentes básicos de las proteínas. Cuando los aminoácidos se unen secuencialmente en una cadena larga, se pliegan en una estructura tridimensional única que otorga a la proteína sus propiedades biológicas y su funcionalidad. Los errores de traducción pueden dar lugar a errores de plegado y, posteriormente, a trastornos fisiológicos, tanto leves como graves.
Las instrucciones de producción de proteínas se entregan al ribosoma como codones, secuencias de tres "letras" del código de nucleótidos genéticos, que especifican la identidad y el orden de los aminoácidos que el ribosoma agregará a la cadena de proteínas. Por ejemplo, el codón UUU indica la adición del aminoácido fenilalanina, mientras que el codón UAC indica la adición de tirosina. De esta forma, la secuencia de codones codifica la secuencia única de aminoácidos característica de cada proteína. Este mapeo de codones genéticos a aminoácidos utilizados en la traducción es común a todas las criaturas vivientes del planeta y se considera un mecanismo primitivo.
Como si todo esto no fuera lo suficientemente complicado, es importante señalar que hay 61 codones que se decodifican en solo 20 aminoácidos. En otras palabras, todos menos dos aminoácidos están codificados por múltiples codones.
Aquí es donde la presente investigación entra en escena. Basado en experimentos llevados a cabo en las décadas de 1960 y 1970, el dogma aceptado establece que las proteínas no tienen "memoria" del codón específico a partir del cual se tradujo cada aminoácido, siempre que la identidad del aminoácido permanezca sin cambios. Estos primeros experimentos sobre el plegamiento de proteínas utilizaron desnaturalizantes químicos para desplegar proteínas completamente formadas y luego demostraron que al eliminar estos químicos, la cadena de proteínas podía replegarse espontáneamente para recuperar su estructura y función originales. Estos experimentos sugirieron que solo la secuencia de aminoácidos, y no la secuencia específica de codones, determina la estructura de una proteína. En vista de este dogma, las mutaciones que cambian la codificación genética sin cambiar el aminoácido se denominan ampliamente como "silenciosas" y se consideran intrascendentes para la estructura y función de la proteína.
El equipo de investigación de Technion ha descubierto una asociación entre la identidad del codón y la estructura local de la proteína traducida, lo que sugiere que este puede no ser el caso general y que las proteínas pueden "recordar" las instrucciones específicas a partir de las cuales fueron sintetizadas. El equipo de investigación analizó miles de estructuras de proteínas tridimensionales utilizando herramientas dedicadas que desarrollaron, que integran métodos informáticos avanzados, aprendizaje automático y estadísticas. De esta forma, compararon con precisión las distribuciones de los ángulos formados en estas estructuras bajo diferentes códigos genéticos sinónimos. Sus hallazgos muestran que para ciertos codones, existe una dependencia estadística significativa entre la identidad del codón y la estructura local de la proteína, en la posición del aminoácido codificado por ese codón.
Los investigadores enfatizan que los hallazgos aún no pueden arrojar luz sobre la dirección de la relación causal, lo que significa que aún no es posible decir si un cambio en la codificación genética puede causar un cambio en la estructura de la proteína local o si los cambios estructurales pueden causar codificación diferente, por ejemplo a través de procesos evolutivos. Esta pregunta es la base para un estudio de investigación posterior que ahora está llevando a cabo el grupo. According to Dr. Marx, a biologist by training and education, "If we find in subsequent research that the codon indeed has a causal effect on protein folding, this is likely to have a huge impact on our understanding of protein folding, as well as on future applications, such as engineering new proteins."
Dr. Marx emphasizes that the discovery presented in the article would not have been possible without Prof. Bronstein's computer and analysis skills. "This research is truly interdisciplinary, because biology alone cannot cope with such vast quantities of data without the help of data science, and computer scientists cannot themselves perform research of this kind, since they lack familiarity with the complex biological processes being probed. Therefore, our research highlights the huge advantage of interdisciplinary research that integrates skills from different fields to create a whole that is greater than the sum of its parts."