Un grupo de investigación ha construido herramientas genéticas versátiles para Saccharolobus islandicus REY15A, uno de los pocos modelos de arqueas para la investigación en biología de arqueas y biología CRISPR.
Dichas herramientas incluyen la edición eficiente del genoma, sistemas robustos de expresión de proteínas, ensayos de plásmidos de interferencia, silenciamiento de genes y edición de genes basada en CRISPR. Sin embargo, los vectores plasmídicos construidos hasta ahora para esta crenarchaeon se basan únicamente en el plásmido críptico pRN2.
El estudio, que aparece en mLife , fue dirigido por el Prof. Qunxin She y el Dr. Guanhua Yuan, ambos de la Universidad de Shandong en Qingdao, China.
"Se necesita un sistema dual huésped-vector para enriquecer la caja de herramientas genéticas de este arqueón modelo", afirma la profesora She.
De hecho, en una etapa temprana del desarrollo del vector de arqueas, tanto los plásmidos pRN1 como pRN2 que coexisten en Sa. islandicus REN1H1 se emplearon para construir vectores lanzadera para Sa. islandicus REY15A basado en estos dos plásmidos; Los plásmidos derivados de pRN2 obtuvieron una alta tasa de transformación y produjeron transformantes verdaderos, mientras que los vectores basados en pRN1 produjeron sólo muy pocas colonias en las que aparentemente los plásmidos estaban ausentes.
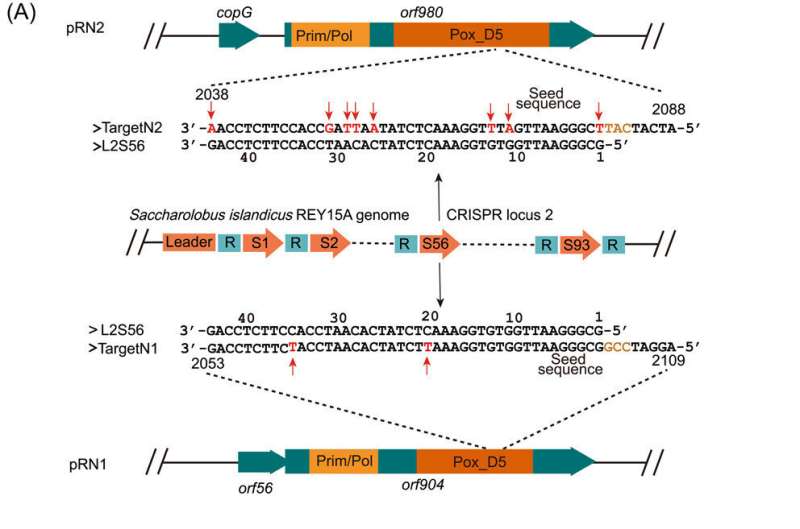
"Dado que las matrices CRISPR a menudo llevan espaciadores que coinciden con varios plásmidos en Sulfolobales, sospechamos que el genoma de Sa. islandicus REY15A podría llevar un espaciador que coincida con una secuencia en pRN1 pero no en pRN2", dice el Dr. Yuan.
Después de determinar la secuencia completa del genoma de Sa. islandicus REY15A4, el genoma del huésped lleva un espaciador (L2S56) que solo muestra dos errores de coincidencia con un segmento de ADN (objetivo N1) en la secuencia codificante del gen de la replicasa pRN1. Los experimentos de eficiencia de transformación demostraron que los ARNcr L2S56 se expresaron a un nivel que podría ser suficiente para provocar la inmunidad I-A pero insuficiente para desencadenar la inmunidad III-B para la eliminación del plásmido en Sa. islandicus REY15A.
Para obtener un derivado funcional de N1 objetivo que evade la inmunidad CRISPR del huésped, el equipo diseñó tres segmentos de ADN (N1a, N1b y N1c) basados en el objetivo pRN1, mientras que las mutaciones diseñadas en N1a eran sinónimas, N1b y N1c tenían mutaciones sin sentido. Los resultados mostraron que ninguno de los tres objetivos mutados fue el objetivo del sistema CRISPR en el huésped arqueal. Sin embargo, experimentos posteriores demostraron que N1c porta mutaciones sin sentido que pueden haber inactivado la proteína de replicación.
Mediante la construcción de una serie de vectores, el Saccharolobus–E. coli pN1dAA con las mutaciones N1a, el marcador de selección argD, el origen de replicación p15A y un marcador resistente a kanamicina se diseñaron basándose en la columna vertebral de pRN1, que puede lograr una coexistencia estable con el plásmido pSeSD derivado de pRN2 en Sa. células islandicus REY15A. Esto produjo un sistema de plásmido dual para el estudio genético con este importante modelo de arquea.
Dado que el examen de los conflictos huésped-plasmido proporciona un medio útil para la identificación de sistemas plásmido-vector compatibles, una vez que el conflicto se resuelve experimentalmente, los plásmidos diseñados son muy útiles para desarrollar sistemas huésped-vector, como se informa en este artículo.
Más información: Pengpeng Zhao et al, Diseño racional de derivados de pRN1 no restringidos y su aplicación en la construcción de un sistema de vector plasmídico dual para Saccharolobus islandicus, mLife (2024). DOI:10.1002/mlf2.12107
Proporcionado por Tsinghua University Press