Primulina hunanensis es una hierba perenne del género Primulina Hance de la familia Gesneriaceae. Es muy adaptable a ambientes de cuevas áridas y con poca luz, y es único en la evolución de su especie y adaptación ambiental. P. hunanensis tiene hermosas flores y una forma elegante que la hace valiosa para el cultivo hortícola.
Sin embargo, su hábitat es único y su rango de distribución es extremadamente estrecho, y se encuentra en un solo condado de la provincia de Hunan. Se estima que la población silvestre es de menos de 800 plantas y está clasificada como una planta en peligro de extinción, lo que representa una población muy pequeña de plantas silvestres en Hunan.
La investigación del genoma mitocondrial (ADNmt) también se ha aplicado a la conservación de plantas y la mejora genética. En Gesneriaceae, se ha informado de ADNmt de sólo dos especies.
Investigadores del Jardín Botánico de Wuhan de la Academia de Ciencias de China, en colaboración con investigadores de la Universidad Normal de Hunan, completaron por primera vez la secuenciación y el ensamblaje del ADNmt completo de P. hunanensis utilizando la última tecnología de secuenciación con la ventaja de larga duración. -lee (Nanopore).
Los resultados fueron publicados en BMC Genomics. titulado "Ensamblaje y análisis comparativo del genoma mitocondrial completo inicial de Primulina hunanensis (Gesneriaceae):una planta cavernícola en peligro de extinción".
Los investigadores analizaron las características del ADNmt de P. hunanensis, incluido el contenido genético, la preferencia de uso de codones, las secuencias repetitivas, la edición de ARN y las relaciones filogenéticas comparativas y la covarianza del genoma con el ADNmt publicado en Labiatae.
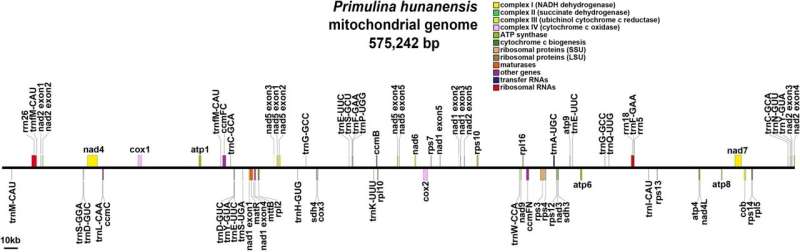
Descubrieron que el ADNmt de P. hunanensis tenía una longitud total de 575.242 pb, con un contenido de GC del 43,54%. Los datos de lectura larga respaldaron el ensamblaje en una estructura lineal que codifica un total de 60 genes, incluidos 37 genes codificadores de proteínas, 20 genes de ARN de transferencia y tres genes de ARN ribosómico.

Además, los genes codificadores de proteínas representaron el 9,32% de la longitud total del ADNmt, y se detectaron un total de 31 codones con valores relativos de uso de codones sinónimos superiores a 1.116 repeticiones simples, siete repeticiones en tándem y 362 secuencias repetidas dispersas, y 455 secuencias potenciales. Se identificaron sitios de edición de ARN.
Los resultados de covarianza indicaron una gran cantidad de reordenamientos genómicos entre P. hunanensis y especies estrechamente relacionadas. La topología de la filogenia basada en el genoma mitocondrial mostró que P. hunanensis estaba estrechamente relacionada con Boea hygrometrica.
Este es el primer ADNmt completo de Primulina y el tercer ADNmt completo de Gesneriaceae que se reporta, lo cual es una adición importante a la limitada base de datos de ADNmt de plantas.
Más información: Lingling Chen et al, Ensamblaje y análisis comparativo del genoma mitocondrial completo inicial de Primulina hunanensis (Gesneriaceae):una planta cavernícola en peligro de extinción, BMC Genomics (2024). DOI:10.1186/s12864-024-10247-9
Información de la revista: BMC Genómica
Proporcionado por la Academia China de Ciencias