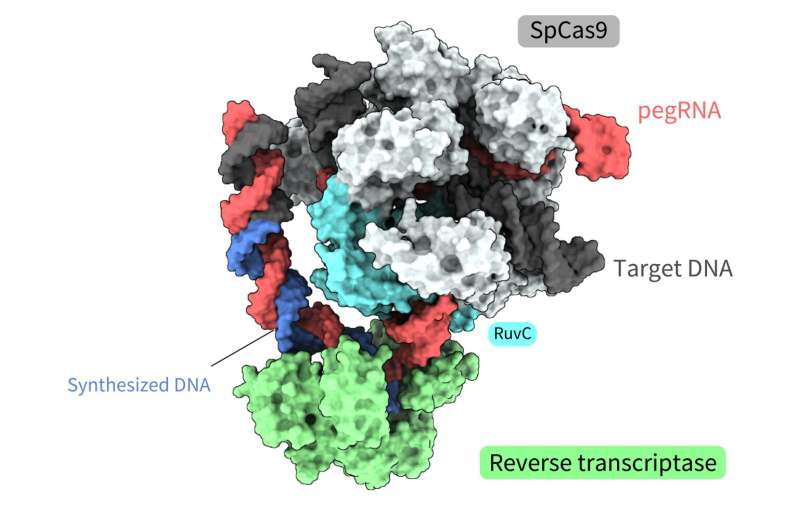
Una investigación conjunta dirigida por Yutaro Shuto, Ryoya Nakagawa y Osamu Nureki de la Universidad de Tokio determinó la estructura espacial de varios procesos de una novedosa herramienta de edición de genes llamada "editor principal". El análisis funcional basado en estas estructuras también reveló cómo un "editor principal" podría lograr la transcripción inversa, sintetizando ADN a partir de ARN, sin "cortar" ambas hebras de la doble hélice.
Aclarar estos mecanismos moleculares contribuye en gran medida al diseño de herramientas de edición de genes lo suficientemente precisas para los tratamientos de terapia génica. Los hallazgos se publican en la revista Nature. .
El Premio Nobel de Química 2020 fue otorgado a Jennifer Doudna y Emmanuelle Charpentier por desarrollar una forma innovadora pero sencilla de editar el ADN, el "modelo" de los organismos vivos. Si bien su descubrimiento abrió nuevas vías para la investigación, la precisión del método y las preocupaciones de seguridad sobre "cortar" ambas hebras de ADN limitaron su uso para tratamientos de terapia génica. Por ello, se han realizado investigaciones para desarrollar herramientas que no tengan estos inconvenientes.
El sistema de edición principal es una de esas herramientas, un complejo molecular que consta de dos componentes. Uno de los componentes es el editor principal, que combina una proteína SpCas9, utilizada en la primera tecnología de edición de genes CRISPR-Cas, y una transcriptasa inversa, una enzima que transcribe el ARN en ADN.
El segundo componente es el ARN guía de edición principal (pegRNA), un ARN guía modificado que identifica la secuencia objetivo dentro del ADN y codifica la edición deseada. En este complejo, el editor principal funciona como un "procesador de textos", reemplazando con precisión la información genómica. La herramienta ya se ha implementado con éxito en células vivas de organismos como plantas, peces cebra y ratones. Sin embargo, no está claro exactamente cómo este complejo molecular ejecuta cada paso del proceso de edición, principalmente debido a la falta de información sobre su estructura espacial.
"Sentimos curiosidad por saber cómo funcionan juntas la combinación antinatural de las proteínas Cas9 y la transcriptasa inversa", dice Shuto, el primer autor del artículo.
El equipo de investigación utilizó microscopía electrónica criogénica, una técnica de obtención de imágenes que permite realizar observaciones a una escala casi atómica. El método requería que las muestras estuvieran en hielo vítreo para protegerlas del daño potencial de los haces de electrones, lo que plantea algunos desafíos adicionales.
"Encontramos que el complejo del editor principal era inestable en condiciones experimentales", explica Shuto. "Por lo tanto, fue muy difícil optimizar las condiciones para que el complejo se mantuviera estable. Durante mucho tiempo, sólo pudimos determinar la estructura de Cas9".
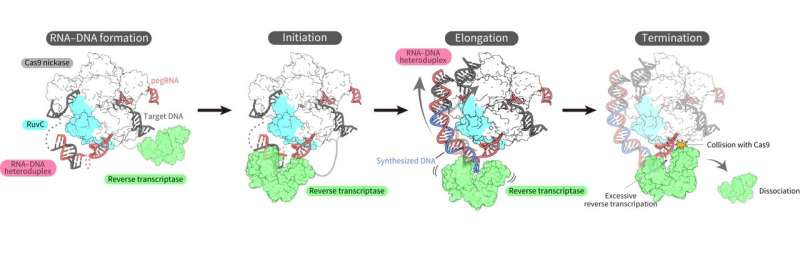
Finalmente, superando los desafíos, los investigadores lograron determinar la estructura tridimensional del complejo editor principal en múltiples estados durante la transcripción inversa en el ADN objetivo.
Las estructuras revelaron que la transcriptasa inversa se unía al complejo ARN-ADN que se formó a lo largo de la "parte" de la proteína Cas9 asociada con la escisión del ADN, la división de una sola hebra de la doble hélice. Mientras realizaba la transcripción inversa, la transcriptasa inversa mantuvo su posición relativa a la proteína Cas9. Los análisis estructurales y bioquímicos también indicaron que la transcriptasa inversa podría provocar inserciones adicionales no deseadas.
Estos hallazgos han abierto nuevas vías para la investigación tanto básica como aplicada. Entonces, Shuto establece los siguientes pasos.
"Nuestra estrategia de determinación de estructura en este estudio también se puede aplicar a editores principales compuestos por una proteína Cas9 diferente y una transcriptasa inversa. Queremos utilizar la información estructural recientemente obtenida para conducir al desarrollo de editores principales mejorados".
Más información: Base estructural para la transcripción inversa guiada por pegRNA por el editor principal, Nature (2024). DOI:10.1038/s41586-024-07497-8, https://www.nature.com/articles/s41586-024-07497-8
Información de la revista: Naturaleza
Proporcionado por la Universidad de Tokio