Cuando se intenta medir estructuras moleculares con precisión nanométrica, cada bit de ruido aparece en los datos:alguien que pasa junto al microscopio, pequeñas vibraciones en el edificio e incluso el tráfico exterior. Una nueva técnica de procesamiento elimina el ruido de los datos del microscopio óptico en tiempo real, lo que permite a los científicos rastrear moléculas individuales con una precisión 10 veces mayor de lo que era posible antes.
Un equipo de investigadores de bioingeniería de la Universidad de Illinois Urbana-Champaign ha introducido un algoritmo llamado maximización de intersección adaptativa, o AIM, que elimina el ruido de alta frecuencia de los datos de microscopio óptico de súper resolución mucho más rápido que los métodos estándar y da como resultado resoluciones de imagen mucho más altas. .
El algoritmo permitirá a los científicos estudiar sistemas químicos y biológicos con mucha más facilidad y precisión que antes. Esta investigación fue publicada en la revista Science Advances.
"Al principio, sólo queríamos desarrollar un algoritmo rápido porque nuestro laboratorio produce demasiados datos para que los manejen los algoritmos tradicionales, pero descubrimos que AIM también puede lograr una precisión subnanométrica, algo inaudito en nuestro campo", dijo Hongqiang Ma. , profesor investigador de bioingeniería y autor principal del estudio. "Además, no requiere una inmensa potencia informática como las herramientas tradicionales. Puede ejecutarse en una computadora portátil. Queremos que sea una herramienta plug-and-play para todos los usuarios de microscopios".
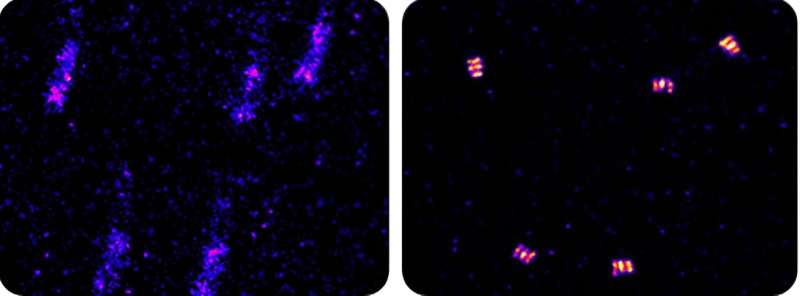
En las últimas décadas, la técnica de microscopía de localización de moléculas individuales ha permitido a los científicos visualizar estructuras a escala molecular, superando lo que se pensaba que era una limitación fundamental de los microscopios ópticos. Sin embargo, en la práctica está limitado por un ruido incontrolable o "deriva", que esencialmente desenfoca las imágenes e impide que la microscopía de súper resolución alcance su resolución más alta.
"La localización de una sola molécula en realidad utiliza un instrumento bastante simple, pero la parte complicada que realmente afecta la resolución de la imagen es la deriva", dijo Yang Liu, profesor de bioingeniería y líder del proyecto. "Muchos investigadores solo eliminan la deriva de baja frecuencia. Eliminar la deriva de alta frecuencia (vibraciones diminutas causadas por el ruido ambiental) requiere un gran esfuerzo computacional y requiere grandes cantidades de tiempo y recursos".
Los métodos estándar para eliminar la deriva se basan en correlaciones matemáticas entre fotogramas de imágenes. Según Liu, los microscopios de su laboratorio generan un volumen tan grande de datos de imágenes que los métodos de correlación de imágenes tardan días incluso con recursos de supercomputación.
AIM también compara cuadros adyacentes, pero procede colocando cada punto de datos en el centro de un círculo (definido por la precisión de localización) y buscando puntos dentro de ese círculo en otros cuadros. Los puntos superpuestos dentro del "radio de intersección" se condensan en una única localización. Luego, se repite el proceso una vez más con los puntos condensados. Estos pasos utilizan recursos computacionales mínimos y son más rápidos que el tiempo de adquisición de una cámara de microscopio. Por lo tanto, las imágenes con corrección de deriva se pueden producir en tiempo real.
Los investigadores probaron AIM utilizando datos simulados y estructuras llamadas origami de ADN que tienen características bien definidas. El algoritmo localizó con éxito las estructuras y el grado de precisión, inferior a 1 nanómetro, resultó ser mucho mayor que el de los métodos estándar de correlación de imágenes, de unos 10 nanómetros.
El laboratorio de Liu incorporará AIM en técnicas de microscopía de alto rendimiento que se están desarrollando para mejorar la detección de enfermedades. Sin embargo, Liu también cree que el algoritmo encontrará usos en toda la biología y la bioingeniería. "Es una herramienta rápida y fácil de usar, y queremos que sea ampliamente accesible para toda la comunidad", afirmó. "Estamos haciendo que nuestro software sea de acceso público. Queremos que las personas obtengan un aumento en la resolución de su imagen solo con este pequeño proceso de posprocesamiento".
Más información: Hongqiang Ma et al, Hacia una nanoscopia de alto rendimiento sin deriva mediante la maximización adaptativa de intersecciones, Avances científicos (2024). DOI:10.1126/sciadv.adm7765
Información de la revista: Avances científicos
Proporcionado por la Facultad de Ingeniería Grainger de la Universidad de Illinois