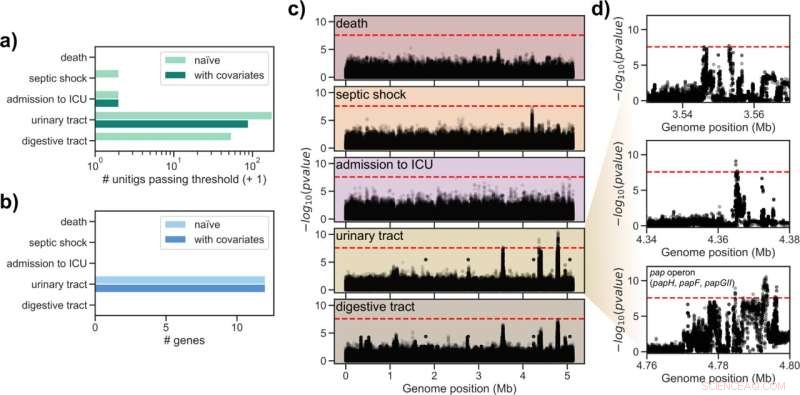
Resultados del análisis de asociación de todo el genoma en el conjunto de datos combinado. a) Número de unitigs que superan el umbral del valor p de corrección de pruebas múltiples para cada fenotipo objetivo. b) Número de genes con unitigs significativamente asociados asignados a ellos para cada fenotipo objetivo. c) Gráficos de Manhattan para mapeo de unitigs probados a E. coli IAI39 para cada fenotipo objetivo; la línea discontinua roja indica el umbral del valor p utilizado para llamar asociaciones significativas. d) Gráficos de Manhattan ampliados para el rasgo del tracto urinario. Los genes anotados con un nombre de gen en E. coli IAI39 y con unitigs asociados se indican en el tercer subpanel. Crédito:PLOS Genetics (2022). DOI:10.1371/journal.pgen.1010112
La bacteria Escherichia coli se encuentra en el intestino humano y en otros lugares. Allí es inofensivo, pero en determinadas condiciones puede convertirse en patógeno. Puede causar infecciones de la vejiga o incluso sepsis. Un equipo de investigadores dirigido por el profesor de RESIST Marco Galardini en TWINCORE, junto con colegas de la facultad de medicina de la Universidad de París, ahora ha investigado si ciertos genes de la bacteria están asociados con la gravedad de las enfermedades causadas. Ahora han publicado sus hallazgos en la revista PLOS Genetics .
Escherichia coli, a menudo abreviada como E. coli, forma parte de la flora intestinal humana. Como un llamado comensal, normalmente no causa ningún daño allí. Sin embargo, puede convertirse en un patógeno en el intestino y en otros órganos. En el tracto urogenital, por ejemplo, E. coli causa infecciones de la vejiga y en el torrente sanguíneo puede causar sepsis. El envenenamiento de la sangre es una consecuencia temida de las infecciones bacterianas e incluso puede ser fatal en 10 a 30 por ciento de los casos. Hasta ahora, no era posible predecir la gravedad de una infección de este tipo en función de la composición genética del patógeno.
Investigadores de TWINCORE en Hannover ahora han analizado si ciertas variantes genéticas de E. coli están asociadas con un curso más severo. "Llevamos a cabo el llamado estudio de asociación de todo el genoma", dice Marco Galardini, jefe del grupo de investigación RESIST Biología de sistemas de comunidades microbianas. "Para esto, secuenciamos muestras bacterianas de dos grandes estudios de pacientes y las correlacionamos con el curso de la infección". También se incluyeron en el análisis características como la edad, el sexo o enfermedades previas conocidas.
El equipo de Galardini no pudo identificar los genes que determinan la gravedad de la enfermedad. Sin embargo, hicieron otro descubrimiento interesante:"Cierto casete genético estaba claramente asociado con infecciones que comenzaron en el tracto urinario", dice Galardini. A partir de esto, se puede derivar una estrategia para evitar enfermedades potencialmente mortales. "En el futuro, se podría secuenciar los patógenos en el caso de una infección de la vejiga y luego decidir si el tratamiento farmacológico debe ajustarse como medida de precaución", dice Galardini.
El hecho de que los investigadores no hayan podido probar una conexión entre el genoma de la bacteria y el curso de la enfermedad no significa necesariamente que no la haya. "Bien podría ser que el número de muestras que estudiamos fuera demasiado pequeño", dice Galardini. "Una simulación mostró que se necesitarían diez veces más muestras para detectar o excluir el enlace con más confianza".
Por eso, Galardini y sus socios de la cooperación francesa están preparando un estudio de seguimiento más amplio. Ya han presentado una solicitud para la financiación necesaria.