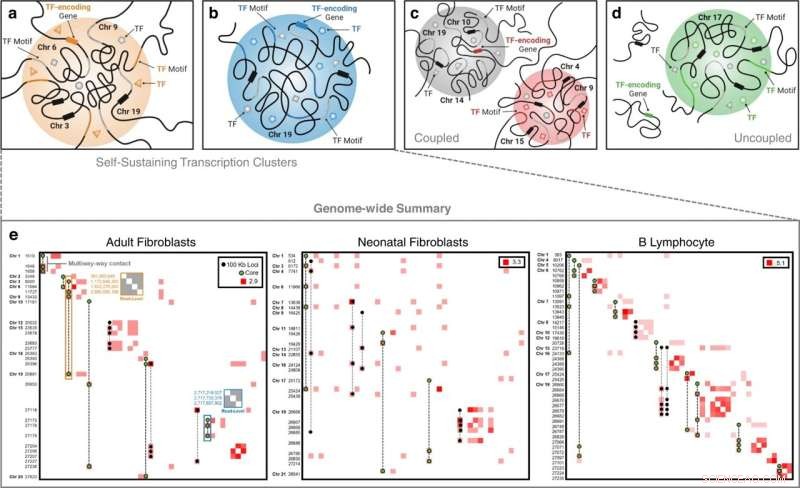
Clases de grupos de transcripción. En un grupo de transcripción autosostenible, están presentes un TF y el gen que codifica ese TF. Los ejemplos intercromosómicos e intracromosómicos en (a) y (b), respectivamente, ilustran este fenómeno donde en a vemos el TF de interés (triángulo naranja) circulando en el grupo, su motivo de unión presente en la cromatina (porción naranja ), y su gen correspondiente expresado (rectángulo naranja en el cromosoma 6). Las formas grises representan TF adicionales con motivos de unión (porción gris de cromatina) en el grupo. Los rectángulos negros en los cromosomas 3, 9 y 19 representan genes adicionales presentes en el grupo. c Una clase de grupos de transcripción independientes de los análogos donde observamos un enlace TF (cuadrado rojo) en un grupo de transcripción (grupo rojo) y su gen correspondiente expresado en un grupo de transcripción separado (grupo gris), pero no en el mismo grupo. d Una clase independiente de análogos de grupos de transcripción donde observamos un TF (círculo verde) que se une a un grupo de transcripción (grupo verde) y su gen correspondiente expresado pero no dentro de un grupo de transcripción. e Grupos de transcripción autosuficientes específicos del tipo de célula de todo el genoma extraídos de datos de contacto multidireccional y descompuestos en matrices de contacto Hi-C a una resolución de 100 kb. Las frecuencias de contacto se transforman logarítmicamente para una mejor visualización. Las frecuencias a lo largo de la diagonal indican la interacción entre dos o más loci multidireccionales únicos que se encuentran dentro del mismo contenedor de 100 kb. Las etiquetas de eje son coordenadas bin de 100 kb no contiguas en orden cromosómico. Se superponen los contactos multidireccionales que componen los grupos de transcripción autosuficientes. Los contactos multidireccionales con loci de color verde representan grupos de transcripción "centrales", grupos de transcripción que contienen un regulador maestro y su gen análogo. Un ejemplo de mapa de contacto de nivel de lectura para el grupo de transcripción autosostenible intercromosómico FOXO3 se indica mediante el cuadro resaltado en naranja en la matriz de contacto de fibroblastos adultos y un mapa de contacto de nivel de lectura para el grupo de transcripción autosostenible intracromosómico ZNF320 es indicado por el cuadro resaltado en azul. Los valores a lo largo del eje izquierdo de estas matrices de contacto de nivel de lectura son posiciones de pares de bases de los loci de contacto en el genoma. Crédito:Comunicaciones de la naturaleza (2022). DOI:10.1038/s41467-022-32980-z
La tercera ley del escritor de ciencia ficción Arthur C. Clarke dice que "cualquier tecnología suficientemente avanzada es indistinguible de la magia".
Indika Rajapakse, Ph.D., es creyente. El ingeniero y matemático ahora se encuentra a sí mismo como biólogo. Y él cree que la belleza de combinar estas tres disciplinas es crucial para desentrañar cómo funcionan las células.
Su último desarrollo es una nueva técnica matemática para empezar a entender cómo se organiza el núcleo de una célula. La técnica, que Rajapakse y sus colaboradores probaron en varios tipos de células, reveló lo que los investigadores denominaron grupos de transcripción autosostenidos, un subconjunto de proteínas que desempeñan un papel clave en el mantenimiento de la identidad celular.
Esperan que esta comprensión exponga las vulnerabilidades que pueden orientarse para reprogramar una célula para detener el cáncer u otras enfermedades.
"Cada vez más biólogos del cáncer piensan que la organización del genoma juega un papel muy importante en la comprensión de la división celular incontrolable y si podemos reprogramar una célula cancerosa. Eso significa que debemos comprender más detalles sobre lo que sucede en el núcleo", dijo Rajapakse, profesor asociado de computación. medicina y bioinformática, matemáticas e ingeniería biomédica en la Universidad de Michigan. También es miembro del U-M Rogel Cancer Center.
Rajapakse es el autor principal del artículo, publicado en Nature Communications . El proyecto fue dirigido por un trío de estudiantes graduados con un equipo interdisciplinario de investigadores.
El equipo mejoró una tecnología más antigua para examinar la cromatina, llamada Hi-C, que mapea qué partes del genoma están juntas. Puede identificar translocaciones cromosómicas, como las que ocurren en algunos tipos de cáncer. Sin embargo, su limitación es que solo ve estas regiones genómicas adyacentes.
La nueva tecnología, llamada Pore-C, utiliza muchos más datos para visualizar cómo interactúan todas las piezas dentro del núcleo de una célula. Los investigadores utilizaron una técnica matemática llamada hipergrafías. Piensa:diagrama de Venn tridimensional. Permite a los investigadores ver no solo pares de regiones genómicas que interactúan, sino la totalidad de las relaciones complejas y superpuestas de todo el genoma dentro de las células.
"Esta relación multidimensional que podemos entender sin ambigüedades. Nos da una forma más detallada de entender los principios organizacionales dentro del núcleo. Si entiendes eso, también puedes entender dónde se desvían estos principios organizacionales, como en el cáncer", dijo Rajapakse. "Esto es como juntar tres mundos (tecnología, matemáticas y biología) para estudiar con más detalle el interior del núcleo".
Los investigadores probaron su enfoque en fibroblastos neonatales, fibroblastos adultos biopsiados y linfocitos B. Identificaron organizaciones de grupos de transcripción específicos para cada tipo de célula. También encontraron lo que llamaron grupos de transcripción autosuficientes, que sirven como firmas transcripcionales clave para un tipo de célula.
Rajapakse describe esto como el primer paso de un panorama más amplio.
"Mi objetivo es construir este tipo de imagen sobre el ciclo celular para comprender cómo una célula pasa por diferentes etapas. El cáncer es una división celular incontrolable", dijo Rajapakse. "Si entendemos cómo cambia una célula normal con el tiempo, podemos comenzar a examinar los sistemas controlados y no controlados y encontrar formas de reprogramar ese sistema". Revelando los misterios de la estructura del genoma en el núcleo de la célula humana usando una simulación computacional 3D