Crédito:Universidad de Rice
Los científicos de la Universidad de Rice que dicen que los sensores biológicos no son lo suficientemente sensibles están haciendo algo al respecto.
El laboratorio del biólogo sintético Jeffrey Tabor ha introducido una nueva técnica para aumentar o disminuir la sensibilidad de los sistemas de dos componentes:una clase de proteínas que utilizan las bacterias para detectar una amplia variedad de estímulos.
La técnica podría permitir la ingeniería de biosensores hechos a medida para el diagnóstico de bacterias intestinales, detección de contaminantes ambientales o control automatizado de los niveles de nutrientes en el suelo.
Sensores de dos componentes, el enfoque de un nuevo artículo que describe el trabajo en Nature Communications, son una gran familia de sensores codificados genéticamente que las bacterias utilizan para detectar una entrada específica y activar un gen específico en respuesta a cambios en su entorno.
Si bien estos sensores se conocen desde hace tres décadas, no eran lo suficientemente sensibles para Tabor. Él y el ex alumno de Rice y autor principal, Brian Landry, se propusieron disminuir la cantidad de información necesaria para activarlos.
Combinando modelos y experimentos por computadora, lograron precisamente eso. En el papel, describen su descubrimiento de que la actividad de la fosfatasa, que es esencial para la regulación y la señalización en las células, puede tener un efecto dramático en los umbrales de detección de los sistemas de dos componentes.
Investigaciones anteriores habían demostrado que las mutaciones en el primer componente, una proteína sensora de señalización conocida como histidina quinasa, se puede utilizar para controlar el grado de fosforilación del segundo componente, una proteína reguladora de la respuesta. Pero nadie había sugerido que las mutaciones de fosfatasa podrían usarse para alterar la sensibilidad de estas vías para sus entradas, Dijo Tabor.
Un día, frente a un sensor de nitrato que no se enciende como se esperaba en el intestino de un ratón enfermo, Landry planteó la hipótesis de que las mutaciones de la fosfatasa podrían aumentar drásticamente la sensibilidad de la vía.
Primero validó la hipótesis usando un modelo matemático y luego realizó experimentos que mostraron que las mutaciones aumentaron la eficiencia de un sensor de nitrato en un factor de 100. El laboratorio de Tabor pasó a validar el enfoque en sistemas de dos componentes que detectan una amplia variedad de entradas , incluso en especies de bacterias muy diferentes.
Landry trabajó con otro estudiante de bioingeniería de Rice, el coautor Lucas Hartsough, para identificar un aminoácido de "punto caliente" presente en el 64 por ciento de todas las histidina quinasas sensoras que se pueden mutar para ajustar la sensibilidad. Ellos validaron el enfoque en dos vías mínimamente caracterizadas, que sugirió que se puede aplicar ampliamente, Dijo Tabor.
Landry también utilizó la tecnología para diseñar una bacteria del suelo, Bacillus subtilis, para detectar y responder a una amplia gama de concentraciones de nitratos (fertilizantes) en el suelo. Tabor y Landry sugieren que este sistema podría acoplarse a las vías que está desarrollando un colega del MIT, biólogo sintético Chris Voigt, y su laboratorio para diseñar bacterias del suelo que mantengan niveles óptimos de nitrógeno en el suelo sin necesidad de fertilizantes.
Los sistemas de dos componentes pueden activarse mediante muchos tipos de entradas, incluidos los gases, moléculas de hemo en sangre, azúcares polisacáridos intestinales, hormonas humanas o vegetales o incluso luz.
"Por eso estamos tan emocionados con esto, "Dijo Tabor." Todos estos sensores funcionan de la misma manera. Todos tienen la fosfatasa y la quinasa. Hemos identificado más de 25, 000 de ellos en genomas bacterianos y creemos que nuestra estrategia funcionará en la mayoría de ellos ".
Dijo que la técnica de su laboratorio se basa en la vía de señalización por la cual las moléculas sensoras naturales le dicen a las células que se detengan. iniciar o modificar la producción de proteínas. Ajustar los sensores naturales para aplicaciones sintéticas ha sido difícil hasta ahora, él dijo.
"Una de las grandes limitaciones es que cuando tomas un biosensor de la naturaleza, puede detectar la sustancia química que le interesa, pero no con la concentración adecuada. Esto se debe a que ha evolucionado para permitir que una bacteria sobreviva mejor en un entorno natural en lugar de satisfacer las necesidades de ingeniería de un biólogo sintético. " él dijo.
"Llamamos a este nivel el umbral de detección del sensor, y no ha habido muchas buenas tecnologías para ajustar ese umbral para satisfacer nuestras necesidades de ingeniería, "Dijo Tabor." Esa ha sido una limitación importante en el diseño de biosensores. Pero ahora, Hemos encontrado un truco general que nos permite ajustar los umbrales de detección de esta familia de sensores bacterianos de una manera muy racional. y funciona bastante bien ".
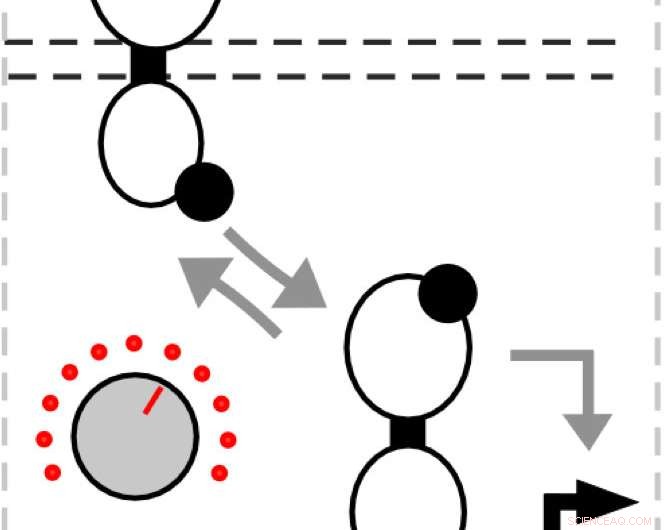
En sus simulaciones y experimentos, el laboratorio mutó la proteína histidina quinasa, que detecta una entrada y libera un grupo fosfato que se une al regulador de respuesta. Ese, Sucesivamente, se une al ADN para activar la expresión génica.
"Cuando activas la expresión genética, esa es tu señal, "Tabor dijo." Lo bueno de estas vías es que cuando la entrada está ausente, el sensor de histidina quinasa actúa como una fosfatasa y elimina el grupo fosfato. Cuando eso pasa, el regulador de respuesta se desprende del ADN y ya no activa la expresión génica ".
Dijo que la mutación de la histidina quinasa para favorecer el encendido o apagado de la señal puede usarse para controlar las acciones de los reguladores de respuesta, y por tanto la cantidad de proteínas deseadas producidas.
"Pensamos que es como un tira y afloja entre la actividad de la quinasa y la actividad de la fosfatasa, "Dijo Tabor." Ese equilibrio determina cuánta entrada se necesita para encender el sensor. Así que esto es como una perilla donde podemos hacer que la actividad de la fosfatasa sea más fuerte o más débil para ajustar la entrada ".
Dijo que la generalidad del proceso debería impulsar la biología sintética en general. "Hay métodos alternativos para hacer lo que hicimos aquí, pero son mucho más intensivos en mano de obra, "Dijo Tabor." Es más probable que fallen, y tomarían un doctorado completo. para hacerlos trabajar, mientras que podemos hacer esto en una semana y hacerlo funcionar ".