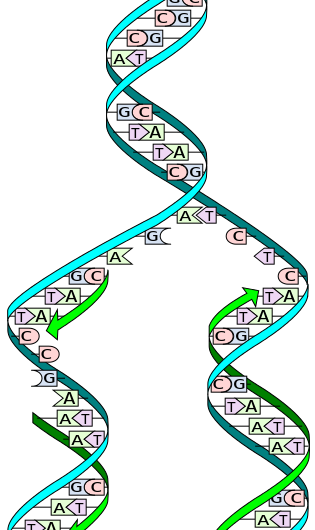
Durante la replicación, las dos hebras están separadas. Cada hebra de la molécula de ADN original sirve entonces como plantilla para la producción de su contraparte complementaria. Crédito:Wikimedia Commons
El análisis de ADN es útil para varias aplicaciones vitales. Esto incluye el diagnóstico y seguimiento de enfermedades, identificación de criminales, y estudiar la función de un segmento específico de ADN. Sin embargo, Los métodos utilizados para los análisis a menudo requieren más ADN del que puede estar disponible en una muestra típica. 'Por lo tanto, la amplificación es necesaria, pero no siempre sencillo. El método de fotocopia o amplificación más utilizado es la reacción en cadena de la polimerasa (PCR). Un nuevo método de PCR podría ayudar al proceso de amplificación, y así desarrollar ensayos robustos que antes no hubieran sido posibles.
El ácido desoxirribonucleico (ADN) es una molécula que se encuentra en el núcleo de las células y lleva las "instrucciones" para el desarrollo y funcionamiento de los organismos vivos. A menudo se compara con un conjunto de planos, ya que contiene las instrucciones necesarias para construir celdas. Estas instrucciones se dividen en segmentos a lo largo de una hebra de ADN.
Replicación
El ADN está formado por una doble hélice de dos hebras complementarias formadas por nucleótidos, una estructura a menudo comparada con una escalera. Cuando llega el momento de replicar, las dos hebras de ADN, o "lados" de la escalera, se desenrollan y se separan. Una enzima llamada ADN polimerasa lee las hebras individuales y hace coincidir las bases complementarias, los 'peldaños' de la escalera, con la hebra original. Se producen nuevas hebras en ambos lados del ADN original, haciendo dos hélices dobles de ADN idénticas compuestas por una hebra original y una nueva. Este proceso ocurre en todos los organismos vivos y es la base de la herencia biológica.
PCR
La replicación también puede procesarse artificialmente. A veces llamado "fotocopiado molecular, "la reacción en cadena de la polimerasa (PCR) es una técnica rápida y relativamente económica que se utiliza para amplificar, o hacer muchas copias de, pequeños segmentos de ADN. Esto es necesario porque los métodos utilizados para analizar el ADN o determinar la secuencia del par de bases del ADN requieren más ADN del que puede haber en una muestra típica. Por eso, el objetivo de la PCR es amplificar una región específica de una cadena de ADN.
Para realizar PCR, las dos hebras de la doble hélice del ADN se separan físicamente mediante la aplicación de alta temperatura (93-98 ° C) en un proceso llamado fusión del ADN. En el segundo paso, se baja la temperatura y las dos hebras de ADN, ahora separados, se convierten en plantillas para los denominados cebadores. Los cebadores son moléculas de ADN monocatenarias cortas, complementaria a la región de ADN destinada a la amplificación. Después de bajar la temperatura, los cebadores añadidos al proceso de PCR encuentran y se unen a su objetivo específico. Una enzima llamada ADN polimerasa alargará, o construirá, una nueva hebra de ADN a partir del cebador utilizando la molécula de ADN monocatenaria subyacente como plantilla.
Construyendo una escalera
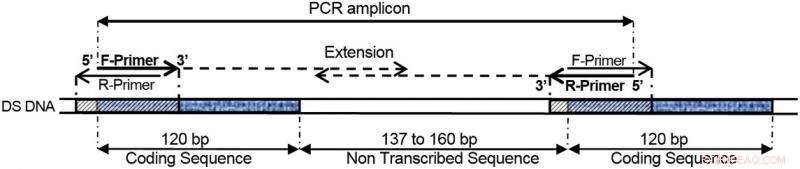
COMPAS-PCR utilizando cebadores altamente complementarios aplicados a genes de repetición directa de ADN ribosómico 5S, como se muestra en este ejemplo. Crédito:Instituto Noruego de Investigación del Agua (NIVA)
Como se ha mencionado más arriba, los cebadores son moléculas cortas de ADN monocatenarias. Suelen tener alrededor de 20 nucleótidos de longitud. Esta cadena de nucleótidos, se adhiere específicamente al comienzo de la hebra de la plantilla mediante el emparejamiento de bases, encontrando las bases complementarias. Entonces, la ADN polimerasa puede agregar el siguiente nucleótido complementario. La polimerasa continúa agregando más nucleótidos complementarios al ADN molde hasta que se completa una nueva doble hebra de ADN. o para usar la metáfora anterior; se construye una nueva escalera, con una cara original y una cara nueva.
Los cebadores duales permiten definir con precisión el área de una molécula de ADN que se amplifica mediante la PCR. Estos dos cebadores flanqueantes especifican dónde debe comenzar y terminar la nueva cadena.
Un pequeño cambio de paradigma
Una regla central fuertemente anclada en la mente del científico molecular es que los cebadores deben ser complementarios solo a la secuencia objetivo, y no el uno con el otro. Esto para evitar que los cebadores se utilicen entre sí como plantillas y, por lo tanto, deshabiliten una posible participación adicional en la amplificación tradicional de la diana.
En un estudio reciente, publicado en la revista científica Más uno , El científico investigador principal Marc Anglès d´Auriac del Instituto Noruego de Investigación del Agua (NIVA) muestra que es posible utilizar cebadores altamente complementarios y, sin embargo, evitar las desafortunadas consecuencias mencionadas anteriormente. El nuevo método ha sido denominado COMPAS-PCR, abreviatura de COMplementary Primer Asymmetric PCR.
En breve, Anglès d'Auriac observó que los cebadores complementarios entre ellos y un objetivo (superposición triple) aún podrían definir un producto de amplificación cuando forma parte de un motivo o estructura de ADN repetido. Esto se muestra en la siguiente figura. Más lejos, el impedimento físico de la amplificación de la diana debido a la complementariedad del cebador se alivió mediante la introducción de concentraciones de cebador asimétricas. Asimétrico a este respecto significa un número impar de moléculas entre los dos cebadores. Un cebador consta de un número reducido de moléculas, el otro con un número alto.
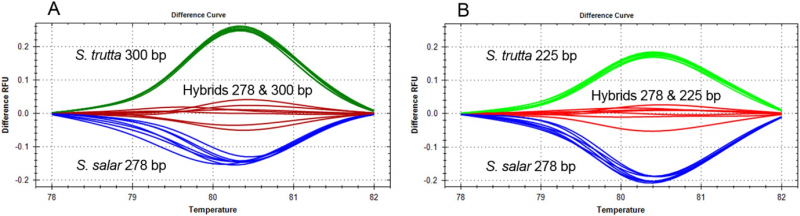
Análisis de fusión de alta resolución de dos COMPAS-PCR dúplex de tres cebadores para la diferenciación de S. salar, S. trutta e híbridos. Crédito:Instituto Noruego de Investigación del Agua (NIVA)
"Si usa concentraciones iguales, como suele hacer para la PCR, las cartillas, presente en cantidades iguales, se pegarán el uno al otro, "Dice Anglès d'Auriac.
"Con concentraciones asimétricas, el cebador en exceso o muy concentrado tiene un número de moléculas que no se pegan al cebador limitante, y por lo tanto está disponible para la amplificación de la diana ".
Trabajando solo
"Este enfoque contrario a la intuición aislará, pero también proteger, la imprimación de baja concentración, "Dice Anglès d'Auriac.
Con el cebador limitador pegado, y así protegido, es posible que la imprimación de alta concentración funcione sola, es decir, hacer una copia de una sola hebra del ADN objetivo. Cuando se producen suficientes amplicones monocatenarios, pueden servir como material de plantilla para la imprimación de baja concentración. Se libera el cebador limitante, y la reacción cambia a la amplificación por PCR exponencial clásica.
"Esto amplía las posibilidades de aplicación de la PCR para el científico, Anglès d'Auriac aclara ".
Estructuras repetitivas
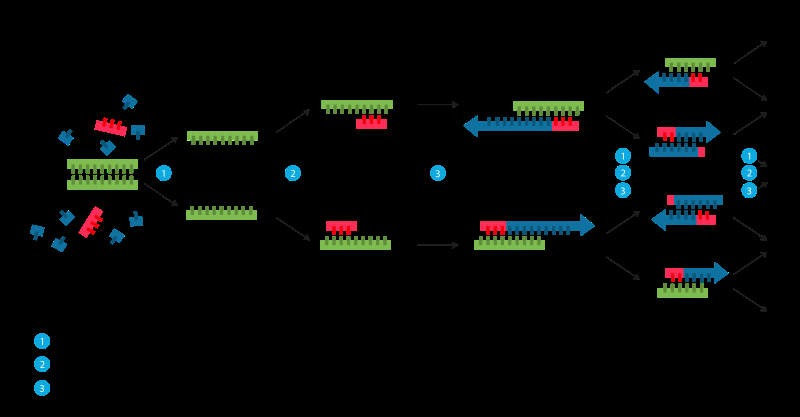
Reacción en cadena de la polimerasa (PCR). Crédito:Wikimedia
En muchos organismos, una fracción significativa del ADN genómico es altamente repetitiva, con más de la mitad de la secuencia que consta de elementos repetitivos en humanos.
De hecho, fue una estructura de ADN repetitiva lo que hizo que Anglès d'Auriac pensara fuera de la caja y propusiera el principio COMPAS-PCR, un pequeño cambio de paradigma en el campo de la fotocopia molecular.
En el proceso de realizar un diagnóstico basado en ADN para salmónidos, en particular la identificación de la trucha marrón (Salmo trutta) y el salmón del Atlántico (Salmo salar) estrechamente relacionados, Anglès d'Auriac luchó por separar estas especies, incluidos los híbridos entre ellas. La identificación rápida y precisa ayudaría a mejorar el seguimiento y los estudios de los ecosistemas fluviales. ya que la identificación de híbridos es importante para estimar la salud ecológica de las cuencas hidrográficas. Después de usar COMPAS-PCR, Anglès d'Auriac pudo aplicar un método de análisis de producto de PCR llamado análisis de fusión de alta resolución para identificar la trucha marrón, Salmón del Atlántico y sus híbridos en una prueba.
Sin embargo, Anglès d'Auriac hace hincapié en que el principio COMPAS-PCR no se limita a la identificación de especies de peces. El uso de cebadores casi completamente complementarios dirigidos a la misma secuencia puede aplicarse a cualquier copia que se encuentre adyacente entre sí, en motivos de ADN de interés como secuencias diana.
"Se informa que las secuencias de repetición de ADN comprenden más del 50% del genoma humano y están presentes en muchas familias de genes" domésticos ", como el gen ribosómico 5S utilizado en este estudio. Por lo tanto, Los principios generales de COMPAS-PCR ayudarán a desarrollar nuevas estrategias de amplificación de ADN aprovechando estas estructuras de ADN repetidas. "Concluye Marc Anglès d'Auriac.