Debido a la alta transparencia de las células, es muy difícil observar los orgánulos que contienen. Los biólogos pueden etiquetar orgánulos específicos para su observación mediante tinción fluorescente. Esto es algo análogo a estar en un ambiente sin luz donde todos están vestidos completamente de negro, lo que dificulta encontrar a tus amigos. Si hacemos que nuestros amigos sostengan un palo fluorescente, podremos localizarlos fácilmente.
Una pregunta interesante es:si el ángulo de la barra fluorescente que sostiene mi amigo representa un tipo de señal, ¿cómo podemos detectar esa información angular?
Al igual que en este rompecabezas, debido a la naturaleza altamente transparente de las células, es muy difícil observar los orgánulos que contienen. Con la tinción fluorescente, los biólogos pueden etiquetar orgánulos específicos para su observación. La mayoría de las moléculas fluorescentes aparecen como dipolos direccionales durante la absorción o la emisión.
La orientación de los fluoróforos puede revelar información importante sobre la estructura y dinámica de sus orgánulos asociados. La microscopía de polarización de fluorescencia también se ha desarrollado como una herramienta indispensable para estudiar las características de orientación de las biomoléculas.
Para superar el desafío de la microscopía de polarización de fluorescencia convencional limitada por la difracción óptica, se han propuesto técnicas mejoradas de microscopía de polarización de fluorescencia de superresolución, como la microscopía de orientación-localización de una sola molécula (SMOLM) y la modulación de polarización (por ejemplo, SDOM, SPoD, etc. ).
Sin embargo, desde el punto de vista biotecnológico, a pesar del importante papel de los filamentos biológicos (p. ej., filamentos de actina y microtúbulos) en las funciones celulares, faltan enfoques con resolución de orientación 3D y alta resolución temporal-espacial para estudiarlos in vivo.
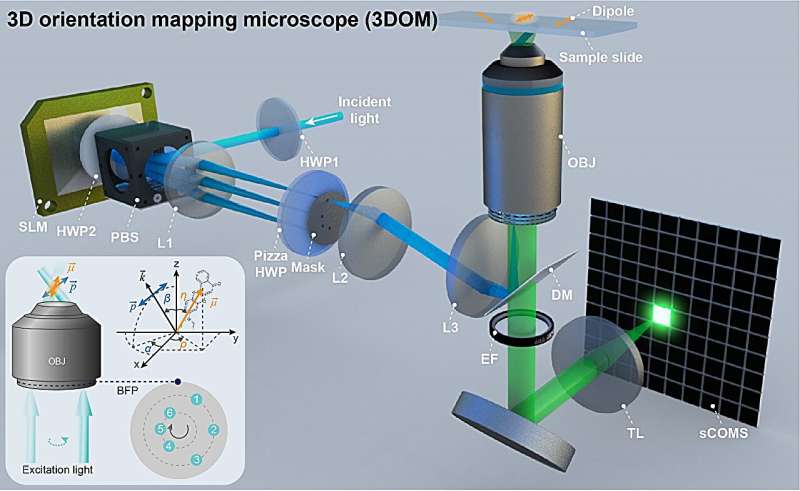
Para abordar el problema de la resolución de la orientación de los dipolos, el grupo de investigación del profesor Xi Peng de la Universidad de Pekín ha desarrollado un método de mapeo de orientación de dipolos 2D, SDOM, y un mapeo de orientación de dipolos de superresolución con detección óptica de bloqueo, OLID-SDOM. En PhotoniX , el grupo de investigación presenta un microscopio de mapeo de orientación 3D de súper resolución denominado 3DOM.
El método 3DOM se basa en la microscopía de iluminación estructurada polarizada desarrollada por el grupo de investigación. Invirtiendo el principio de interferencia de doble rendija de Young y combinándolo con el principio de trayectorias de luz reversibles, se utilizan diferentes ángulos de las franjas para producir haces de primer orden positivos y negativos en diferentes direcciones.
Además, se puede producir una única dirección de iluminación inclinada simplemente bloqueando la correspondiente luz negativa de primer orden. Al proyectar esta inclinación a diferentes ángulos del eje z y reconstruir la imagen usando el algoritmo FISTA, se puede lograr una resolución de alta precisión de la orientación del dipolo combinando los coeficientes de modulación de polarización y los resultados de la reconstrucción en el espacio recíproco.
En general, el método 3DOM propuesto supera eficazmente las limitaciones de la microscopía de polarización de fluorescencia en la resolución espacial y el mapeo de orientación 3D mediante imágenes de campo amplio.
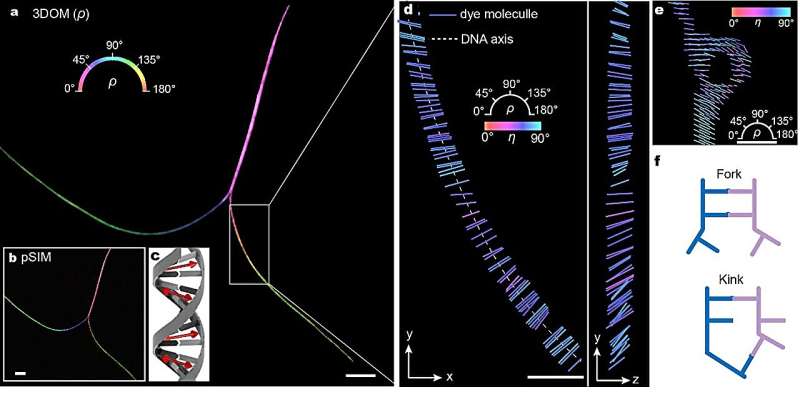
3DOM proporciona una comprensión más completa de la estructura espacial 3D de las moléculas de fluoróforo. Esto nos permite no sólo distinguir varias organizaciones citoesqueléticas (filamentos de actina y microtúbulos), sino también obtener información valiosa sobre la compacidad de unión de los filamentos y el orden de las estructuras subcelulares.
Además, 3DOM tiene un potencial significativo en la flexión del ADN y la orientación de orgánulos membranosos. Una de las ventajas clave de 3DOM es su facilidad de actualización a sistemas de campo amplio existentes. La implementación simple, la información precisa de orientación dipolo 3D y la resolución espaciotemporal superior de 3DOM lo hacen adecuado para una amplia gama de aplicaciones, mejorando su accesibilidad y usabilidad en diferentes entornos de investigación.
Esta poderosa herramienta permite a los investigadores desentrañar las intrincadas complejidades de la estructura subcelular, la biomecánica y la biodinámica, revolucionando nuestra comprensión de los procesos celulares. Los investigadores prevén que 3DOM avanzará en la comprensión de una multitud de estructuras biológicas e interacciones operativas a nanoescala.
Más información: Suyi Zhong et al, Mapeo de orientación dipolo tridimensional con alta resolución temporal-espacial utilizando modulación de polarización, PhotoniX (2024). DOI:10.1186/s43074-024-00127-6
Proporcionado por la Universidad de Pekín