Aunque es posible que no los aprecies, o que ni siquiera hayas oído hablar de ellos, en todo tu cuerpo, innumerables máquinas microscópicas llamadas espliceosomas están trabajando arduamente. Mientras usted se sienta y lee, ellos vuelven a unir fiel y rápidamente la información rota en sus genes mediante la eliminación de secuencias llamadas "intrones" para que sus ARN mensajeros puedan producir las proteínas correctas que necesitan sus células.
Los intrones son quizás uno de los mayores misterios de nuestro genoma. Son secuencias de ADN que interrumpen la información sensible de codificación de proteínas en los genes y necesitan ser "cortadas". El genoma humano tiene cientos de miles de intrones, alrededor de 7 u 8 por gen, y cada uno es eliminado por un complejo de proteínas de ARN especializado llamado "spliceosoma" que corta todos los intrones y une las secuencias codificantes restantes, llamadas exones. Se desconoce cómo evolucionó este sistema de genes rotos y el espliceosoma en nuestros genomas.
A lo largo de su larga carrera, Manny Ares, distinguido profesor de biología molecular, celular y del desarrollo de UC Santa Cruz, se ha propuesto aprender todo lo que pueda sobre el empalme de ARN.
"Me refiero al empalme", dijo Ares. "Sólo quiero saber todo lo que hace el espliceosoma, incluso si no sé por qué lo hace".
En un nuevo artículo publicado en la revista Genes and Development Ares informa sobre un descubrimiento sorprendente sobre el espliceosoma que podría decirnos más sobre la evolución de diferentes especies y la forma en que las células se han adaptado al extraño problema de los intrones. Los autores muestran que una vez que el espliceosoma termina de empalmar el ARNm, permanece activo y puede participar en más reacciones con los intrones eliminados.
Este descubrimiento proporciona la indicación más sólida que tenemos hasta ahora de que los espliceosomas podrían reinsertar un intrón en el genoma en otra ubicación. Esta es una capacidad que antes no se creía que poseyeran los espliceosomas, pero que es una característica común de los "intrones del Grupo II", primos lejanos del espliceosoma que existen principalmente en bacterias.
Se cree que el espliceosoma y los intrones del Grupo II comparten un ancestro común que fue responsable de la propagación de los intrones por todo el genoma, pero mientras que los intrones del Grupo II pueden separarse del ARN y luego regresar directamente al ADN, los "intrones espliceosómicos" que se encuentran en la mayoría de los organismos de nivel superior requieren el espliceosoma para el empalme y no se cree que se reinserten en el ADN. Sin embargo, el hallazgo del laboratorio de Ares indica que el espliceosoma aún podría estar reinsertando intrones en el genoma en la actualidad. Esta es una posibilidad intrigante a considerar porque los intrones que se reintroducen en el ADN agregan complejidad al genoma, y comprender mejor de dónde provienen estos intrones podría ayudarnos a comprender mejor cómo continúan evolucionando los organismos.
Los genes de un organismo están hechos de ADN, en el que cuatro bases, adenina (A), citosina (C), guanina (G) y timina (T), están ordenadas en secuencias que codifican instrucciones biológicas, como cómo producir proteínas específicas en el cuerpo. necesidades. Antes de que se puedan leer estas instrucciones, el ADN se copia en ARN mediante un proceso conocido como transcripción, y luego los intrones de ese ARN deben eliminarse antes de que un ribosoma pueda traducirlo en proteínas reales.
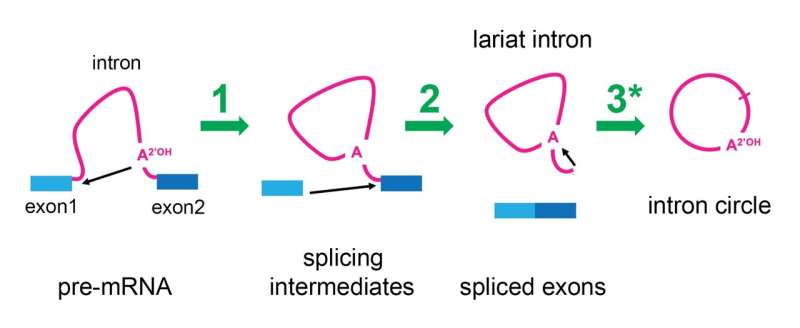
El espliceosoma elimina los intrones mediante un proceso de dos pasos que da como resultado que el ARN del intrón tenga uno de sus extremos unido a su centro, formando un círculo con una cola que parece un "lazo" o lazo de vaquero. Esta apariencia ha llevado a que se les denomine "intrones de lazo". Recientemente, investigadores de la Universidad de Brown que estaban estudiando las ubicaciones de los sitios de unión en estos lazos hicieron una observación extraña:algunos intrones eran en realidad circulares en lugar de tener forma de lazo.
Esta observación llamó inmediatamente la atención de Ares. Algo parecía estar interactuando con los intrones del lazo después de que fueron eliminados de la secuencia de ARN para cambiar su forma, y el espliceosoma era su principal sospechoso.
"Pensé que era interesante debido a esta muy, muy antigua idea sobre el origen de los intrones", dijo Ares. "Existe mucha evidencia de que las partes de ARN del espliceosoma, los snRNA, están estrechamente relacionados con los intrones del Grupo II".
Debido a que el mecanismo químico para el empalme es muy similar entre los espliceosomas y sus primos lejanos, los intrones del Grupo II, muchos investigadores han teorizado que cuando el proceso de auto-empalme se volvió demasiado ineficiente para que los intrones del Grupo II lo completaran de manera confiable por sí solos, partes de Estos intrones evolucionaron hasta convertirse en el espliceosoma. Si bien los intrones del Grupo II pudieron insertarse directamente nuevamente en el ADN, no se pensaba que los intrones esplicosomales que requerían la ayuda de los espliceosomas se insertaran nuevamente en el ADN.
"Una de las preguntas que en cierto modo faltaba en esta historia en mi mente era:¿es posible que el esplicosoma moderno todavía sea capaz de tomar un intrón de lazo e insertarlo en algún lugar del genoma?" Dijo Ares. "¿Es todavía capaz de hacer lo que hizo el complejo de los ancestros?"
Para comenzar a responder a esta pregunta, Ares decidió investigar si efectivamente era el espliceosoma el que estaba realizando cambios en los intrones del lazo para eliminar sus colas. Su laboratorio ralentizó el proceso de empalme en células de levadura y descubrió que después de que el espliceosoma liberara el ARNm del que había terminado de empalmar los intrones, se aferraba a los lazos de intrones y los transformaba en verdaderos círculos. El laboratorio de Ares pudo volver a analizar los datos publicados de secuenciación de ARN de células humanas y descubrió que los espliceosomas humanos también tenían esta capacidad.
"Estamos entusiasmados con esto porque, si bien no sabemos qué podría hacer este ARN circular, el hecho de que el espliceosoma todavía esté activo sugiere que puede catalizar la inserción del intrón Lariat nuevamente en el genoma", dijo Ares.
Si el espliceosoma es capaz de reinsertar el intrón en el ADN, esto también agregaría peso significativo a la teoría de que los espliceosomas y los intrones del Grupo II compartieron un ancestro común hace mucho tiempo.
Ahora que Ares y su laboratorio han demostrado que el espliceosoma tiene la capacidad catalítica de colocar hipotéticamente intrones nuevamente en el ADN como lo hicieron sus antepasados, el siguiente paso para los investigadores es crear una situación artificial en la que "alimenten" una cadena de ADN a un empalme que todavía está unido a un intrón de lazo y ver si realmente pueden lograr que inserte el intrón en algún lugar, lo que presentaría una "prueba de concepto" para esta teoría.
Si el espliceosoma es capaz de reinsertar intrones en el genoma, es probable que sea un evento muy poco frecuente en humanos, porque los espliceosomas humanos tienen una demanda increíblemente alta y, por lo tanto, no tienen mucho tiempo para dedicar a los intrones eliminados. Sin embargo, en otros organismos donde el espliceosoma no está tan ocupado, la reinserción de intrones puede ser más frecuente. Ares está trabajando estrechamente con el profesor de ingeniería biomolecular de la UCSC, Russ Corbett-Detig, quien recientemente dirigió una búsqueda sistemática y exhaustiva de nuevos intrones en los genomas disponibles de todas las especies que contienen intrones que se publicó en la revista Proceedings of the National Academy. de Ciencias (PNAS ) el año pasado.
El artículo publicado en PNAS demostró que los eventos de "explosión" de intrones muy atrás en la historia evolutiva probablemente introdujeron miles de intrones en un genoma al mismo tiempo. Ares y Corbett-Detig ahora están trabajando para recrear artificialmente un evento de explosión, lo que les daría una idea de cómo reaccionaron los genomas cuando esto sucedió.
Ares dijo que su asociación interdisciplinaria con Corbett-Detig les ha abierto las puertas para profundizar realmente en algunos de los mayores misterios sobre los intrones que probablemente les sería imposible comprender completamente sin su experiencia combinada.
"Es la mejor manera de hacer las cosas", dijo Ares. "Cuando encuentras a alguien que tiene el mismo tipo de preguntas en mente pero con un conjunto diferente de métodos, perspectivas, prejuicios e ideas extrañas, eso se vuelve más emocionante. Eso te hace sentir que puedes salir y resolver un problema como este. lo cual es muy complejo."
Más información: Manuel Ares et al, Los esplicosomas de lazos de intrones convierten lazos en círculos verdaderos:implicaciones para la transposición de intrones, Genes y desarrollo (2024). DOI:10.1101/gad.351764.124
Información de la revista: Actas de la Academia Nacional de Ciencias , Genes y desarrollo
Proporcionado por la Universidad de California - Santa Cruz