Un estudio ha aprovechado el genoma del melocotón y ha creado redes de coexpresión genética (GCN) a gran escala que predicen las funciones de los genes y agilizan el proceso de mejoramiento del melocotón. Este enfoque innovador aborda la compleja tarea de identificar genes relacionados con rasgos genéticos deseables en los melocotones.
La genética del melocotón enfrenta desafíos como tiempos intergeneracionales prolongados y sistemas de transformación genética limitados, lo que hace que la identificación y validación de genes sean complejas. Los enfoques tradicionales a menudo no logran predecir las funciones genéticas debido a estas limitaciones.
Las redes de coexpresión genética (GCN), que mapean las relaciones entre las expresiones genéticas, ofrecen una solución prometedora. Al aprovechar el principio de "culpable por asociación", los GCN pueden predecir funciones genéticas basadas en patrones de expresión compartidos. Debido a estos desafíos, existe una necesidad apremiante de herramientas innovadoras como los GCN para mejorar nuestra comprensión de la genética del melocotón.
Investigadores del Institut de Recerca i Tecnologia Agroalimentàries (IRTA) y del Centre de Recerca en Agrigenòmica (CRAG) publican un estudio en la revista Horticulture Research el 2 de enero de 2024. El estudio presenta una nueva herramienta que aprovecha los GCN para predecir funciones genéticas en melocotones, revolucionando potencialmente el mejoramiento de frutas.
El estudio creó cuatro GCN a partir de 604 bibliotecas Illumina RNA-Seq y evaluó su rendimiento utilizando un algoritmo de aprendizaje automático basado en el principio de "culpable por asociación". Entre las redes, COO300 mostró el mejor rendimiento, abarcando 21.956 genes. Para validar COO300, los investigadores realizaron dos estudios de caso.
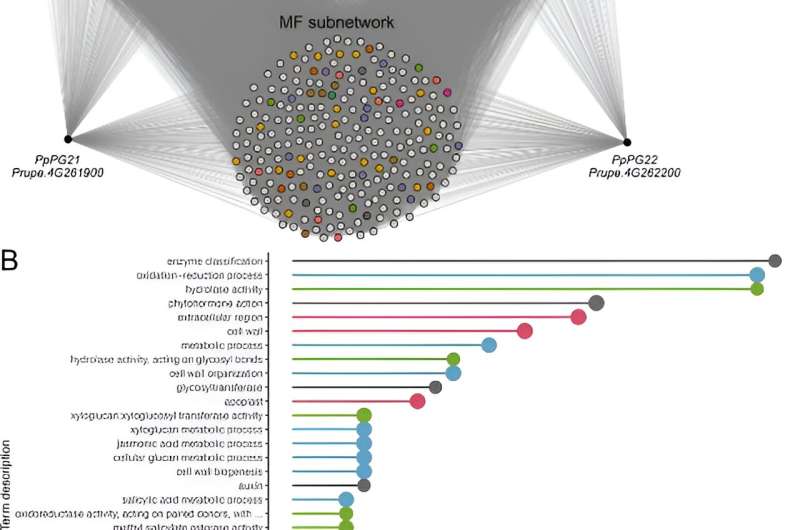
En el primer caso de estudio se analizaron los genes PpPG21 y PpPG22, implicados en el ablandamiento del fruto del melocotón. Los genes coexpresados formaron la red de carne derretida (MF), que se enriqueció con términos relacionados con la organización de la pared celular y la señalización de fitohormonas. La red MF incluyó genes asociados con el desmontaje de la pared celular y fitohormonas relacionadas con la maduración, lo que demuestra la capacidad de COO300 para predecir las funciones genéticas con precisión.
En el segundo estudio de caso se examinó el factor de transcripción PpMYB10.1, que regula la acumulación de antocianinas. La red de color de fruta (FC) identificó genes implicados en el metabolismo y la regulación de las antocianinas. Al comparar la red FC con ortólogos de Vinevine, los investigadores identificaron redes regulatorias conservadas, validando aún más las capacidades predictivas de COO300.
El Dr. Iban Eduardo, investigador destacado del CRAG, afirmó:"El desarrollo de la red COO300 representa un avance significativo en la genética del melocotón. Al predecir con precisión las funciones genéticas, esta herramienta no sólo mejora nuestra comprensión de la biología del melocotón, sino que también allana el camino para estrategias de reproducción más específicas y eficientes."
El estudio ofrece a los obtentores una poderosa herramienta para mejorar las variedades de melocotón. Al decodificar las funciones genéticas, se espera que PeachGCN v1.0 y sus scripts, accesibles para todos, impulsen avances en el sabor, la longevidad y la nutrición de los melocotones. Este conocimiento genómico es un presagio de cambio, que indica un giro hacia el mejoramiento genético basado en datos en la agricultura.
Más información: Felipe Pérez de los Cobos et al, Explorando redes de coexpresión genética a gran escala en melocotón (Prunus persica L.):una nueva herramienta para predecir la función genética, Horticulture Research (2024). DOI:10.1093/hora/uhad294
Información de la revista: Investigación en horticultura
Proporcionado por la Universidad Agrícola de NanJing