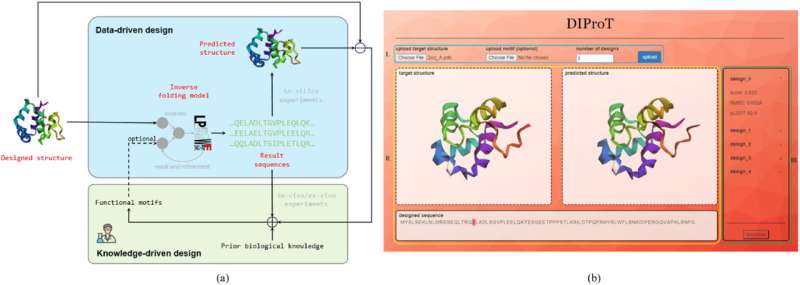
Los científicos han desarrollado DIProT, un conjunto de herramientas innovador y fácil de usar para el diseño de proteínas. El kit de herramientas utiliza un modelo generativo profundo no autorregresivo para abordar el problema del plegamiento inverso de proteínas, integrando la experiencia humana en el ciclo de diseño para un diseño de proteínas eficiente y efectivo.
El diseño de proteínas, un aspecto crucial de las ciencias biológicas, implica la creación de secuencias de aminoácidos que se pliegan en las estructuras proteicas deseadas. Este proceso, conocido como problema de plegamiento inverso de proteínas, ha sido un desafío en el campo.
Con ese fin, un equipo de investigadores de la Universidad de Tsinghua (THU) en China presentó DIProT, un conjunto de herramientas interactivo de diseño de proteínas que aprovecha un modelo generativo profundo no autorregresivo para abordar este problema.
"Las proteínas desempeñan un papel crucial en numerosas funciones biológicas", explica el autor correspondiente del estudio, Xiaowo Wang, profesor del Departamento de Automatización de la Universidad de Tsinghua. "Tanto predecir la estructura de una secuencia proteica determinada, como lo ejemplifica AlphaFold, como diseñar secuencias de aminoácidos que se ajusten a una estructura proteica determinada plantean desafíos únicos".
Para desarrollar DIProT, los investigadores integraron modelos de aprendizaje profundo con experiencia humana directamente en el proceso de diseño, mejorando así la eficiencia y eficacia del diseño de proteínas.
"El enfoque único de DIProT permite a los usuarios especificar la estructura objetivo y arreglar partes de la secuencia que desean preservar, mejorando la flexibilidad del proceso de diseño", añade Wang. "El conjunto de herramientas también incorpora un modelo de predicción de la estructura de proteínas para evaluar diseños in silico, formando un bucle de diseño virtual que mejora significativamente la eficiencia del diseño de proteínas".
Una de las características clave de DIProT es su interfaz gráfica de usuario (GUI) fácil de usar, que integra múltiples algoritmos para facilitar un ciclo de diseño de retroalimentación rápido e intuitivo. La GUI permite a los usuarios interactuar visualmente con los resultados del diseño, lo que ayuda a comprender e interpretar los resultados.
Los autores, que publicaron su estudio en la revista Synthetic and Systems Biotechnology , espere que DIProT sea muy útil para tareas prácticas de diseño de proteínas. "Esperamos que DIProT estimule más investigaciones en este campo y sirva como una herramienta útil para abordar desafíos de diseño de proteínas cada vez más complejos y diversos".
Los investigadores planean perfeccionar su modelo de plegamiento inverso y su conjunto de herramientas para abordar desafíos de diseño de proteínas cada vez más complejos y diversos en el futuro.
Más información: Jieling He et al, DIProT:un conjunto de herramientas interactivo basado en aprendizaje profundo para el diseño de proteínas, biotecnología sintética y de sistemas eficiente y eficaz (2024). DOI:10.1016/j.synbio.2024.01.011
Proporcionado por KeAi Communications Co., Ltd.