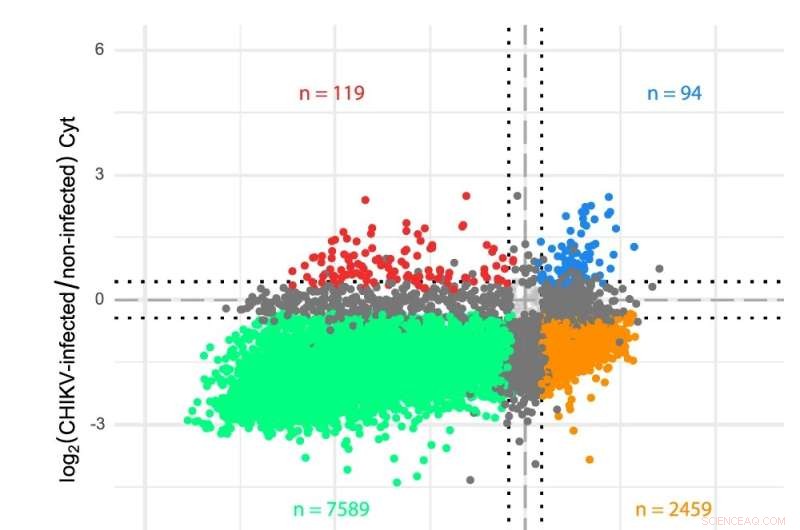
un esquema del genoma del ARN de CHIKV. b Análisis de transcriptoma del citosol (Cyt) y compartimentos ER en células infectadas por CHIKV. Los genes se trazaron según sus cambios log2 en células infectadas con CHIKV frente a células no infectadas para ambos compartimentos de traducción. Las lecturas de secuenciación se analizaron utilizando una anotación de transcriptoma personalizada y la expresión diferencial se determinó a través de limma-voom (ver Métodos). Los colores indican genes que están significativamente (FDR < 0.05) regulados a la baja en ambos compartimentos (verde), regulados al alza en ambos compartimentos (azul), regulados al alza o a la baja en un solo compartimento (gris oscuro) o muestran un comportamiento opuesto en los dos compartimentos (naranja y rojo). Las líneas punteadas indican cambios log2 de ±0,433 (es decir, un cambio del 35 % en cualquier dirección) utilizados como umbral mínimo para declarar que los niveles de ARNm han cambiado. Se infectaron células HEK 293T a una MOI de 4 y se recogieron muestras doce horas después de la infección. Se realizaron tres réplicas independientes para cada condición. Crédito:Comunicaciones de la naturaleza DOI:10.1038/s41467-022-31835-x
El Grupo de Investigación en Virología Molecular de la Universidad Pompeu Fabra (UPF), en colaboración con el grupo de Epitranscriptómica y Dinámica del ARN del Centro de Regulación Genómica (CRG), ha descubierto un nuevo mecanismo por el que los virus modifican la maquinaria celular para leer mejor las instrucciones del genoma del virus invasor y así producir grandes cantidades de progenie viral. El estudio ha sido publicado en Nature Communications y estuvo dirigido por Juana Díez.
Los genes contienen la información requerida para la formación de proteínas, moléculas complejas que son esenciales para la vida, formadas a partir de aminoácidos. La lectura de esta información tiene lugar en dos etapas principales, siendo la primera la transcripción, en la que la información del gen (ADN) se transfiere a una molécula llamada ARN mensajero (ARNm). El ARNm consiste en un "texto" formado por tripletes de nucleótidos (las letras GCT, CAT, etc.). Cada triplete corresponde a un aminoácido. La segunda fase es la traducción, en la que una molécula llamada ARN de transferencia (ARNt) reconoce cada triplete y actúa como traductor trayendo el aminoácido correspondiente. Las proteínas se construyen a través de este proceso.
Hay 61 codones y 20 aminoácidos, y tantos tripletes codifican el mismo aminoácido. Cada organismo utiliza preferentemente uno de estos tripletes (triplete óptimo) porque tiene una mayor concentración del tRNA que reconoce ese triplete. Así, cuando el "texto" del mRNA está enriquecido en tripletes óptimos, las proteínas se generarán rápida y eficientemente mientras que cuando están enriquecidos en tripletes no óptimos, la eficiencia de la expresión disminuirá porque los tRNA relacionados son escasos.
Los virus son muy simples y para multiplicarse y expresar sus proteínas necesitan secuestrar la maquinaria celular del huésped. Los virus generan su propio ARNm en las células que infectan, que estas últimas leen y generan proteínas virales para producir más virus. Pero los ARNm de muchos virus, incluido el SARS-CoV-2 y los virus transmitidos por los mosquitos dengue, zika y chikungunya, están enriquecidos en tripletes no óptimos y aún expresan proteínas virales con gran eficacia. "Para abordar este dilema, hemos utilizado el virus chikungunya como modelo porque su genoma se multiplica a niveles extremadamente altos", explican Jennifer Jungfleisch y René Böetcher, coautores del estudio.
“Nuestros hallazgos muestran por primera vez que los virus modifican el ARNt del huésped para adaptar la maquinaria de traducción del huésped al texto del ARNm viral”, dice Marc Talló, también coautor del artículo. “En otras palabras, la infección viral induce un cambio de lenguaje en la célula, de modo que expresa las proteínas virales de manera muy eficiente. Como las proteínas virales son esenciales para la producción de virus, en última instancia, este cambio será el responsable de generar una gran cantidad de virus. en la célula infectada", añade.
“Aunque el estudio se ha centrado en el virus chikungunya, nuestra propuesta es que la modificación de los tRNA inducida por la infección viral es un mecanismo general seguido por muchos virus”, explica Juana Díez, catedrática del Departamento de Medicina y Ciencias de la Vida de la UPF.
“Además, nuestros resultados proporcionan una base para considerar la regulación del ARNt como una nueva y prometedora diana terapéutica para el desarrollo de antivirales de amplio espectro que sean efectivos contra múltiples virus”, concluye Díez. En el estudio también ha participado el grupo de investigación coordinado por Eva María Novoa en el CRG, y los otros autores son Gemma Pérez-Vilaró y Andres Merits (Instituto Tecnológico, Universidad de Tartu). Los virus se adaptan al 'lenguaje de las células humanas' para secuestrar la síntesis de proteínas