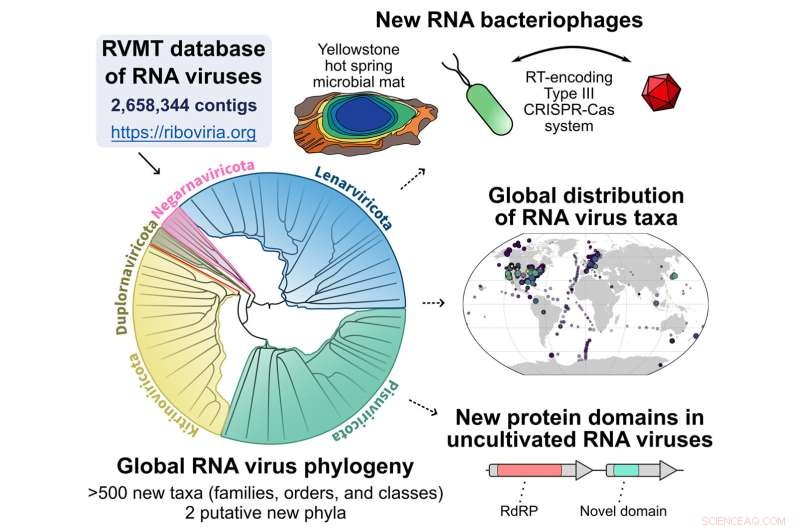
Resumen gráfico de la tubería que comienza con la base de datos de metatranscriptomas de virus de ARN (RVMT) para descubrir la expansión en la diversidad de virus de ARN. Crédito:Simón Roux
Una vez, un zoológico ofreció un libro para colorear con osos polares en escenas invernales que venía con crayones en varios tonos de blanco. Para los investigadores que buscan secuencias de virus de ARN en grandes conjuntos de datos, su trabajo puede ser similar a encontrar un solo copo de nieve en una página coloreada de ese libro.
Publicado en línea el 28 de septiembre de 2022, en Celda , un equipo dirigido por investigadores de la Universidad de Tel Aviv en Israel, el Centro Nacional de Información Biotecnológica y el Instituto Conjunto del Genoma (JGI) del Departamento de Energía de EE. UU. (DOE), una instalación para usuarios de la Oficina de Ciencias del DOE ubicada en el Laboratorio Nacional Lawrence Berkeley ( Berkeley Lab) describen una tubería computacional que puede escanear específicamente esos copos de nieve o secuencias de virus de ARN. Con este flujo de trabajo, el equipo analizó más de 5000 conjuntos de datos de secuencias de ARN (metatranscriptomas) generados a partir de diversas muestras ambientales de todo el mundo, lo que resultó en un aumento de cinco veces en la diversidad de virus de ARN.
"El mundo de los virus que nos rodea es enorme y ahora tenemos los medios para explorarlo", dijo Eugene Koonin, investigador principal del NCBI y uno de los autores principales del artículo, sobre la diversidad viral descubierta. "Aunque los desafíos técnicos del análisis de datos a esta escala son formidables".
Tamices computacionales para filtrar secuencias
Hay más microbios en el planeta que partículas en un puñado de tierra, y los virus superan en número a los microbios. Los avances en tecnologías de secuenciación y herramientas computacionales han descubierto una diversidad de virus que infectan no solo cultivos, animales y humanos, sino también microbios cuya presencia o ausencia puede afectar los ciclos de nutrientes del planeta.
Mientras que la información genética de la mayoría de los organismos está codificada en el ADN, con el ARN entregando las instrucciones dentro del ADN a la célula, los virus de ARN almacenan su información genética en el ARN sin una etapa de ADN. "Yo diría que los virus de ARN a nivel mundial son incluso menos conocidos que los virus de ADN", dijo Simon Roux, científico de JGI y uno de los codirectores del proyecto. "Pero al igual que los virus de ADN, los virus de ARN infectan microbios en todo el mundo y provocan la muerte celular y/o cambios profundos en la fisiología celular durante la infección".
Si bien todos los virus de ARN tienen un gen que codifica una enzima llamada ARN polimerasa dirigida por RNS (RdRP), necesaria para replicar la replicación del genoma de ARN, detectarlo ha sido un desafío. Encontrar los copos de nieve del virus de ARN en la tormenta de nieve de datos genómicos implicó el desarrollo de tamices computacionales especiales para filtrar secuencias que era poco probable que contuvieran la secuencia RdRP.
El trabajo fue el resultado de una colaboración a tres bandas que comenzó en 2019, recordó Uri Neri de la Universidad de Tel Aviv, uno de los codirectores del proyecto y primer autor del estudio. Los miembros de los equipos de Tel Aviv y NCBI, que ya estaban trabajando juntos en la minería de virus procarióticos, supieron de Nikos Kyrpides de JGI que su grupo de Microbiome Data Science también estaba trabajando en la minería de virus de ARN. Después de un par de reuniones virtuales de los tres equipos, quedó claro que un esfuerzo de colaboración más grande sería mucho más efectivo para lograr resultados de mayor calidad en comparación con esfuerzos individuales más pequeños. Este es también el tipo de espíritu comunitario colaborativo y sinérgico que JGI defiende y promueve activamente.
El equipo utilizó todos los conjuntos de datos de metatranscriptomas disponibles públicamente del sistema Integrated Microbial Genomes &Microbiomes (IMG/M) de JGI. "Luego analizamos muchas más muestras y refinamos nuestra metodología", dijo Neri. "Nuestro equipo creció y también lo hizo el alcance del proyecto". Con este fin, enfatizó Kyrpides, las contribuciones de los numerosos usuarios científicos de JGI en la recolección y el envío de sus muestras de microbioma para la secuenciación en el JGI no pueden subestimarse. Su cooperación y apoyo, dijo, y en varios casos, su permiso para usar datos de secuencias aún no publicados, fue absolutamente fundamental para el éxito de este esfuerzo, al igual que el reconocimiento de su contribución.
Tanto Roux como Koonin señalaron que la plétora de secuencias de virus de ARN descubiertas "cambia significativamente la visión global de la diversidad de virus", aunque no en las clasificaciones de nivel superior de los grupos de virus (phyla). Las nuevas secuencias están llenando algunos vacíos en los virus existentes. grupos al mismo tiempo que agrega nuevas sucursales. Además, los virus de ARN no parecen estar distribuidos uniformemente en todo el mundo.
Un grupo ampliado es el de virus asociados con bacterias; hasta ahora, la mayoría de los virus de ARN conocidos se han asociado con eucariotas. Junto con la expansión de los virus de ARN asociados con las bacterias, se encuentra el hallazgo de que "algunas bacterias usan CRISPR para defenderse del ARN", anotó Roux, "aunque no está claro por qué esto se detecta tan raramente".
Desarrollo de enfoques para conciliar Big Data 'real'
Para el equipo, el trabajo computacional que condujo a la abundancia descubierta de virus de ARN es solo el comienzo. "A menudo digo que solo identificar una secuencia como viral no es ni la mitad de la historia". Neri dijo. "Invertimos muchos de nuestros esfuerzos en los análisis posteriores al descubrimiento; lo mejor que pudimos, tratamos de describir los dominios de proteínas que lleva cada virus y quién es su probable huésped. Hemos hecho que toda esa información sea totalmente gratuita y abierta. disponible para la comunidad científica en general".
Uri Gophna de la Universidad de Tel Aviv y Koonin señalaron que otras investigaciones en paralelo han informado "expansiones dramáticas" similares del viroma de ARN global. "Ahora necesitamos comparar y reconciliar los hallazgos, generando un conjunto de datos único y no redundante", dijo Koonin. "Con suerte, relativamente pronto podremos estimar el tamaño real del viroma de ARN. Sin embargo, esto ahora es Big Data real, estamos tratando con miles de millones de secuencias, y pronto, con billones. El desarrollo de enfoques eficientes y automatizados para analizar y clasificar los datos de secuencia a esta escala es esencial". Una herramienta automatizada para evaluar la calidad de los datos de virus