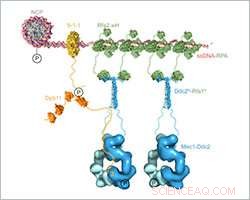
Modelo estructural compuesto a escala del ensamblaje y activación de Mec1-Ddc2-RPA en los sitios de daño del ADN. Crédito:Instituto Friedrich Miescher de Investigación Biomédica
Ishan Deshpande y sus colegas del grupo de Susan Gasser y la instalación de estructura de proteínas en el FMI dilucidaron el mecanismo de cómo Mec1-Ddc2, un supresor de tumores involucrado en la respuesta al daño del ADN se ensambla en los sitios de daño del ADN. Se centran en la interacción Ddc2-RPA, que lleva el complejo de quinasa Mec1-Ddc2 al ADN monocatenario en los sitios de daño del ADN. Con sus datos estructurales, los científicos del FMI proporcionan el eslabón perdido entre dos estructuras publicadas anteriormente, permitiéndoles ensamblar un modelo estructural compuesto a escala de Mec1-Ddc2 en ssDNA-RPA en los sitios de daño del ADN.
La quinasa ATR ha atraído interés durante más de 20 años. Esta quinasa es una de las dos quinasas centrales que detectan daños en el ADN en células de mamíferos, y también es un supresor de tumores. Dos compuestos que inhiben la quinasa ATR se encuentran en ensayos clínicos para el tratamiento de la leucemia y los tumores sólidos. Todavía, cómo se regula esta enzima a nivel molecular sigue siendo difícil de alcanzar.
Ishan Deshpande, un estudiante de doctorado en la instalación de estructura de proteínas y el grupo de Susan Gasser en el Instituto Friedrich Miescher de Investigación Biomédica (FMI), ahora ha dilucidado el mecanismo de cómo el homólogo de levadura de ATR, Mec1, se ensambla en los sitios de daño del ADN para la activación específica del sitio.
En levadura como en los humanos, Mec1 forma un complejo con una subunidad reguladora llamada Ddc2 (ATRIP en humanos). Tras el daño del ADN, se exponen grandes tramos de ADN monocatenario (ADNss), que son rápidamente cubiertos por la proteína de replicación A (RPA). Mec1-Ddc2 reconoce estos estiramientos de ssDNA-RPA y se acumula en estos sitios para iniciar los mecanismos de reparación.
Deshpande y sus colegas ahora han resuelto la estructura 3-D del extremo N de Ddc2 en asociación con parte de RPA, proporcionando así datos estructurales sobre el vínculo entre RPA y Mec1, que hasta ahora había sido difícil de alcanzar. Obtuvimos una estructura cocristalina de alta resolución del extremo N de Ddc2 junto con una subunidad de RPA, Deshpande explicó. Con estos datos, podríamos demostrar que Ddc2 forma homodímeros y se une a RPA por su extremo N-terminal. Curiosamente, nuestra estructura Ddc2-RPA es el eslabón perdido entre dos estructuras publicadas anteriormente, y esto nos permite ensamblar un modelo estructural compuesto a escala de Mec1-Ddc2 en ssDNA-RPA en sitios de daño del ADN.
El extremo N de Ddc2 no solo facilita la interacción de Mec1 con RPA, sino que también funciona como espaciador de la quinasa Mec1. Deshpande explica:El extremo N-terminal alargado de Ddc2 permite que el gran módulo de quinasa Mec1 se mueva sin encontrar el ADN dañado ni la maquinaria de reparación que trabaja en la lesión. Además, el espaciador Ddc2 puede permitir que Mec1 fosforile múltiples sustratos espacialmente distintos mientras permanece unido al sitio del daño del ADN. Puede pensar en el extremo N de Ddc2 como el cuello de una jirafa que permite que la jirafa alcance la hierba del suelo y las hojas de un árbol alto.
La actividad adecuada de Mec1-Ddc2 es importante para todo tipo de reparación de daños en el ADN. Sin embargo, los datos funcionales de Deshpande y sus colegas indican que el extremo N de Ddc2 es particularmente importante para la supervivencia celular después del daño del ADN por la luz ultravioleta. Nuestro modelo sugiere que el mero reclutamiento de Mec1-Ddc2 a ssDNA puede contribuir a su homodimerización y por lo tanto a la activación del complejo Mec1-Ddc2, explica Deshpande.
Además, Susan Gasser señala "que esta ha sido una colaboración maravillosa entre mi laboratorio y las instalaciones de estructura del FMI, permitiéndonos pasar de la estructura a la función ".