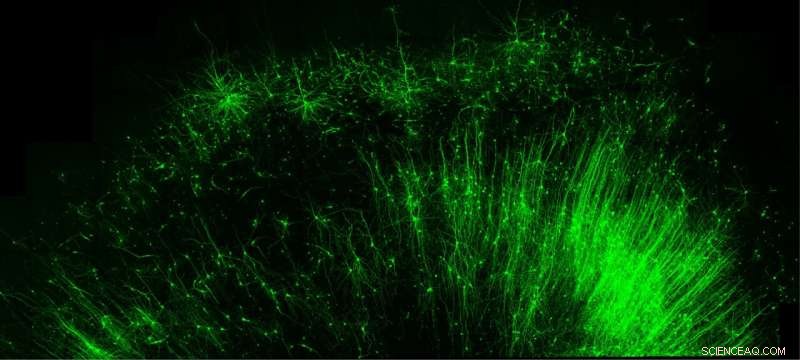
Etiquetar e iluminar solo las células inhibidoras de "freno" (verde) en el tejido cerebral humano es solo una de las muchas cosas que puede hacer la nueva herramienta de la Universidad de Duke, CellREADR. Crédito:Derek Southwell, Universidad de Duke
Investigadores de la Universidad de Duke han desarrollado una herramienta de edición basada en ARN que se dirige a células individuales, en lugar de genes. Es capaz de apuntar con precisión a cualquier tipo de célula y agregar selectivamente cualquier proteína de interés.
Los investigadores dijeron que la herramienta podría permitir modificar células y funciones celulares muy específicas para controlar enfermedades.
Usando una sonda basada en ARN, un equipo dirigido por el neurobiólogo Z. Josh Huang, Ph.D. y el investigador postdoctoral Yongjun Qian, Ph.D. demostraron que pueden introducir etiquetas fluorescentes en las células para marcar tipos específicos de tejido cerebral; un interruptor de encendido/apagado sensible a la luz para silenciar o activar las neuronas de su elección; e incluso una enzima de autodestrucción para eliminar con precisión algunas células pero no otras. La obra aparece el 5 de octubre en Nature .
Su sistema de control y seguimiento celular selectivo se basa en la enzima ADAR, que se encuentra en las células de todos los animales. Si bien estos son los primeros días de CellREADR (acceso celular a través de la detección de ARN por ADAR endógeno), las posibles aplicaciones parecen ser infinitas, dijo Huang, al igual que su potencial para funcionar en todo el reino animal.
"Estamos entusiasmados porque esto proporciona una tecnología simplificada, escalable y generalizable para monitorear y manipular todos los tipos de células en cualquier animal", dijo Huang. "En realidad, podríamos modificar tipos específicos de función celular para controlar enfermedades, independientemente de su predisposición genética inicial", dijo. "Eso no es posible con las terapias o medicamentos actuales".
CellREADR es una cadena personalizable de ARN compuesta por tres secciones principales:un sensor, una señal de alto y un conjunto de planos.
En primer lugar, el equipo de investigación decide qué tipo de célula específica quiere investigar e identifica un ARN objetivo que es producido únicamente por ese tipo de célula. La notable especificidad tisular de la herramienta se basa en el hecho de que cada tipo de célula fabrica ARN característico que no se ve en otros tipos de células.
A continuación, se diseña una secuencia sensora como la cadena complementaria del ARN diana. Así como los peldaños del ADN están formados por moléculas complementarias que se atraen de forma inherente entre sí, el ARN tiene el mismo potencial magnético para unirse con otra pieza de ARN si tiene moléculas coincidentes.
Después de que un sensor ingresa a una célula y encuentra su secuencia de ARN objetivo, ambas piezas se juntan para crear una pieza de ARN de doble cadena. Este nuevo mashup de ARN activa la enzima ADAR para inspeccionar la nueva creación y luego cambiar un solo nucleótido de su código.
The ADAR enzyme is a cell-defense mechanism designed to edit double-strand RNA when it occurs, and is believed to be found in all animal cells.
Knowing this, Qian designed CellREADR's stop sign using the same specific nucleotide ADAR edits in double-stranded RNA. The stop sign, which prevents the protein blueprints from being built, is only removed once CellREADR's sensor docks to its target RNA sequence, making it highly specific for a given cell type.
Once ADAR removes the stop sign, the blueprints can be read by cellular machinery that builds the new protein inside the target cell.
In their paper, Huang and his team put CellREADR through its paces. "I remember two years ago when Yongjun built the first iteration of CellREADR and tested it in a mouse brain," Huang said. "To my amazement, it worked spectacularly on his first try."
The team's careful planning and design paid off as they were then able to demonstrate CellREADR accurately labelled specific brain cell populations in living mice, as well as effectively added activity monitors and control switches where directed. It also worked well in rats, and in human brain tissue collected from epilepsy surgeries.
"With CellREADR, we can pick and choose populations to study and really begin to investigate the full range of cell types present in the human brain," said co-author Derek Southwell, M.D., Ph.D., a neurosurgeon and assistant professor in the department of neurosurgery at Duke.
Southwell hopes CellREADR will improve his and others' understanding of the wiring diagram for human brain circuits and the cells within them, and in doing so, help advance new therapies for neurological disorders, such as a promising new method to treat drug-resistant epilepsy he is piloting.
Huang and Qian are especially hopeful about CellREADR's potential as a "programmable RNA medicine" to possibly cure diseases—since that's what drew them both to science in the first place. They have applied for a patent on the technology.
"When I majored in pharmacology as an undergraduate, I was very naïve," Qian said. "I thought you could do a lot of things, like cure cancer, but actually it's very difficult. However, now I think, yes maybe we can do it." New technology identifies molecular properties of cells and maps their location within tissues