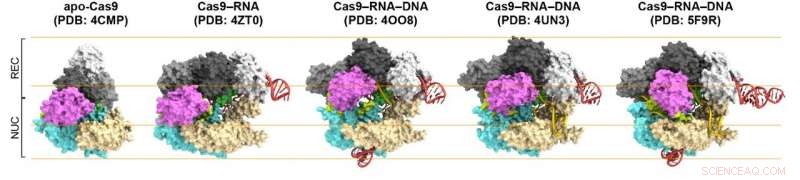
Figura 1. Estructuras de Cas9. De izquierda a derecha:Cas9 solo (apo-Cas9), Cas9 unido al ARN (Cas9 – ARN), Cas9 – ARN unido a su objetivo de ADN monocatenario (Cas9 – ARN – ADN), Cas9 – ARN unido a un dúplex de ADN parcial (Cas9 – ARN – ADN) y Cas9 – ARN unido a su objetivo de ADN de doble hebra (Cas9 – ARN – ADN). Crédito:Universidad de Kanazawa
Investigadores de la Universidad de Kanazawa y la Universidad de Tokio informan en Comunicaciones de la naturaleza la visualización de la dinámica de las 'tijeras moleculares', el mecanismo principal de la técnica de ingeniería genética CRISPR-Cas9.
Una de las técnicas utilizadas en ingeniería genética, el proceso de modificación artificial del genoma de un organismo vivo, involucra el llamado sistema de nucleasa CRISPR-Cas9. Usando este sistema, el ADN de una célula se puede cortar en un sitio deseado, donde los genes se pueden eliminar o agregar. La selección del sitio a cortar se realiza mediante una molécula de "ARN guía" unida a la proteína Cas9. Ahora, un equipo de investigadores dirigido por Mikihiro Shibata de la Universidad de Kanazawa y Osamu Nureki de la Universidad de Tokio ha visualizado la dinámica del complejo CRISPR-Cas9, en particular, cómo corta el ADN, proporcionando información valiosa sobre el mecanismo de escisión del ADN mediado por CRISPR-Cas9.
Para sus estudios de visualización, los científicos utilizaron microscopía de fuerza atómica de alta velocidad (HS-AFM), un método para obtener imágenes de superficies. Una superficie se palpa moviendo un pequeño voladizo sobre ella; la fuerza experimentada por la sonda se puede convertir en una medida de altura. Un escaneo de toda la superficie da como resultado un mapa de altura de la muestra. La configuración experimental de alta velocidad de Shibata y sus colegas permitió exploraciones repetidas, convertibles en películas, de las biomoléculas que participan en la acción de tijera molecular.
Primero, los científicos compararon Cas9 sin y con ARN adjunto (Cas9-ARN). Descubrieron que el primero podía adoptar de manera flexible varias conformaciones, mientras que este último tiene un fijo, estructura de dos lóbulos, destacando la capacidad de estabilización conformacional del ARN guía. Luego, Shibata y sus colegas observaron cómo el complejo Cas9-ARN estabilizado se dirige al ADN. Confirmaron que se une a un sitio de motivo adyacente protoespaciador (PAM) preseleccionado en el ADN. Un PAM es una secuencia de nucleótidos corta ubicada junto al sitio objetivo del ADN, que es complementario al ARN guía.
Las películas de alta velocidad del equipo de investigación revelaron además que la focalización ("interrogación del ADN") se logra mediante la difusión en 3-D del complejo Cas9-ARN. Finalmente, los investigadores lograron visualizar la dinámica del proceso de escisión en sí:observaron cómo la región de las 'tijeras moleculares' sufre fluctuaciones conformacionales después de que Cas9-ARN desenrolla localmente el ADN de doble hebra (Película 1 [URL]).
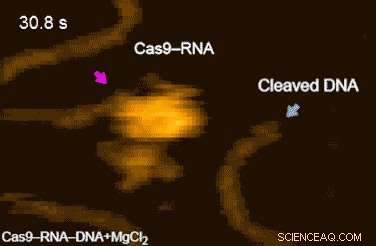
Figura 2. Películas HS-AFM de escisión de ADN por Cas9-RNA. Las fluctuaciones del dominio de nucleasa se indican mediante flechas magenta. Los productos de escisión liberados de Cas9-RNA se indican mediante flechas azules. Crédito:Universidad de Kanazawa
El trabajo de Shibata avanza nuestra comprensión del mecanismo de edición del genoma CRISPR-Cas9. En palabras de los investigadores:"... este estudio proporciona detalles sin precedentes sobre la dinámica funcional de CRISPR-Cas9, y destaca el potencial de HS-AFM para dilucidar los mecanismos de acción de nucleasas efectoras guiadas por ARN de distintos sistemas CRISPR-Cas ".
CRISPR-Cas9
CRISPR, abreviatura de "repeticiones palindrómicas cortas agrupadas regularmente interespaciadas", se refiere a un conjunto de secuencias de ADN bacteriano que contienen fragmentos del ADN de virus que han atacado anteriormente a las bacterias. Estos fragmentos son utilizados por las bacterias para prevenir futuros ataques de los mismos virus. "Cas" se refiere a genes asociados a CRISPR; "Cas9" es una proteína asociada a CRISPR con dos dominios nucleasa (una nucleasa es una enzima capaz de escindir ácidos nucleicos, moléculas orgánicas presentes en el ADN y el ARN).
En años recientes, se ha desarrollado una técnica de ingeniería genética en la que un complejo CRISPR-Cas9 actúa como 'tijeras moleculares'; la nucleasa Cas9 se une a una molécula de ARN guía que contiene información sobre el sitio del ADN al que se dirige. Usando microscopía de fuerza atómica de alta velocidad, Mikihiro Shibata de la Universidad de Kanazawa y sus colegas ahora han estudiado la dinámica del complejo CRISPR-Cas9 con gran detalle.
Fuerza atómica microscópica
La microscopía de fuerza atómica (AFM) es una técnica de imagen en la que la imagen se forma escaneando una superficie con una punta muy pequeña. El movimiento de exploración horizontal de la punta se controla mediante elementos piezoeléctricos, while vertical motion is converted into a height profile, resulting in a height distribution of the sample's surface. As the technique does not involve lenses, its resolution is not restricted by the so-called diffraction limit. In a high-speed setup, AFM can be used to produce movies of a sample's evolution in real time. High-speed AFM has been used successfully to study protein dynamics, for example myosin V walking on an actin filament, the photo-induced conformational change of bacteriorhodopsin, and the degradation of cellulose. Shibata and colleagues have now applied the high-speed AFM technique for visualizing the dynamics of DNA cleavage by CRISPR-Cas9.