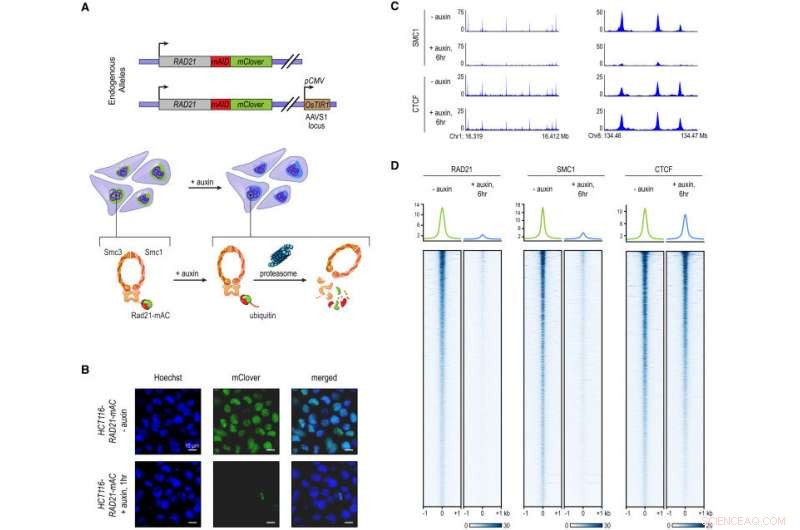
El etiquetado de RAD21 endógeno con un Degron inducible por auxina permite Pérdida de cohesina casi completa. (A) En células HCT-116-RAD21-mAC, ambos alelos RAD21 están marcados con degrons inducibles por auxina y un reportero mClover, y el gen OsTIR1 está integrado en el locus AAVS1. El tratamiento con auxinas conduce a la degradación proteasomal de RAD21. (B) Imágenes de células vivas después de la tinción Hoechst 33342 para marcar núcleos. La fluorescencia nuclear de mClover correspondiente al RAD21 marcado se perdió después de 1 hora de tratamiento con auxina. (C) Señal ChIP-seq SMC1 y CTCF con y sin tratamiento con auxina. (D) RAD21, SMC1, y señal CTCF ChIP-seq (izquierda, medio, derecha) en todos los picos llamados para cada una de las proteínas en las células RAD21-mAC no tratadas. Arriba:Enriquecimientos promedio para cada proteína. Después de la degradación de RAD21, el complejo de cohesina ya no se une a la cromatina. La unión de CTCF no se ve afectada. Crédito: Celda . DOI:10.1016 / j.cell.2017.09.026
Un equipo multiinstitucional que abarca la Facultad de Medicina de Baylor, Universidad de Rice, La Universidad de Stanford y el Instituto Broad del MIT y Harvard han creado el primer mapa 4-D de alta resolución del plegamiento del genoma, rastreando un genoma humano completo a medida que se pliega con el tiempo. El informe, que puede conducir a nuevas formas de entender las enfermedades genéticas, aparece en la portada de Celda .
Hacer conexiones
Por décadas, Los investigadores han sospechado que cuando una célula humana responde a un estímulo, Los elementos del ADN que se encuentran muy separados en el genoma se encuentran rápidamente entre sí, formando bucles a lo largo del cromosoma. Al reorganizar estos elementos de ADN en el espacio, la célula puede cambiar qué genes están activos.
En 2014, el mismo equipo de científicos demostró que era posible mapear estos bucles. Pero los primeros mapas eran estáticos, sin la capacidad de ver el cambio de bucles. No estaba claro si, en el abarrotado espacio del núcleo, Los elementos del ADN podrían encontrarse entre sí lo suficientemente rápido como para controlar las respuestas celulares.
"Antes, podríamos hacer mapas de cómo se plegaba el genoma cuando estaba en un estado particular, pero el problema con una imagen estática es que si nada cambia, es difícil averiguar cómo funcionan las cosas, "dijo Suhas Rao, primer autor del nuevo estudio. "Nuestro enfoque actual es más como hacer una película; podemos ver los pliegues a medida que desaparecen y reaparecen".
Un anillo para gobernarlos a todos
Para realizar un seguimiento del proceso de plegado a lo largo del tiempo, el equipo de investigación comenzó por interrumpir la cohesión, un complejo de proteínas en forma de anillo que se encontraba en los límites de casi todos los bucles conocidos. En 2015, el equipo propuso que la cohesina crea bucles de ADN en el núcleo celular mediante un proceso de extrusión.
"La extrusión funciona como el ajustador de la longitud de la correa en una mochila, "explicó el Dr. Erez Lieberman Aiden, director del Centro de Arquitectura del Genoma de Baylor College of Medicine y autor principal del nuevo estudio. "Cuando pasa la correa por cualquier lado, forma un bucle. El ADN parece estar haciendo lo mismo, excepto que los anillos de cohesina parecen desempeñar el papel de ajustador ".
Aiden dijo que una predicción crucial del modelo de 2015 es que todos los bucles deberían desaparecer en ausencia de cohesión. En la nueva investigación, Aiden, Rao y sus colegas probaron esa suposición.
"Descubrimos que cuando interrumpimos cohesin, miles de bucles desaparecieron, "dijo Rao, estudiante de medicina en la Universidad de Stanford, Becario de Hertz y miembro del laboratorio de Aiden. "Luego, cuando volvemos a poner cohesin, todos esos bucles volvieron, a menudo en cuestión de minutos. Esto es precisamente lo que predeciría a partir del modelo de extrusión, y sugiere que la velocidad a la que la cohesina se mueve a lo largo del ADN es una de las más rápidas para cualquier proteína humana conocida ".
Bucles versus grupos
Pero no todo sucedió como esperaban los investigadores. En algunos casos, los bucles hicieron exactamente lo contrario de lo que anticiparon los investigadores.
"A medida que observamos que miles de bucles en el genoma se debilitaban, notamos un patrón gracioso, "dijo Aiden, también un becario de McNair, Miembro de Hertz e investigador principal del Centro de Física Biológica Teórica de la Universidad de Rice. "Hubo algunos bucles extraños que en realidad se estaban volviendo más fuertes. Entonces, mientras volvemos a poner cohesin, la mayoría de los bucles se recuperaron por completo, pero estos extraños bucles volvieron a hacer lo contrario:¡desaparecieron! "
Al analizar cómo cambiaron los mapas a lo largo del tiempo, el equipo se dio cuenta de que la extrusión no era el único mecanismo que unía los elementos del ADN. Un segundo mecanismo, llamada compartimentación, no involucró cohesin.
"El segundo mecanismo que observamos es bastante diferente a la extrusión, "explicó Rao." La extrusión tiende a unir los elementos del ADN de dos en dos, y solo si se encuentran en el mismo cromosoma. Este otro mecanismo puede conectar grandes grupos de elementos entre sí, incluso si se encuentran en diferentes cromosomas. Y parece ser tan rápido como la extrusión ".
Eric Lander, director del Broad Institute, un coautor del estudio, dijo, "Estamos comenzando a comprender las reglas por las cuales los elementos del ADN se unen en el núcleo. Ahora que podemos rastrear los elementos a medida que se mueven en el tiempo, los mecanismos subyacentes están empezando a aclararse ".