Al unir moléculas, los científicos de la Universidad de Montreal creen haber descubierto cómo los sistemas moleculares en el origen de la vida evolucionaron para crear funciones complejas de autorregulación.
Publicado en Angewandte Chemie , sus hallazgos prometen proporcionar a los químicos y nanotecnólogos una estrategia simple para crear la próxima generación de nanosistemas dinámicos.
La vida en la Tierra se sustenta en millones de pequeñas nanoestructuras o nanomáquinas diferentes que han evolucionado a lo largo de millones de años, explicó Alexis Vallée-Bélisle, profesor de la UdeM e investigador principal del estudio.
Estas estructuras, a menudo más pequeñas que 10.000 veces el diámetro de un cabello humano, suelen estar compuestas de proteínas o ácidos nucleicos. Si bien algunos están hechos de un solo componente o pieza (a menudo polímeros lineales que se pliegan en una estructura específica), la mayoría de ellos se fabrican utilizando varios componentes que se ensamblan espontáneamente en conjuntos grandes y dinámicos.
"Estos conjuntos moleculares son muy dinámicos y se activan o desactivan precisamente en respuesta a diversos estímulos, como una variación de temperatura, oxígeno o nutrientes", afirmó Vallée-Bélisle.
"De manera similar a los automóviles que requieren encendido secuencial, liberación de frenos, cambio de marcha y entrada de gas para avanzar, los sistemas moleculares requieren la activación o desactivación secuencial de varias nanomáquinas para realizar tareas específicas que van desde moverse, respirar hasta pensar".
Los investigadores plantearon una pregunta fundamental:¿cómo se han creado, programado y ajustado ensamblajes moleculares dinámicos para sustentar la vida?
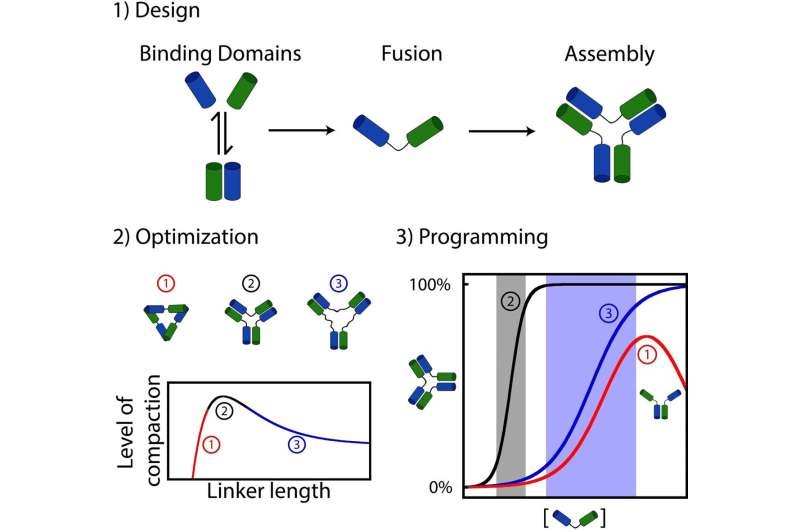
Lo que descubrieron es que muchos ensamblajes biológicos probablemente se formaron uniendo aleatoriamente moléculas que interactúan (por ejemplo, proteínas o ácidos nucleicos como el ADN o el ARN) con conectores que actúan como un "conector" entre cada parte.
"Dado que estos conjuntos biomoleculares desempeñan un papel crucial al permitir que los organismos vivos respondan a su entorno, hemos planteado la hipótesis de que la naturaleza de la conectividad entre los componentes adjuntos también puede contribuir a la evolución de sus respuestas dinámicas", dijo Vallée-Bélisle, titular de la Cátedra de Investigación de Canadá en Bioingeniería y Bionanotecnología.
Para explorar esta cuestión, Dominic Lauzon, estudiante de doctorado en el momento del estudio, decidió sintetizar y unir docenas de moléculas de ADN que interactúan entre sí para explorar el impacto de la conectividad en la dinámica de ensamblaje.
"La química programable y fácil de usar de los ácidos nucleicos como el ADN lo convierte en una molécula conveniente para estudiar cuestiones fundamentales relacionadas con la evolución de las biomoléculas", dijo Lauzon, el primer autor del estudio. "Además, también se cree que los ácidos nucleicos son la molécula que dio origen a la vida en la Tierra."
Lauzon y Vallée-Bélisle descubrieron que una simple variación en la longitud del "enlazador" entre las moléculas que interactúan conduce a variaciones significativas en su dinámica de ensamblaje. Por ejemplo, ciertas asambleas exhibieron una alta sensibilidad a la variación de los estímulos, mientras que otras carecían de dicha sensibilidad o incluso requirieron cambios mucho mayores en los estímulos para promover la asamblea.
Lo que es más sorprendente, algunos enlazadores incluso crearon nuevas funciones reguladoras complejas, como propiedades de autoinhibición, donde la adición de un estímulo promovería tanto su montaje como su desmontaje. Todos estos diferentes comportamientos de respuesta también se observan a menudo en nanomáquinas "vivas" naturales.
Utilizando experimentos y ecuaciones matemáticas, los investigadores también pudieron explicar por qué una variación tan simple de la longitud del conector era tan eficaz para modificar la dinámica del ensamblaje molecular.
"Los enlazadores que crearon los conjuntos más estables fueron los que también crearon los mecanismos de activación más sensibles, mientras que los enlazadores que crearon los conjuntos menos estables crearon los mecanismos de activación menos sensibles, incluso hasta el punto de introducir la autoinhibición", explicó Lauzon. P>
La capacidad de detectar señales moleculares con precisión es crucial para los ensamblajes biológicos, pero también en el desarrollo de la nanotecnología que depende de la detección e integración de información molecular.
Por lo tanto, los investigadores creen que su descubrimiento también puede proporcionar el marco fundamental para crear nanomáquinas o nanosistemas más programables con actividades reguladas de manera óptima, por ejemplo, simplemente uniendo moléculas que interactúan con diferentes enlaces. Estos ensamblajes moleculares ya están encontrando aplicaciones en biodetección o administración de fármacos.
Además de proporcionar una estrategia de diseño simple para crear la próxima generación de nanosistemas autorregulados, los descubrimientos de los científicos también arrojan luz sobre cómo los ensamblajes biomoleculares naturales pueden haber adquirido su dinámica óptima.
"Una estrategia de evolución molecular bien conocida de los organismos vivos es la fusión genética, en la que el ADN que codifica dos dominios proteicos que interactúan se fusiona aleatoriamente", dijo Vallée-Bélisle.
"Nuestros hallazgos también proporcionan la comprensión fundamental necesaria para comprender cómo una simple variación en la longitud del conector entre las proteínas fusionadas puede haber creado de manera eficiente ensamblajes biológicos que muestran una variedad de dinámicas, algunas más adecuadas que otras para proporcionar una ventaja a los organismos vivos". /P>
Más información: Dominic Lauzon et al, Principios de diseño y termodinámica para programar la cooperatividad de ensamblajes moleculares, Edición internacional Angewandte Chemie (2023). DOI:10.1002/anie.202313944
Información de la revista: Edición internacional Angewandte Chemie , Angewandte Chemie
Proporcionado por la Universidad de Montreal