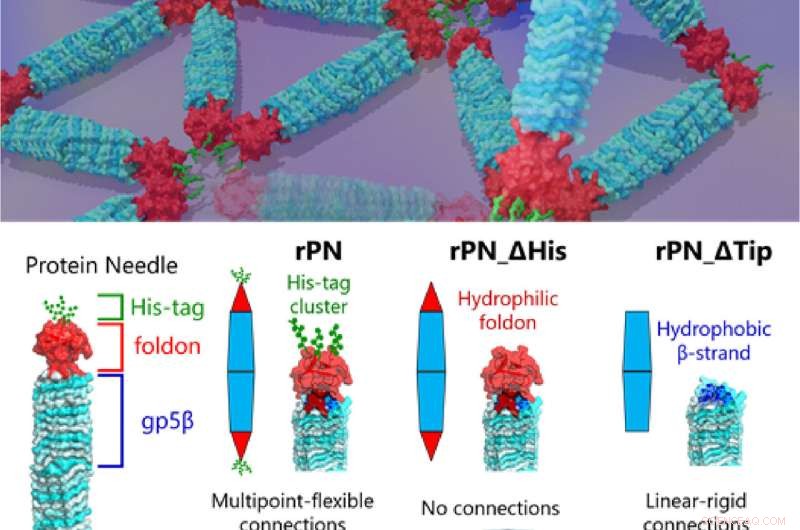
Fig. 1. Los científicos llevan mucho tiempo intentando descifrar las complejas subestructuras de las proteínas. Ahora, los investigadores de Tokyo Tech finalmente han arrojado luz en este frente con la investigación del autoensamblaje de proteínas diseñadas utilizando agujas de proteínas. Crédito:Takafumi Ueno del Instituto de Tecnología de Tokio
El ensamblaje de proteínas es esencial para la formación de estructuras biológicas ordenadas, pero imagine una ingeniería. Esto es exactamente lo que los investigadores de Tokyo Tech han logrado ahora con agujas de proteínas. Al regular las interacciones de punta a punta de estas agujas, permitieron su autoensamblaje en estructuras reticulares, estados monoméricos ordenados y ensamblajes de fibras, allanando el camino para la construcción controlada de más de estas arquitecturas de proteínas.
Las proteínas son los componentes básicos de nuestros cuerpos. Sin embargo, sus estructuras moleculares y macroscópicas son complejas y variadas, con múltiples patrones de plegamiento y subestructuras. Los científicos han estado tratando de descifrar estas estructuras durante algún tiempo y se ha avanzado mucho gracias a la microscopía de fluorescencia (FM), la microscopía de fuerza atómica (AFM) y la AFM de alta velocidad (HS-AFM). Sin embargo, no han podido observar directamente los movimientos dinámicos de las proteínas durante el ensamblaje. Esto se debe principalmente a la intrincada estructura de las proteínas, que son demasiado pequeñas para medirlas con las técnicas existentes.
Un equipo colaborador de investigadores del Instituto de Tecnología de Tokio (Tokyo Tech), la Universidad de Kyushu, la Universidad de Nagoya y los Institutos Nacionales de Ciencias Naturales ahora han desarrollado una aguja de proteína anisotrópica (PN) especializada para ayudar a determinar el ensamblaje de proteínas anisotrópicas similares, dándonos pistas sobre su microestructura y ensamblaje.
El profesor Takafumi Ueno de Tokyo Tech, quien dirigió el estudio, explica la premisa de su trabajo:"Nuestra PN es una proteína en forma de aguja compuesta por el cuerpo rígido (hélice β), la tapa terminal (foldon) y un enlace motivo (etiqueta de hexahistidina, etiqueta de His). Al modificar estos PN eliminando el motivo de la etiqueta de His y la tapa plegable, podemos producir tres tipos diferentes de PN. Esto nos permitió regular y observar diferentes patrones de ensamblaje y cómo cambian , dándonos pistas sobre la mecánica de las diferentes interacciones proteína-proteína que encontramos en la naturaleza". Los resultados de este estudio se publicaron en la revista Small.
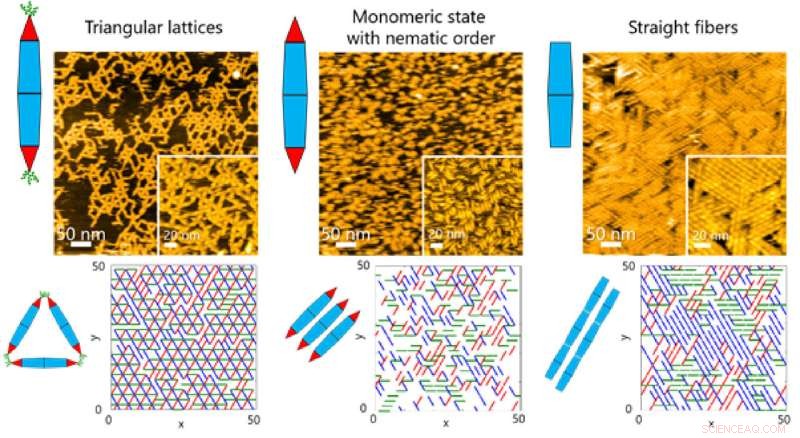
Fig. 2. Las cadenas de proteínas básicas a menudo experimentan un autoensamblaje dinámico para formar estructuras supramoleculares complejas. Los científicos de Tokyo Tech ahora han logrado explorar la dinámica de ensamblaje utilizando agujas de proteínas diseñadas. Crédito:Takafumi Ueno del Instituto de Tecnología de Tokio
En solución, las PN forman espontáneamente una estructura altamente estable con una longitud de aproximadamente 20 nm y un ancho de aproximadamente 3,5 nm, lo suficientemente pequeña como para seguir el movimiento de rotación de moléculas individuales pero mecánicamente fuerte.
En las superficies, el equipo observó diferentes tipos de estructuras ordenadas a medida que las PN se autoensamblaban. Estas estructuras iban desde redes triangulares y estados monoméricos con orden nemático (orientación unidimensional) hasta ensamblajes de fibras (Figura 1).
Esto, a su vez, permitió al equipo investigar los procesos dinámicos involucrados en el ensamblaje de proteínas a través de una combinación de HS-AFM y simulaciones (Figura 2). Los resultados revelaron que la formación de la estructura de celosía triangular fue guiada por los movimientos dinámicos de PN, que contribuyen a formar celosías ordenadas (Figura 3).
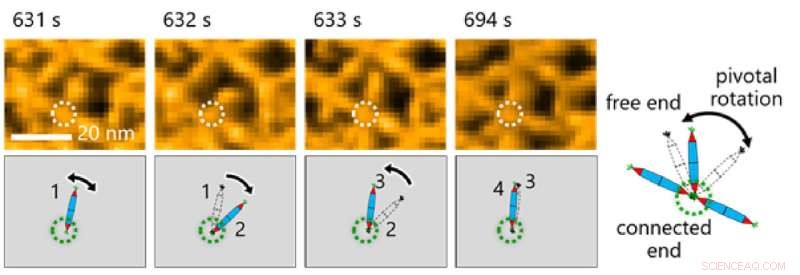
Fig. 3. La observación del movimiento molecular de las PN fue crucial para realizar observaciones en este estudio. Aquí, los investigadores observaron una rotación fundamental en torno a la interacción His-tag entre las agujas de proteínas. Crédito:Takafumi Ueno del Instituto de Tecnología de Tokio
Estos hallazgos han entusiasmado a los investigadores, que están contemplando sus posibles ramificaciones. "Estas moléculas juegan un papel tan crucial en los sistemas biológicos que comprender su estructura avanzaría significativamente en el campo. Por ejemplo, podríamos usar esto para sentar las bases para construir estructuras supramoleculares mediante el diseño de los movimientos colectivos dinámicos de las proteínas. Este concepto puede conducir a la ingeniería de materiales laminares biocompatibles, transportes de fármacos dirigidos e incluso nanorobots basados en proteínas", dice el profesor Ueno. Codifica vías de ensamblaje jerárquico de proteínas