Durante la última década, los modelos generativos de aprendizaje profundo se han aplicado con éxito al diseño de nuevas moléculas de fármacos, rutas de síntesis orgánica y moléculas funcionales adaptadas a dispositivos electrónicos/optoelectrónicos. Esto se debe en gran medida a la disponibilidad de la representación SMILES para moléculas, una representación invertible e invariante muy adecuada para modelos de procesamiento del lenguaje natural como redes neuronales recurrentes, transformadores, etc.
Sin embargo, diseñar sólidos inorgánicos cristalinos con las propiedades deseadas sigue siendo un desafío formidable. Esto se debe principalmente a la falta de una representación cristalina "equivalente a SMILES" para unir materiales periódicos de estado sólido y arquitecturas de aprendizaje profundo de última generación.
Los métodos anteriores para el diseño de cristales inversos se basaban principalmente en cuadrículas de vóxeles 3D o coordenadas espaciales absolutas para representar estructuras. Pero estos enfoques carecen intrínsecamente de invariancia rotacional. También hay intentos de utilizar gráficos cristalinos, que son invariantes pero no invertibles debido a la ausencia de información explícita sobre periodicidad o composición. Para abordar este desafío, propusimos una nueva representación de cristal llamada SLICES. El estudio se publica en la revista Nature Communications. .
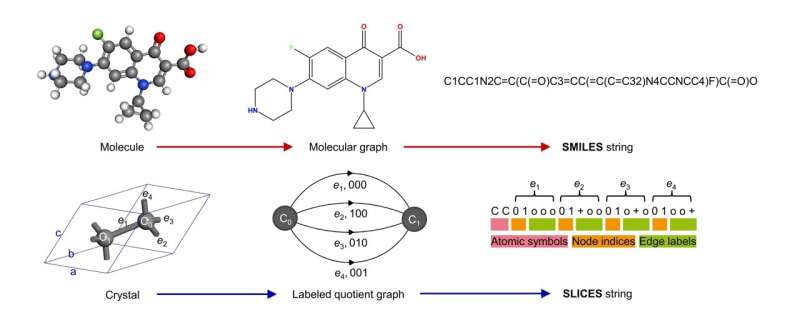
La motivación clave detrás del desarrollo de SLICES es crear una representación cristalina que sea invertible e invariante, análoga a la representación SMILES utilizada ampliamente para el diseño molecular inverso (Figura 1). La invertibilidad significa que la representación se puede convertir sin ambigüedades a la estructura cristalina original. Esto es esencial para que los modelos generativos realicen un diseño inverso, donde los modelos crean nuevas estructuras cristalinas que se decodifican a partir de la representación.
La invariancia indica que la representación permanece sin cambios bajo traslaciones, rotaciones y permutaciones de la estructura cristalina. Satisfacer las invariancias permite que la representación se centre exclusivamente en codificar la información topológica y compositiva esencial de un sistema en lugar de características superficiales que cambian bajo las transformaciones. Esto reduce la redundancia y mejora la eficiencia del aprendizaje.
Al satisfacer la invertibilidad y las invariancias, SLICES permite la exploración eficiente del vasto espacio de compuestos químicos para materiales cristalinos utilizando modelos generativos profundos.
Conceptualmente, SLICES codifica la topología y composición de estructuras cristalinas en cadenas, de forma muy similar a como SMILES convierte gráficos moleculares en notaciones lineales. Más específicamente, SLICES aprovecha el concepto matemático de "gráficos de cocientes etiquetados" para representar estructuras cristalinas periódicas. Los átomos y enlaces dentro de una celda unitaria se asignan a nodos y bordes del gráfico de cociente. Se asignan etiquetas adicionales a los bordes que indican los vectores de desplazamiento periódicos necesarios para conectar átomos equivalentes en celdas unitarias vecinas.
Un ejemplo es la estructura cristalina del diamante (Figura 1), que contiene dos átomos de carbono unidos en la celda unitaria primitiva. La cadena SLICES codifica explícitamente los símbolos atómicos "C" y la etiqueta de borde "001" que denota el enlace periódico que se propaga a lo largo de la dirección [001]. Al analizar la cadena SLICES, se pueden obtener tanto la composición como la conectividad de la estructura del diamante.
En particular, SLICES solo codifica información de topología y composición. Atributos como coordenadas atómicas y parámetros de red no están integrados explícitamente. Esto hace que SLICES sea invariante a traslaciones, rotaciones y permutaciones de índices atómicos por diseño.
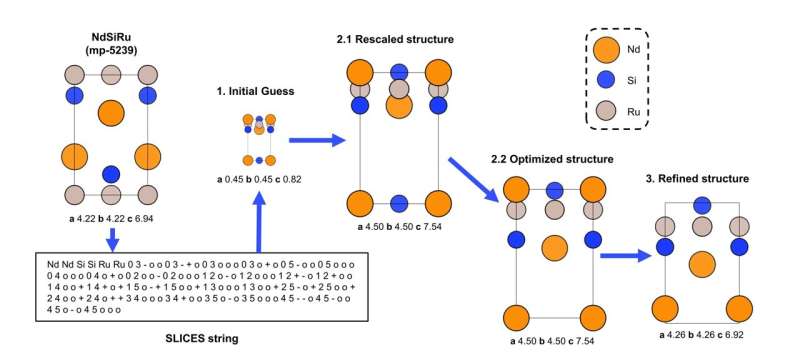
Reconstrucción de estructuras cristalinas a partir de SLICES
Si bien codificar cristales en SLICES es relativamente sencillo, el desafío radica en garantizar la invertibilidad:la capacidad de reconstruir con precisión estructuras cristalinas a partir de cadenas SLICES. Para lograr la invertibilidad, desarrollamos un proceso de reconstrucción (Figura 2) para SLICES que contiene tres pasos clave:
El rendimiento de la reconstrucción se comparó con una base de datos que contiene más de 40.000 materiales conocidos experimentalmente con hasta 20 átomos por celda unitaria. El proceso de reconstrucción de SLICES pudo reconstruir el 94,95% de las estructuras originales, superando sustancialmente a los métodos anteriores. Esta invertibilidad de SLICES permite la generación de nuevas estructuras a partir de representaciones aprendidas, lo cual es clave para el diseño de materiales inversos.

Aplicación en diseño inverso de materiales funcionales
Como demostración, aplicamos SLICES en el diseño inverso de semiconductores directos de banda estrecha para dispositivos optoelectrónicos utilizando redes neuronales recurrentes (RNN). El flujo de trabajo consta de (Figura 3):
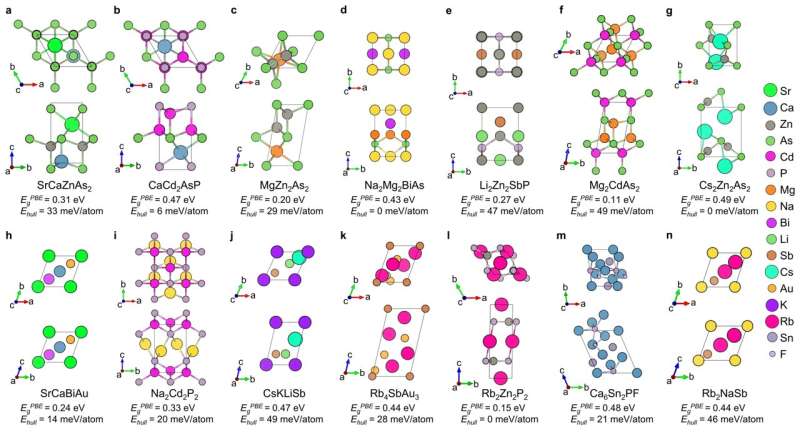
A través de este flujo de trabajo que combina SLICES, RNN y cálculos de alto rendimiento, se descubrieron 14 semiconductores novedosos con bandas prohibidas directas en el rango óptimo (Figura 4). Esto muestra la promesa de SLICES como facilitador del descubrimiento acelerado de materiales funcionales utilizando IA generativa.
Generación dirigida de nuevos materiales con energías de formación específicas
Además, empleamos una arquitectura de red neuronal recurrente condicional (cRNN), como se ilustra en la Figura 5, para generar cadenas SLICES correspondientes a cristales con una energía de formación deseada especificada por el usuario. La distribución de las energías de formación de las estructuras generadas se acerca al valor objetivo especificado en relación con la distribución del conjunto de datos. El cRNN basado en SLICEs supera significativamente a los modelos de última generación anteriores. Este enfoque marca un avance significativo en la capacidad de diseñar y descubrir nuevos materiales de manera controlada y precisa.

Como la primera representación cristalina invertible e invariante basada en cuerdas, SLICES abre muchas oportunidades interesantes en el diseño inverso de sólidos cristalinos, tal como lo ha hecho SMILES con las moléculas en la última década. Tan solo en los últimos años, hemos sido testigos de enormes avances en modelos generativos que van desde imágenes, vídeos, voz hasta proteínas y moléculas. Imaginamos que los materiales sólidos serán la próxima frontera, gracias a esta nueva capacidad de exploración integrada con química y con datos eficientes, potenciada por representaciones como SLICES.
Esta historia es parte de Science X Dialog, donde los investigadores pueden informar los hallazgos de sus artículos de investigación publicados. Visite esta página para obtener información sobre ScienceX Dialog y cómo participar.
Más información: Hang Xiao et al, Una representación de cristal invariante e invertible para el diseño inverso de materiales de estado sólido utilizando el aprendizaje profundo generativo, Nature Communications (2023). DOI:10.1038/s41467-023-42870-7
Información de la revista: Comunicaciones sobre la naturaleza
Hang Xiao está afiliado a la Escuela de Estudios Interdisciplinarios de la Universidad Lingnan; Obtuvo su doctorado en la Universidad de Columbia. Yan Chen está afiliado al Laboratorio de Mecánica Multiescala y Ciencias Médicas, SV LAB, Escuela de Aeroespacial, Universidad Xi'an Jiaotong, donde también obtuvo su doctorado.