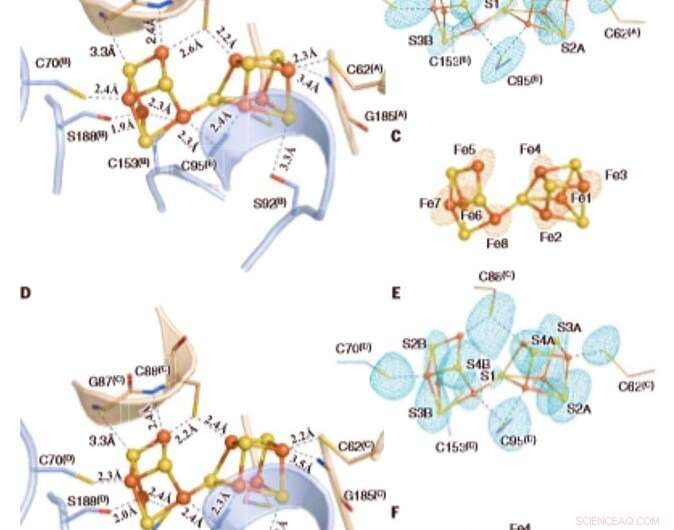
Los grupos de P oxidados en Av1 *. Estructuras de grupos P en (A a C) la interfaz de las cadenas A y B [grupo P (A / B)] y (D a F) la interfaz de las cadenas C y D [grupo P (C / D) ] de Av1 *. Las cadenas A y C son las subunidades a, y las cadenas B y D son las subunidades b de los dos dímeros ab de Av1 *. [(A) y (D)] Los grupos P se muestran en una presentación de bola y palo, y los residuos clave que interactúan con los grupos P se indican como barras. Las cadenas A y C se muestran como cintas de trigo, y las cadenas B y D se muestran como cintas azul claro. [(B) y (C)] P-cluster (A / B) y [(E) y (F)] P-cluster (C / D) superpuesto con [(B) y (E)] los mapas de densidad anómalos calculado a 7100 eV a una resolución de 2,18 Å y contorneado a 4,0 s, mostrando la posición de los átomos de azufre (malla azul menta); y con [(C) y (F)] los mapas de densidad anómala calculados a 7141 eV a una resolución de 2,1 Å y contorneados a 15,0 s, mostrando la posición de los átomos de hierro (malla roja). Los átomos se colorean de la siguiente manera:Fe, naranja; S, amarillo; Oh rojo; NORTE, azul. Las abreviaturas de una sola letra para los residuos de aminoácidos son las siguientes:C, Cys; GRAMO, Gly; H Su; R, Arg; S, Ser. Crédito:Ciencia, doi:10.1126 / science.aaz6748
La enzima nitrogenasa es un catalizador biológico que puede reducir el dinitrógeno (N2) a amoníaco en presencia de un conjunto de metalocofactores complejos. Sin embargo, los detalles mecanicistas de la reacción siguen siendo escasos. En un nuevo informe sobre Ciencias , Wonchull Kang y un equipo de investigación en química, biología molecular y bioquímica en la Universidad de California-Irvine, NOSOTROS., informó una estructura cristalina de 1,83 angstrom para la proteína nitrogenasa molibdeno-hierro (MoFe), que capturaron en condiciones fisiológicas de recambio de dinitrógeno. Los resultados del estudio pueden evaluar los posibles mecanismos de N 2 reducción y el papel de los sitios de cinturón de azufre durante el proceso.
La nitrogenasa es un catalizador de un paso crítico en el ciclo global del nitrógeno, durante la reducción ambiental de dinitrógeno atmosférico (N 2 ) al amoníaco biodisponible (NH 3 ). La enzima nitrogenasa de molibdeno contiene dos componentes proteicos:uno que contiene la proteína de hierro (Fe) en un azufre de hierro (Fe 4 S 4 ) grupo con un sitio de unión de trifosfato de adenosina (ATP) dentro de cada subunidad. El otro, proteína de molibdeno hierro (MoFe) que contiene un α 2 β 2 heterotetramer con dos metaloclusters complejos. Durante la catálisis de molibdeno-nitrogenasa (Mo-nitrogenasa), la asociación y disociación repetidas entre los dos componentes proteicos permitieron la transferencia de electrones dependiente de ATP desde el Fe 4 S 4 se agrupan en la proteína MoFe para la reducción del sustrato. La capacidad de la nitrogenasa para transportar muchos electrones a su cofactor hizo que la enzima fuera muy versátil durante la reducción del sustrato.
Comprender el mecanismo de acción de la enzima nitrogenasa
Se han realizado muchos esfuerzos para comprender los mecanismos de la nitrogenasa desde su descubrimiento, donde algunos se habían centrado en las interacciones entre el sustrato y el inhibidor de la enzima. De estos esfuerzos, Kang y col. determinó una estrategia digna de consideración al limitar el exceso de suministro de electrones que inadvertidamente impulsó la N 2 proceso de reducción hacia adelante. Esto revirtió el sustrato o estado de unión intermedia de la enzima a un estado de reposo o redujo la enzima a un estado mixto indiscernible. El proceso fue relevante ya que las proteínas nitrogenasas se aíslan de forma rutinaria en presencia de un exceso de ditionito como reductor suministrado externamente. y la eliminación de esta fuente de electrones artificiales en ausencia de oxígeno podría ayudar a los científicos a capturar dinitrógeno (N 2 ) o sus intermedios para su análisis.
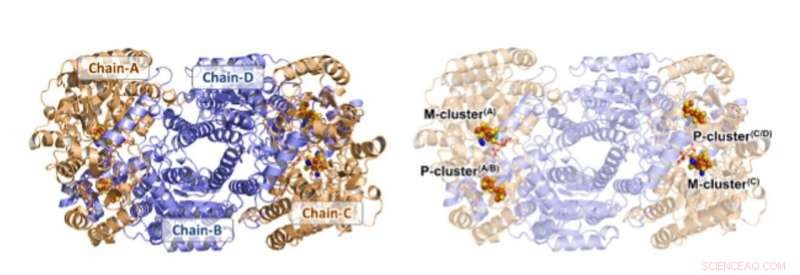
Estructura de Av1 *. Presentaciones de cinta sólida (izquierda) y transparente (derecha) del heterotetrámero de Av1 *, con las subunidades a (Cadena-A y Cadena-C) y las subunidades b (Cadena-B y Cadena-D) coloreadas en trigo y azul, respectivamente. Los grupos M y P se ilustran como modelos de llenado de espacio. Código de color de los átomos:Fe, naranja; S, amarillo; Oh rojo; NORTE, azul; Mes, cian C, gris. Los grupos M en Chain-A y Chain-C se denominan grupo M (A) y grupo M (C), respectivamente. Los grupos P en las interfaces Chain-A / Chain-B y Chain-C / Chain-D se denominan grupo P (A / B) y grupo P (C / D), respectivamente. Se utilizó PYMOL para preparar esta figura. Crédito:Ciencia, doi:10.1126 / science.aaz6748
Como prueba de concepto, Kang y col. preparó el extracto crudo de una cepa bacteriana anaeróbica Azotobacter vinelandii con o sin la adición de ditionito después de la rotura celular. La cepa de A. vinelandii expresó activamente una Mo-nitrogenasa que contenía una proteína MoFe marcada con histidina en ambos casos. Cuando analizaron la actividad de estas muestras, las muestras de extracto crudo sin ditionito estuvieron casi inactivas durante la reducción del sustrato, debido al agotamiento de electrones en los extractos crudos durante la rotura celular. Kang y col. Por lo tanto, podría restaurar completamente la actividad de las muestras agregando ditionito y ATP (es decir, suministrando electrones).
Grupos de nitrogenasa:dos metaloclusters únicos:el grupo P y el grupo M.
Según las condiciones descritas, cuando un cultivo que expresa nitrogenasa que realiza activamente N 2 la fijación se somete a lisis celular sin suministro adicional de electrones, la nitrogenasa permaneció funcional. Aunque potencialmente detenido en un estado "latente" o de enlace intermedio debido a la extracción del flujo de electrones en un metalocluster de hierro-azufre conocido como el grupo M, ubicado dentro de la enzima nitrogenasa. Cuando Kang et al. purificó el extracto crudo libre de ditionito, la proteína MoFe marcada con histidina (designada como AV1 * en el estudio) estuvo activa durante N 2 reducción y también completamente funcional. Cuando el equipo cristalizó AV1 *, observaron cristales marrones que se difractaban a una resolución de 1,83 angstrom (Å). Confirmaron el reordenamiento estructural de los dos grupos P de AV1 * utilizando datos de densidad anómalos y utilizaron resonancia paramagnética de electrones para observar la asignación estructural. Los resultados les proporcionaron respuestas largamente buscadas a la relevancia fisiológica de este estado experimental y señalaron un flujo limitado de electrones entre los dos metaloclusters únicos (grupos P y M) del compuesto en ausencia de ditionito.
Un mecanismo plausible de reducción de N2 por Mo-nitrogenasa. Crédito:Markus Ribbe y Yilin Hu, Ciencias , doi:10.1126 / science.aaz6748 
Un mecanismo de acción plausible que coincidía con las observaciones experimentales incluía la reducción gradual de dinitrógeno (N 2 ) en los tres sitios de azufre del cinturón en el catalizador de nitrogenasa basado en la rotación del grupo M. El mecanismo propuesto comienza con una fuerte unión de N 2 en un sitio específico, seguido de la rotación del límite N 2 a otro sitio posterior (sitios designados como S3A a S2B a S5A en el compuesto). Durante el proceso, la reducción / protonación de N 2 al nivel de diazeno se produjo a través de enlaces de hidrógeno, seguido de una mayor reducción / protonación para su conversión en amoníaco, antes de su liberación de la estructura. La rotación posterior del clúster trajo una nueva N 2 molécula al siguiente sitio para iniciar la siguiente ronda de N paso a paso 2 reducción a través de la rotación continua del racimo en un delicado mecanismo durante la catálisis. Tal ciclo entre diferentes sitios de reacción fue ligeramente análogo al mecanismo de la enzima ATP sintasa. El metalocluster giratorio permitió así efectivamente la reducción de múltiples electrones de N 2 a través de un enfoque de divide y vencerás.
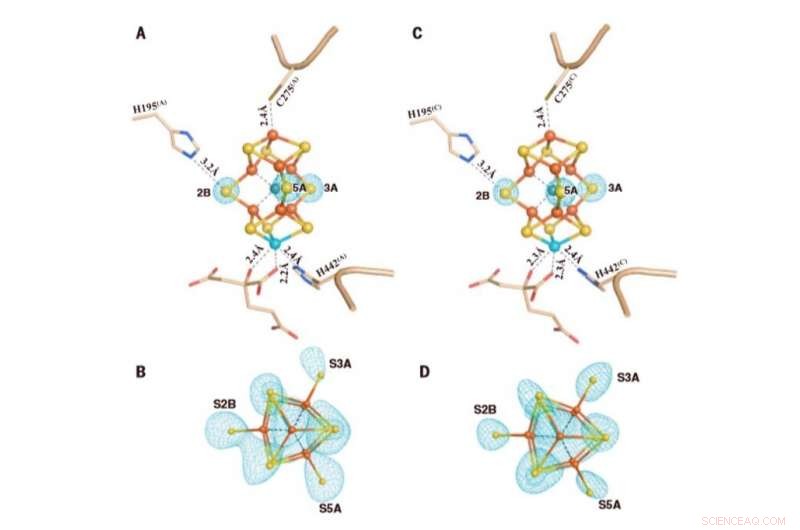
Los grupos M en Av1 * (TOD). Las estructuras de los grupos M en la cadena (A y B) A [grupo M (A)] y la cadena C (C y D) [grupo M (C)] se refinaron a una resolución de 1,73 Å. Vista lateral de (A) grupo M (A) y (C) grupo M (C) con residuos clave que interactúan con los grupos indicados como palos. M-cluster(A) and M-cluster(C) are superimposed with the Fo-Fc omit maps of the belt sulfurs contoured at 13s (mint-blue mesh). View along the Fe1-C-Mo direction of (B) M-cluster(A) and (D) M-cluster(C) superimposed with the anomalous density maps calculated at 7100 eV at a resolution of 2.17 Å and contoured at 4.0s, showing the presence of the anomalous sulfur density (mint-blue mesh) at all belt sulfur positions (S2B, S3A, and S5A) in (B) M-cluster(A) and (D) M-cluster(C). Credit:Science, doi:10.1126/science.aaz6748
To understand the sulfur-displaced conformation of AV1* under limited electron flux, the team formed AV1* turnover with dithionite (designated as AV1*TOD), to yield brown crystals that diffracted to a resolution of 1.73 Å. The observations were consistent with the mechanism of bound dinitrogen species on the compound and illustrated the physiological relevance of the conformation during catalysis. The capacity to displace three different sites by a dinitrogen species was consistent with previous investigations on catalysis-dependent selenium. Kang et al. proposed many mechanisms to explain the observations, however they seek further experimental support to verify them. The team highlighted the possibility for all belt-sulfur sites to be involved in the process of catalysis due to the presence of asymmetric belt-sulfur displacements in the compound. The results aim to provoke a paradigm shift in the mechanistic thinking of nitrogenase activity, ultimately to understand the intricate mechanism of the enzyme.
© 2020 Science X Network