Los científicos del proyecto de software internacional MZmine, dirigidos por el Dr. Robin Schmid y el Dr. Tomáš Pluskal del Instituto de Química Orgánica y Bioquímica de la Academia Checa de Ciencias, han ideado un nuevo software que acelera y simplifica considerablemente la identificación de sustancias químicas en los tejidos.
Permite a los investigadores reconocer con confianza sustancias químicas y visualizar su distribución en los órganos. En comparación con los flujos de trabajo establecidos, el nuevo proceso requiere menos trabajo en el laboratorio para obtener información similar sobre la química que ocurre, por ejemplo, en tumores y lesiones inflamatorias. El artículo que presenta este software a la comunidad científica mundial acaba de publicarse en la revista Nature Communications. .
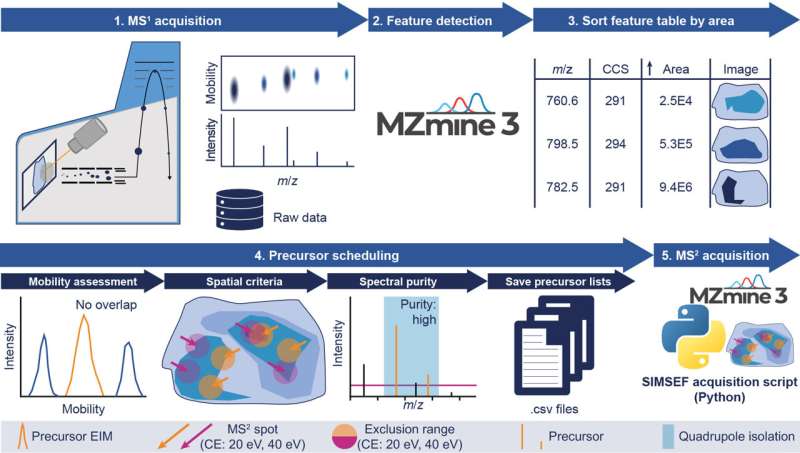
En el IOCB Praga, el Dr. Robin Schmid participa principalmente en el desarrollo del software SIMSEF (fragmentación exhaustiva programada por movilidad iónica espacial) junto con sus colegas del grupo del Prof. Dr. Karst en la Universidad de Münster, Alemania, donde anteriormente estuvo basado.
El trabajo se realiza en colaboración con los desarrolladores del espectrómetro de masas de última generación timsTOF fleX de Bruker Daltonics, que permite la elucidación de alto nivel de la composición de las moléculas midiendo la movilidad de los iones.
"Hasta ahora, los científicos podían decir con un espectrómetro de masas que ésta era la fórmula de la molécula que estaban observando, pero luego, cuando miraron la base de datos e intentaron identificar esta sustancia, fue muy difícil. La razón es que las sustancias biológicas Las muestras pueden contener numerosos lípidos distintos y combinaciones de moléculas que a menudo son biológicamente muy diferentes."
"Ahora, gracias al nuevo algoritmo, es posible mirar dentro de la molécula, saber de qué está compuesta e incluso comparar imágenes entre sí", explica Robin Schmid. Esto puede proporcionar información importante, por ejemplo, que una parte específica del cerebro contiene un tipo de lípido diferente al de otra parte de este órgano esencial y que este lípido en particular está ausente en otro tejido.
El algoritmo SIMSEF se desarrolló en colaboración con expertos médicos de universidades alemanas y suizas. Lo más importante para los médicos es que también se acelera significativamente la detección de biomarcadores clínicos con fines de diagnóstico. Saber muy rápidamente, por ejemplo, si se trata de un lípido o de un metabolito que sólo se encuentra en el tejido canceroso, desempeña un papel fundamental a la hora de decidir un tratamiento adicional. No hace falta decir que esto tiene grandes implicaciones para el resultado.
El nuevo algoritmo forma parte del paquete de software de código abierto MZmine, que ayuda a expertos de todo el mundo a analizar datos de espectrometría de masas desde 2005. Steffen Heuckeroth, del equipo de MZmine y del Instituto de Química Inorgánica y Analítica de la Universidad de Münster, ha sido el principal desarrollador del algoritmo SIMSEF y es el primer autor del artículo publicado ahora en Nature Communications. .
Y añade:"El artículo en cuestión no contiene ningún descubrimiento específico en el campo de la biología. Sin embargo, gracias a nuestro nuevo método, muchos científicos pueden mejorar y acelerar su trabajo, lo que abre espacio para nuevos descubrimientos".
"Siempre pienso que es genial cuando permitimos a otras personas mejorar las condiciones de trabajo para su investigación y también comunicarlo a otros científicos. Gracias a los comentarios de la comunidad científica que utiliza MZmine, el programa en sí impulsa directamente su investigación", añade Robin Schmid del IOCB Praga.
El director del IOCB Praga, profesor Jan Konvalinka, señala:"Hoy en día todo el mundo habla de aprendizaje automático e inteligencia artificial. Nuestros colegas del equipo del Dr. Pluskal los utilizan para resolver problemas muy específicos tanto en la investigación como en la práctica clínica".
La tercera generación de MZmine, publicada en la primavera de 2023 en la revista Nature Biotechnology , puede procesar miles de muestras por hora. Los algoritmos recientemente desarrollados por este equipo internacional mejoran aún más las capacidades existentes para vincular diferentes tipos de datos, incluida la combinación de datos de métodos analíticos y de imágenes, lo que hasta ahora no ha sido posible con ningún software académico o comercial.
Más información: Steffen Heuckeroth et al, Bioimagen MALDI-TIMS-MS2 dependiente de conjuntos de datos en tejidos, Nature Communications (2023). DOI:10.1038/s41467-023-43298-9
Información de la revista: Comunicaciones sobre la naturaleza , Biotecnología de la Naturaleza
Proporcionado por el Instituto de Química Orgánica y Bioquímica de la Academia Checa de Ciencias (IOCB Praga)