Un equipo de la Universidad de Massachusetts Amherst ha logrado un importante avance hacia el modelado y la comprensión de cómo las proteínas intrínsecamente desordenadas (IDP) experimentan una separación de fases espontánea, un importante mecanismo de organización subcelular que subyace a numerosas funciones biológicas y enfermedades humanas.
Los desplazados internos desempeñan un papel crucial en el cáncer, los trastornos neurodegenerativos y las enfermedades infecciosas. Constituyen aproximadamente un tercio de las proteínas que produce el cuerpo humano, y dos tercios de las proteínas asociadas al cáncer contienen segmentos o dominios grandes y desordenados. Identificar las características ocultas cruciales para el funcionamiento y el autoensamblaje de los desplazados internos ayudará a los investigadores a comprender qué falla en estas características cuando ocurren enfermedades.
En un artículo publicado en el Journal of the American Chemical Society , el autor principal Jianhan Chen, profesor de química, describe una forma novedosa de simular separaciones de fases mediadas por desplazados internos, un proceso importante que ha sido difícil de estudiar y describir.
"La separación de fases es un fenómeno muy conocido en la física de polímeros, pero lo que la gente no sabía hasta hace unos 15 años es que también es un fenómeno muy común en biología", explica Chen. "Se puede observar la separación de fases con un microscopio, pero entender este fenómeno a nivel molecular es muy difícil.
"En los últimos cinco o diez años, la gente ha comenzado a descubrir que muchas de estas proteínas desordenadas pueden impulsar la separación de fases, incluidas numerosas proteínas importantes implicadas en el cáncer y los trastornos neurodegenerativos".
El nuevo artículo, basado en una investigación realizada en el laboratorio de biofísica computacional y biomateriales de Chen, constituye un capítulo del trabajo doctoral del autor principal Yumeng Zhang. disertación. Zhang comenzará a trabajar como investigador postdoctoral en el Instituto Tecnológico de Massachusetts (MIT) en febrero. Otro colaborador clave es Shanlong Li, investigador asociado postdoctoral en el laboratorio de Chen.
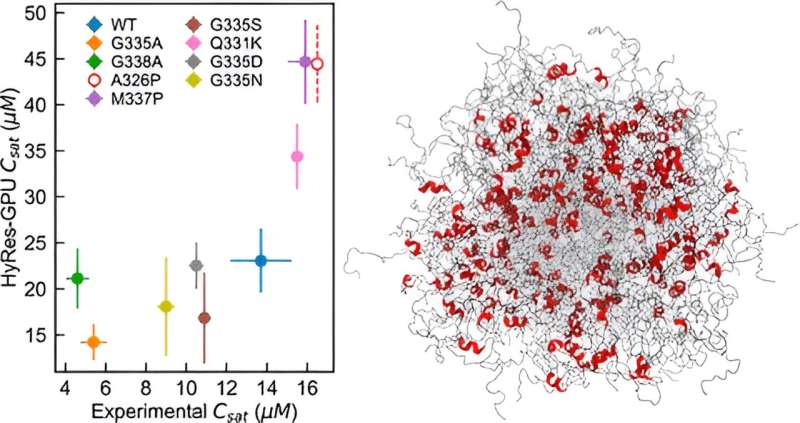
El laboratorio de Chen desarrolló un campo de fuerza preciso de resolución híbrida (HyRes) acelerado por GPU para simular separaciones de fases mediadas por desplazados internos. Este modelo es único en su capacidad para describir con precisión las interacciones del esqueleto peptídico y las estructuras secundarias transitorias, al mismo tiempo que es lo suficientemente eficiente desde el punto de vista computacional para modelar la separación de fases líquido-líquido. Este nuevo modelo llena un vacío crítico en la capacidad existente en la simulación por computadora de la separación de fases IDP.
Chen y su equipo crearon simulaciones HyRes para demostrar por primera vez qué gobierna la estabilidad del condensado de dos importantes desplazados internos.
"En realidad, no anticipé que pudiera hacer un trabajo tan bueno al describir la separación de fases porque es un fenómeno realmente difícil de simular", dice Chen. "Demostramos que este modelo es lo suficientemente preciso como para empezar a observar los impactos de incluso una sola mutación o estructuras residuales en la separación de fases".
La HyRes-GPU de los investigadores proporciona una innovadora herramienta de simulación para estudiar los mecanismos moleculares de separación de fases. El objetivo final es desarrollar estrategias terapéuticas en el tratamiento de enfermedades asociadas a proteínas desordenadas.
"Ésta es realmente la importancia de este trabajo", afirma Chen. "Se cree que importantes procesos biológicos ocurren a través de la separación de fases. Entonces, si podemos comprender mejor qué controla este proceso, ese conocimiento será realmente poderoso, si no esencial, para que podamos pensar en controlar la separación de fases para diversos fines científicos y de ingeniería. Esto nos ayudará a comprender el tipo de intervención que será necesaria para lograr efectos terapéuticos."
Chen dice que el siguiente paso es aplicar lo que su equipo ha aprendido a simulaciones a mayor escala de mezclas biomoleculares más complejas.
"Shanlong está trabajando ahora en la construcción de un modelo similar para los ácidos nucleicos porque la separación de fases a menudo involucra tanto proteínas como ácidos nucleicos desordenados", dice. "Queremos poder describir ambos componentes clave y eso nos permitiría analizar muchos más sistemas".
Más información: Yumeng Zhang et al, Hacia una simulación precisa del acoplamiento entre la estructura secundaria de proteínas y la separación de fases, Revista de la Sociedad Química Estadounidense (2023). DOI:10.1021/jacs.3c09195
Información de la revista: Revista de la Sociedad Química Estadounidense
Proporcionado por la Universidad de Massachusetts Amherst