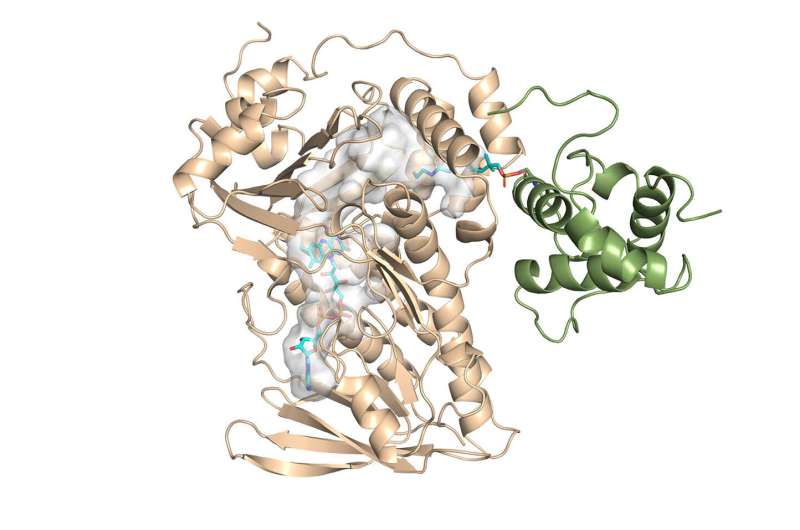
Los químicos han determinado por primera vez la estructura cristalina de un componente clave de la enzima monensina y han descubierto el mecanismo de su actividad reactiva.
"El hallazgo principal fue la primera estructura cristalina para esta familia de enzimas", dijo Chu-Young Kim, profesor de Bioquímica en la Universidad de Illinois Urbana-Champaign (UIUC), quien dirigió la parte experimental del estudio. Él y sus colegas resolvieron la estructura cristalina de MonCI, una enzima clave en las bacterias del suelo que sintetiza naturalmente la monensina.
Lela Vukovic, profesora asociada de la Universidad de Texas, en El Paso (UTEP), realizó los estudios computacionales sobre la investigación de la monensina, publicados en Nature Communications. .
La iniciativa de Infraestructura Cibernética de Investigación de la Universidad de Texas (UTRC) otorgó a Vukovic asignaciones de supercomputadoras en el sistema Lonestar6 en el Centro de Computación Avanzada de Texas (TACC) para enfrentar estos desafíos. UTRC proporciona capacidades informáticas avanzadas a investigadores de las 14 instituciones del Sistema UT.
Lonestar6 de TACC ayudó a revelar la secuencia de reacción que produce monensina. Esta investigación abre la puerta al diseño futuro de antibióticos más seguros y eficaces.
"Descubrimos que MonCI se utiliza para llevar a cabo tres reacciones de epoxidación cruciales", dijo Kim. "Esto es muy inusual y tiene implicaciones sobre cómo podemos diseñar la bacteria para que produzca nuevos antibióticos".
Kim, quien recientemente dejó UTEP para unirse a UIUC, consultó el laboratorio de Vukovic con los resultados de la estructura y lleno de nuevas preguntas. Lo que encontró fue una interesante reacción secuencial dentro de la enzima. Sin embargo, todavía era experimentalmente imposible obtener la estructura cristalina de la enzima con el sustrato en su interior en modo activo.
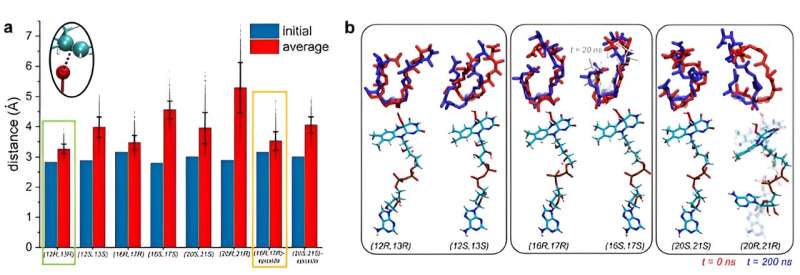
Fue entonces cuando Kim y Vukovic decidieron modelar la enzima y el subestado en simulaciones cuando el sustrato es estable. Si un sustrato es estable en una determinada posición, entonces puede ocurrir una reacción en esa posición.
Vukovic y sus estudiantes Tara Nitka y Anju Yadav desarrollaron modelos completos del sistema de aproximadamente 78.000 átomos basados en las estructuras cristalinas determinadas por los experimentalistas del estudio.
"Los desafíos computacionales surgieron al examinar múltiples sistemas para determinar la posición en la que la premonensina A y sus versiones epoxidadas son más estables y tienen más probabilidades de sufrir una primera, segunda y tercera reacción de epoxidación", dijo Vukovic.
"Las supercomputadoras han sido muy útiles para caracterizar estas moléculas biológicas que producen antibióticos de forma natural", añadió Vukovic. "No podríamos realizar estudios computacionales sin ellos. Los estudios computacionales nos ayudan a descubrir y comprender estas complejas reacciones secuenciales importantes para la sociedad".
Vukovic realizó su investigación postdoctoral en UIUC con el fallecido Klaus Schulten, cuyo legado sigue vivo en el software NAMD que su grupo desarrolló y se utilizó en este estudio y en muchos otros.
"UIUC está trabajando mucho para optimizar NAMD para que se ejecute en las supercomputadoras del país, como Lonertar6 y Stampede2 en TACC", dijo. "NAMD nos permitió profundizar en esta enzima y ver qué reacciones ocurren primero, segundo y tercero para generar monensina".
"La biosíntesis de monensina requiere al menos 14 enzimas diferentes, una de las cuales es MonCI", añadió Kim. "También debemos investigar todas las demás enzimas. En el futuro, esperamos generar versiones mejoradas de monensina para cuidar mejor al ganado vacuno y a las aves de corral. Además, la monensina es tóxica para los caballos y los perros, por lo que estos animales de granja a veces son envenenados accidentalmente. y muerto. Por lo tanto, se necesita una monensina no tóxica."
Los autores son Qian Wang, Tara A. Nitka, Anju Yadav y Lela Vukovic de la Universidad de Texas en El Paso; Ning Liu, Hongli Xiao, Hui Yang y Xi Chen de la Universidad del Noroeste, China; Irimpan I. Mathews del Laboratorio Nacional de Aceleradores SLAC; y Chu-Young Kim de la Universidad de Illinois Urbana-Champaign.
Más información: Qian Wang et al, Formación de triepóxido por una monooxigenasa dependiente de flavina en la biosíntesis de monensina, Nature Communications (2023). DOI:10.1038/s41467-023-41889-0
Proporcionado por la Universidad de Texas en Austin