Un nuevo estudio de la Universidad de Tel Aviv ha descubierto que una práctica conocida en tecnología de la información también se puede aplicar a la química. Los investigadores descubrieron que para mejorar el muestreo en simulaciones químicas, todo lo que hay que hacer es detenerse y reiniciar.
La investigación fue dirigida por Ph.D. estudiante Ofir Blumer, en colaboración con el profesor Shlomi Reuveni y el Dr. Barak Hirshberg de la Escuela de Química Sackler de la Universidad de Tel Aviv. El estudio fue publicado en Nature Communications. .
Los investigadores explican que las simulaciones de dinámica molecular son como un microscopio virtual. Siguen el movimiento de todos los átomos en sistemas químicos, físicos y biológicos, como proteínas, líquidos y cristales. Proporcionan información sobre diversos procesos y tienen diferentes aplicaciones tecnológicas, incluido el diseño de fármacos.
Sin embargo, estas simulaciones se limitan a procesos más lentos que una millonésima de segundo y, por lo tanto, no pueden describir procesos más lentos como el plegamiento de proteínas y la nucleación de cristales. Esta limitación, conocida como problema de escala de tiempo, es un gran desafío en el campo.
Doctor. El estudiante Ofir Blumer dice:"En nuestro nuevo estudio, demostramos que el problema de la escala de tiempo se puede superar mediante el reinicio estocástico de las simulaciones. A primera vista parece contradictorio:¿cómo pueden las simulaciones terminar más rápido cuando se reinician? Sin embargo, resulta que la reacción Los tiempos varían considerablemente entre simulaciones. En algunas simulaciones, las reacciones ocurren rápidamente, pero otras simulaciones se pierden en estados intermedios durante períodos prolongados. El reinicio evita que las simulaciones se queden atascadas en dichos estados intermedios y acorta el tiempo promedio de simulación.
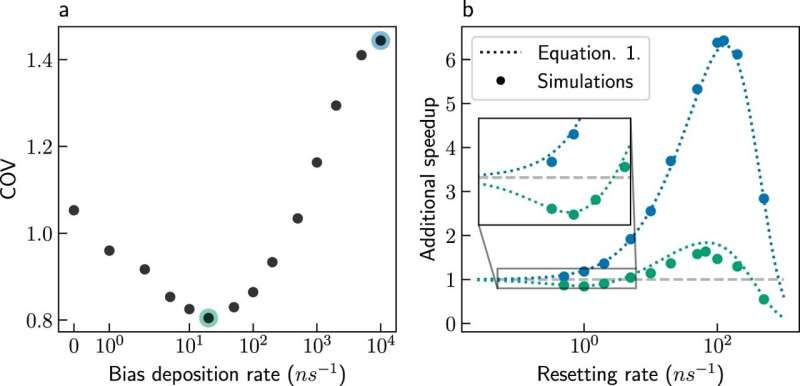
Los investigadores también combinaron el reinicio estocástico con la metadinámica, un método popular para acelerar las simulaciones de procesos químicos lentos. La combinación permite una mayor aceleración que cualquiera de los métodos por separado. Además, la metadinámica se basa en el conocimiento previo:se deben conocer las coordenadas de reacción para acelerar la simulación.
La combinación de Metadinámica con reinicio reduce significativamente la dependencia del conocimiento previo, ahorrando tiempo a los practicantes del método. Finalmente, los investigadores demostraron que la combinación proporciona predicciones más precisas de la velocidad de los procesos lentos. El método combinado se utilizó para mejorar con éxito las simulaciones del plegamiento de una proteína en agua y se espera que se aplique a más sistemas en el futuro.
Más información: Ofir Blumer et al, Combinando el restablecimiento estocástico con la metadinámica para acelerar las simulaciones de dinámica molecular, Nature Communications (2024). DOI:10.1038/s41467-023-44528-w
Información de la revista: Comunicaciones sobre la naturaleza
Proporcionado por la Universidad de Tel-Aviv