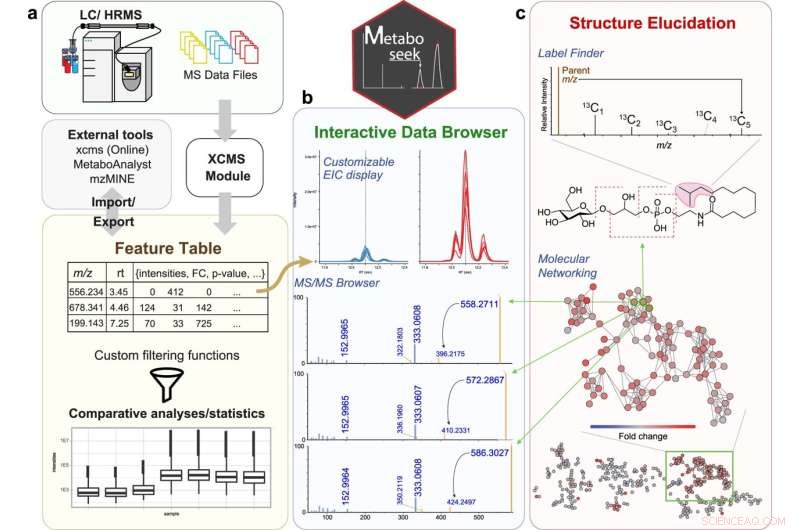
Metabolómica comparativa con Metaboseek. a Metaboseek incluye un módulo XCMS integrado para la detección y agrupación de características (con anotación CAMERA) y acepta tablas de características generadas por otro software. b Las características pueden anotarse y priorizarse utilizando amplias opciones de filtrado y herramientas de estadísticas integradas. Los datos sin procesar para cada característica molecular se pueden examinar rápidamente, incluidos los espectros EIC, MS1 y MS/MS asociados. c El navegador de datos interactúa con un conjunto de herramientas de elucidación de estructuras, por ejemplo, fórmula molecular basada en SIRIUS y predicción de estructuras, el buscador de etiquetas para identificar compuestos marcados con isótopos y el buscador de patrones de MS/MS para identificar características de MS con patrones de fragmentación característicos. Crédito:Comunicaciones de la naturaleza (2022). DOI:10.1038/s41467-022-28391-9
Como investigador asociado postdoctoral en el laboratorio del miembro de la facultad de BTI, Frank Schroeder, Max Helf vio que sus compañeros de laboratorio luchaban continuamente cuando analizaban datos. Entonces, decidió hacer algo al respecto y desarrolló una aplicación gratuita de código abierto llamada Metaboseek, que ahora es esencial para el trabajo del laboratorio.
El laboratorio de Schroeder estudia el gusano redondo Caenorhabditis elegans, uno de los sistemas modelo más exitosos para la biología humana, para descubrir nuevos metabolitos que rigen las vías de señalización conservadas evolutivamente y podrían ser útiles como pistas para el desarrollo de nuevos productos farmacéuticos o agroquímicos. Los investigadores logran esta tarea comparando los metabolitos entre dos poblaciones diferentes de gusanos, un proceso llamado metabolómica comparativa.
Dado que las muestras tienen habitualmente más de 100 000 compuestos, los enfoques computacionales son esenciales para realizar el análisis.
El equipo dependía de paquetes de software que no ofrecían el nivel necesario de flexibilidad para personalizar fácilmente los parámetros de análisis. Esa limitación, y la falta de una interfaz gráfica de usuario adecuada, hizo que los colegas de Helf se enfrentaran a la engorrosa tarea de inspeccionar visualmente montones de datos, por ejemplo, para detectar posibles falsos positivos, y saltar entre varias otras herramientas de software para confirmar y filtrar esos datos sin sentido. resultados.
"Simplemente me pareció muy ineficiente y no pude superar las deficiencias de otras soluciones de software para este problema", dijo Helf. "Pensé que tenía que haber una manera más fácil, así que comencé a escribir código para mi propio software".
Helf desarrolló la versión inicial de su software en 2017 y continuó mejorándolo durante los siguientes dos años. "Además de abordar los problemas que mis compañeros de laboratorio ya estaban enfrentando, les hablé sobre qué más los frenaba (lo que querían hacer pero ni siquiera lo intentaban) y construí esas características en la aplicación", dijo Helf, quien ahora es gerente de productos de bioinformática en la empresa de proteómica Biognosys AG. "Quería que esta nueva herramienta fuera fácil de usar y accesible para cualquier persona que se dedica a la biología química".
El resultado fue Metaboseek, una aplicación con una interfaz gráfica que incorpora múltiples herramientas de análisis de datos que de otro modo no tendrían los investigadores que no se dedican a la codificación. La aplicación agiliza el análisis de datos de metabolómica comparativa al ayudar al investigador a determinar qué características de datos son reales y permitirles profundizar en esas características, todo dentro de la misma herramienta.
"Max hizo esto sin que yo lo pidiera", dijo Schroeder. "Antes de que supiera que esto estaba sucediendo, estaba Metaboseek. Empezamos a usarlo, y ahora nuestro laboratorio y muchos colaboradores no podrían existir sin él".
En un estudio publicado en Nature Communications el 10 de febrero, el equipo de Schroeder proporcionó una prueba de concepto para Metaboseek al aplicarlo a una vía importante del metabolismo de las grasas que aún no se había estudiado:la vía de oxidación α en C. elegans que ayuda a descomponer una clase de ácidos grasos.
Usando Metaboseek, el equipo descubrió que los gusanos redondos que carecen de un gen clave en la vía de oxidación α acumularon cientos de metabolitos no informados anteriormente. Los hallazgos son importantes porque la α-oxidación es una vía bioquímica básica en los gusanos que se conserva en los humanos, dijo Schroeder.
"Bennett Fox hizo el trabajo de química, por lo que este estudio fue una buena colaboración entre los dos posdoctorados", agregó Schroeder, quien también es profesor en el Departamento de Química y Biología Química de la Universidad de Cornell.
Según Schroeder y Helf, hay algunas razones por las que no hay muchas buenas herramientas analíticas para comparar datos de metabolómica. En primer lugar, la metabolómica comparativa es un campo relativamente joven en comparación con otros campos de la biología con muchos datos, como la genómica (que se centra en el ADN) y la proteómica (que se centra en las proteínas), por lo que no ha habido suficiente tiempo para desarrollar herramientas de software e infraestructura de bases de datos. .
Además, durante la última década, la llegada de espectrómetros de masas asequibles y de ultra alta resolución para recopilar datos metabolómicos ha multiplicado quizás más de diez veces la cantidad de datos que puede generar una muestra, creando una necesidad aún mayor de herramientas sofisticadas que puedan mantener con la avalancha de datos.
Metaboseek satisface estas necesidades con una variedad de características para analizar varios tipos de datos para ayudar a la identificación de compuestos, determinación de estructuras, asignación de metabolitos a familias basadas en similitudes estructurales, seguimiento de compuestos radiomarcados y más. Un paquete R para el análisis integral de datos proteómicos ascendentes centrados en péptidos y proteínas