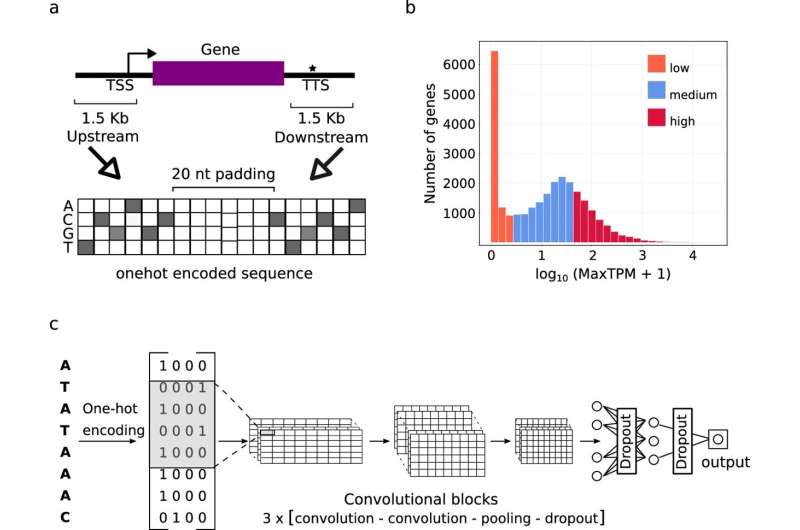
La tecnología de secuenciación del genoma proporciona miles de nuevos genomas de plantas anualmente. En agricultura, los investigadores combinan esta información genómica con datos de observación (que miden varios rasgos de las plantas) para identificar correlaciones entre variantes genéticas y rasgos de los cultivos como el recuento de semillas, la resistencia a infecciones fúngicas, el color o el sabor de la fruta.
Sin embargo, la comprensión de cómo la variación genética influye en la actividad genética a nivel molecular es bastante limitada. Esta brecha en el conocimiento obstaculiza el desarrollo de "cultivos inteligentes" con mayor calidad y menor impacto ambiental negativo logrado mediante la combinación de variantes genéticas específicas de función conocida.
Investigadores del Instituto IPK Leibniz y del Forschungszentrum Jülich (FZ) han logrado un avance significativo para afrontar este desafío. Dirigido por el Dr. Jedrzej Jakub Szymanski, el equipo de investigación internacional entrenó modelos interpretables de aprendizaje profundo, un subconjunto de algoritmos de IA, en un vasto conjunto de datos de información genómica de varias especies de plantas.
"Estos modelos no sólo pudieron predecir con precisión la actividad genética a partir de secuencias, sino también identificar qué partes de la secuencia contribuyen a estas predicciones", explica el jefe del grupo de investigación "Análisis y modelado de redes" del IPK. La tecnología de inteligencia artificial que aplicaron los investigadores es similar a la utilizada en la visión por computadora, que implica reconocer rasgos faciales en imágenes e inferir emociones.
A diferencia de enfoques anteriores basados en el enriquecimiento estadístico, aquí los investigadores combinaron la identificación de las características de la secuencia con la determinación del número de copias del ARNm en el marco de un modelo matemático que ha sido entrenado para tener en cuenta la información biológica sobre la estructura del modelo genético y la homología de secuencia, es decir, el gen. evolución.
"Quedamos realmente sorprendidos por la efectividad. A los pocos días de entrenamiento, redescubrimos muchas secuencias reguladoras conocidas y descubrimos que alrededor del 50% de las características identificadas eran completamente nuevas. Estos modelos se generalizaron excelentemente en especies de plantas en las que no fueron entrenados, lo que hace que resultan valiosos para analizar genomas recién secuenciados", afirma el Dr. Szymanski.
"Y demostramos específicamente su aplicación en diversos cultivares de tomate con datos de secuenciación de lectura larga. Identificamos variaciones específicas de la secuencia reguladora que explicaban las diferencias observadas en la actividad genética y, en consecuencia, las variaciones en la forma, el color y la robustez. Esta es una mejora notable con respecto a asociaciones estadísticas utilizadas clásicamente de polimorfismos de un solo nucleótido."
El equipo compartió abiertamente sus modelos y proporcionó una interfaz web para su uso. "Curiosamente, se dedicó mucho esfuerzo a degradar el rendimiento de nuestro modelo. Para evitar resultados demasiado optimistas debido a que la IA encontró atajos, requirió de mí una inmersión profunda en la biología de la regulación genética para eliminar cualquier posible sesgo, reducir la fuga de datos y el sobreajuste", dice Fritz Forbang Peleke, el investigador principal de aprendizaje automático y primer autor del estudio, que fue publicado en la revista Nature Communications .
El Dr. Simon Zumkeller, coautor y biólogo evolutivo de FZ Jülich, afirma:"Con los análisis presentados podemos investigar y comparar la regulación genética en las plantas e inferir su evolución. Para aplicaciones prácticas, el método también proporciona una nueva base. Nos estamos acercando a la identificación rutinaria de elementos reguladores de genes en genomas de plantas conocidos y recientemente secuenciados, en diversos tejidos y en diferentes condiciones ambientales".
Más información: Fritz Forbang Peleke et al, Aprendizaje profundo del código regulador cis para la expresión genética en plantas modelo seleccionadas, Nature Communications (2024). DOI:10.1038/s41467-024-47744-0
Información de la revista: Comunicaciones sobre la naturaleza
Proporcionado por el Instituto Leibniz de Genética Vegetal e Investigación de Plantas Cultivadas