Un nuevo estudio de la Universidad Hebrea revela conocimientos sobre la regulación del ARNm durante el desarrollo embrionario. El estudio arroja luz sobre el intrincado proceso de regulación del ARNm durante el desarrollo embrionario y proporciona conocimientos novedosos sobre cómo las células pluripotentes adoptan identidades especializadas a través de la expresión genética.
El trabajo está publicado en la revista Nature Communications. .La investigación fue dirigida por Ph.D. El estudiante Lior Fishman y su equipo bajo la dirección del investigador Dr. Michal Rabani del Instituto Alexander Silberman de Ciencias de la Vida de la Universidad Hebrea, y en colaboración con investigadores de los Institutos Nacionales de Salud de EE. UU.
El desarrollo embrionario implica que las células pluripotentes asuman identidades especializadas mediante la adopción de perfiles de expresión genética particulares. Sin embargo, comprender las contribuciones relativas de la transcripción y degradación del ARNm a la configuración de estos perfiles ha sido un desafío, particularmente en embriones con identidades celulares diversas.
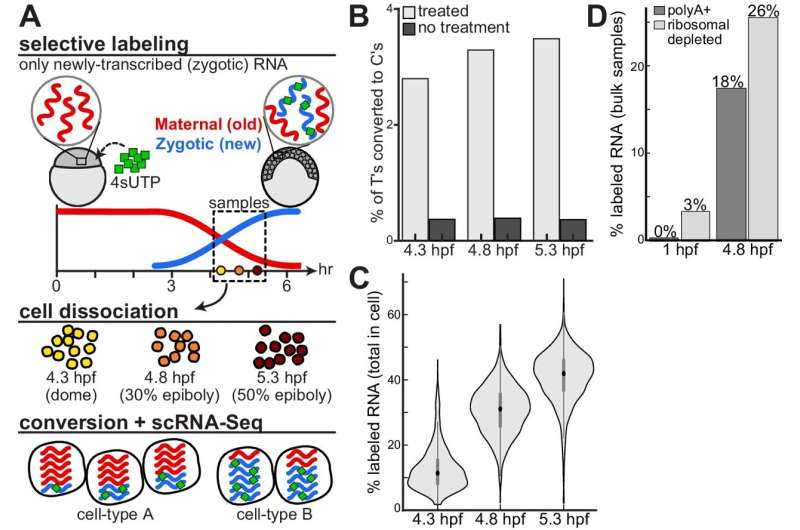
En el estudio, los investigadores utilizaron una técnica llamada secuenciación de ARN unicelular junto con etiquetado metabólico para rastrear cómo se activan y desactivan los genes con el tiempo en embriones de pez cebra. Podían distinguir el ARNm que se había creado nuevo (del propio embrión) y el ARNm que ya estaba allí (de la madre). Utilizando modelos matemáticos, midieron la rapidez con la que se activaban y desactivaban los genes en diferentes tipos de células a medida que se desarrollaban.
Los resultados del estudio revelan tasas regulatorias muy variadas en miles de genes. Los investigadores observaron tasas coordinadas de transcripción y destrucción para muchas transcripciones y vincularon diferencias en la degradación con elementos de secuencia específicos. Es importante destacar que identificaron diferencias en la degradación específicas del tipo de célula, incluida la retención selectiva de transcripciones maternas dentro de las células germinales primordiales y las células de la capa envolvente, dos de los primeros tipos de células especificados.
El Dr. Rabani, autor principal del estudio, comentó:"Nuestro estudio proporciona un enfoque cuantitativo para estudiar la regulación del ARNm durante una respuesta espacio-temporal dinámica. Este trabajo abre nuevas vías para comprender los mecanismos moleculares que subyacen a la determinación del destino celular durante el desarrollo embrionario. "
Los hallazgos de este estudio contribuyen a una comprensión más profunda de la regulación del ARNm y su papel en la configuración de las identidades celulares durante el desarrollo embrionario. El equipo de investigación espera que su trabajo allane el camino para futuros estudios destinados a desentrañar las complejidades de la regulación de la expresión genética en diversos contextos biológicos.
Más información: Lior Fishman et al, Cinética de degradación y transcripción de ARNm específica de tipo celular en la embriogénesis del pez cebra a partir de secuencias de ARN unicelulares metabólicamente marcadas, Nature Communications (2024). DOI:10.1038/s41467-024-47290-9
Información de la revista: Comunicaciones sobre la naturaleza
Proporcionado por la Universidad Hebrea de Jerusalén