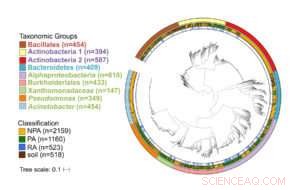
Árbol filogenético de más de 3, 800 genomas bacterianos de alta calidad y no redundantes. El anillo exterior denota el grupo taxonómico, anillo central denota la fuente de aislamiento, y el anillo interior denota los genomas asociados a la raíz dentro de los genomas asociados a las plantas. Los nombres de taxón están codificados por colores según el filo:verde - Proteobacteria, rojo - Firmicutes, azul –Bacteroidetes, púrpura - Actinobacteria. Crédito:Asaf Levy
A medida que aumenta la población mundial, se estima que llegará a casi 10 mil millones para 2050, también lo hace la necesidad de aumentar el rendimiento de los cultivos y producir suficiente material vegetal para la alimentación y los combustibles alternativos sostenibles. Para ayudar a mejorar las estrategias de mejoramiento de cultivos y superar desafíos como hacer que las plantas sean más tolerantes con las tierras marginales, y tensiones como la sequía y la baja disponibilidad de nutrientes, los investigadores se centran en comprender y promover las relaciones beneficiosas entre plantas y microbios.
Publicado el 18 de diciembre de Edición de 2017 en Genética de la naturaleza , un equipo dirigido por investigadores del Instituto Conjunto del Genoma (JGI) del Departamento de Energía de EE. UU. (DOE), una instalación para usuarios de la Oficina de Ciencias del DOE, y el Instituto Médico Howard Hughes de la Universidad de Carolina del Norte en Chapel Hill (UNC) han explotado un catálogo de genomas bacterianos para identificar y caracterizar genes candidatos que ayudan a las bacterias a adaptarse a los entornos de las plantas. específicamente genes implicados en la colonización de raíces bacterianas.
La mayoría de los estudios de campo hasta la fecha se han centrado en la estructura comunitaria del microbioma vegetal, es decir, "quién está allí, "y menos en la función, es decir, "lo que están haciendo, cómo y cuándo lo están haciendo ". Estudios previos que han considerado la función han analizado principalmente una única interacción huésped-microbio, como el que se encuentra entre una planta de Arabidopsis y un patógeno.
"Si queremos diseñar el microbioma adecuado para apoyar el crecimiento de las plantas, necesitamos comprender la función real del microbioma y no solo los genes marcadores de secuencia, "dijo el coautor principal del estudio, Asaf Levy, científico investigador del JGI. "Aquí utilizamos un esfuerzo genómico y computacional masivo para abordar la pregunta fundamental e importante:'¿Cómo interactúa el microbioma de la planta con la planta?'"
La mayor parte de la interacción entre microbios y plantas ocurre en la interfaz entre las raíces y el suelo. Investigadores de UNC, Laboratorio Nacional Oak Ridge, y el Instituto Max Planck aisló bacterias nuevas del entorno de las raíces de Brassicaceae (191), álamos (135), y maíz (51). Los genomas de estos 377 aislamientos bacterianos, más 107 células bacterianas individuales adicionales de raíces de A. thaliana, luego fueron secuenciados, ensamblado y anotado en el JGI.
Luego, los autores combinaron los nuevos genomas con miles de genomas disponibles públicamente que representan los principales grupos de bacterias asociadas a plantas. e incluyó bacterias de múltiples entornos vegetales y no vegetales, como el intestino humano, para comparacion. La base de datos resultante de 3837 genomas, 1160 de los cuales son de plantas, se utilizó en un análisis de genómica comparativa.
Luego, los investigadores identificaron genes que están enriquecidos en los genomas de organismos asociados a plantas y raíces.
"Es muy importante para nosotros comprender qué genes y funciones usan los microbios para colonizar plantas porque solo entonces tendremos la oportunidad de idear racionalmente 'probióticos de plantas' útiles para ayudarnos a producir más cultivos alimentarios y energéticos con menos insumos químicos como fertilizantes y pesticidas o fungicidas, "dijo el autor principal del estudio, Jeff Dangl, investigador del Instituto Médico Howard Hughes y profesor de biología John N. Couch en la Universidad de Carolina del Norte en Chapel Hill
Entre las ideas clave obtenidas del estudio se encuentra que los genomas asociados a plantas y suelos tienden a ser más grandes que los genomas de control del mismo clado. Se descubrió que esto se debía en parte al enriquecimiento de genes implicados en el metabolismo y transporte del azúcar, probablemente una adaptación al carbono vegetal derivado de la fotosíntesis, generado por las "fábricas de dulces de la naturaleza, ", dijo Asaf Levy. Hasta el 20% del carbono fijado por las plantas a través de la fotosíntesis se exuda a través de las raíces como azúcares para atraer microbios.
También se identificaron numerosos genes que parecen imitar las funciones de las plantas, codificando "dominios PA y RA similares a plantas" o PREPARADO. "Es bien sabido que los patógenos vegetales utilizan proteínas que imitan los dominios vegetales necesarios para la función inmunológica, ", dijo Dangl." Imagine que el patógeno inyecta directamente en la célula vegetal una proteína que imita parte de una máquina del sistema inmunológico en particular. Es como poner un engranaje parcialmente defectuoso en una rueda:las ruedas ya no pueden girar. Creemos que los dominios proteicos asociados a plantas que identificamos podrían funcionar de la misma manera ".
Los genes que evolucionan rápidamente son a menudo la firma de una carrera de armamentos moleculares entre organismos que comparten un medio ambiente. Estos genes se utilizan a menudo para atacar o defenderse de otros organismos. En el estudio se identificaron dos nuevas familias de proteínas de rápida evolución asociadas con diferentes "estilos de vida" de bacterias relacionadas asociadas a plantas. Uno, encontrado en bacterias comensales, fue apodado "Jekyll"; el otro, encontrado en bacterias patógenas, fue nombrado "Hyde". Con colaboradores de Virginia Tech y ETH (Suiza), Los científicos de JGI descubrieron que estos últimos son muy eficientes para matar bacterias competidoras, potencialmente para ayudar a estos "Hydes" a hacerse cargo del nicho de las hojas.
El catálogo completo de nuevos genomas y genes asociados a plantas está disponible para la comunidad de investigadores a través de un portal web dedicado:Características genómicas de la adaptación bacteriana a las plantas.
"La base de datos es un recurso valioso para la comunidad de investigación que estudia las interacciones planta-microbio, ya que es una forma imparcial de identificar genes potencialmente interesantes involucrados en la interacción con una planta, incluidos muchos genes totalmente nuevos. Actualmente estamos estudiando experimentalmente la función de muchos de estos genes para obtener una mejor comprensión funcional del microbioma vegetal ". Dijo Levy.