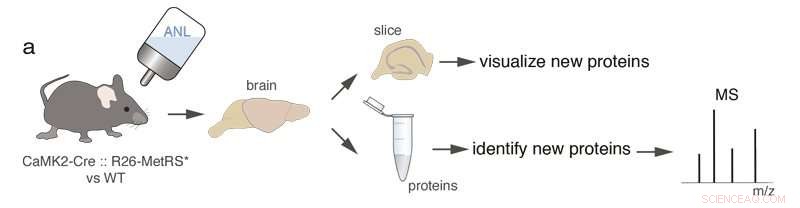
Un ratón transgénico que expresa una mutante-metionil tRNA sintetasa en células específicas (células CamK2a-positivas) se alimenta con un aminoácido no canónico (ANL) que se incorpora a las proteínas recién sintetizadas solo en las células específicas. Usando química de clic, Las proteínas recién sintetizadas se etiquetan para su visualización o identificación. Crédito:Brain Research
La complejidad de los seres vivos está impulsada, en gran parte, por la enorme diversidad de tipos de células. Dado que todas las células de un organismo comparten los mismos genes, la diversidad de células debe provenir de las proteínas particulares que se expresan. Las células del cerebro generalmente se dividen en neuronas y glía. Dentro de estas dos categorías, sin embargo, yace una gran diversidad de tipos de células que apenas estamos empezando a descubrir. La diversidad de tipos de células en el cerebro y otros tejidos se ha ampliado recientemente mediante nuevas técnicas, como la secuenciación de ARN, que identifican y miden los ARNm presentes en una célula, el llamado transcriptoma. Aunque los ARNm son la plantilla para las proteínas, el transcriptoma es un pobre proxy de las proteínas que produce una célula, el proteoma. Los científicos del Instituto Max Planck para la Investigación del Cerebro en Frankfurt ahora desarrollaron nuevos métodos para detectar cambios en tiempo real en el proteoma.
Basándose en tecnología anterior, desarrollado por el Laboratorio de Erin Schuman en el Instituto Max Planck y los colaboradores David Tirrell de Caltech y Daniela Dieterich de la Universidad de Magdeburg, Beatriz Alvarez-Castelao y sus colegas aprovecharon un sistema de etiquetado "metabólico" de proteínas. En este sistema, las proteínas durante la síntesis están "etiquetadas" con un aminoácido, cual es, bajo condiciones normales, no presente en estas células. Para etiquetar proteínas en un tipo de célula en particular exclusivamente, el equipo de investigación utilizó una metionil tRNA sintetasa mutante (MetRS) que reconoce el aminoácido modificado. Luego crearon una línea de ratón en la que MetRS se puede expresar en tipos de células específicos. Cuando el aminoácido no canónico se administra a los ratones MetRS mutantes a través del agua potable, sólo se marcan las proteínas de las células que expresan la metRS mutante.
Las proteínas marcadas en las células se pueden visualizar y reconocer con anticuerpos o se pueden extraer e identificar mediante espectrometría de masas. Álvarez-Castelao:"Usamos la técnica para identificar dos conjuntos diferentes de proteínas cerebrales, los presentes en las neuronas excitadoras del hipocampo, una estructura cerebral importante para la navegación, el aprendizaje y la memoria de los animales, y neuronas inhibidoras en el cerebelo, una estructura involucrada en el comportamiento motor ".
Una característica particularmente llamativa de esta tecnología es que se pueden detectar directamente cambios en las proteínas cerebrales en respuesta a un entorno modificado. Ratones que se criaron en un entorno sensorial enriquecido con un laberinto, rueda corriente, y juguetes de texturas variadas mostraron cambios significativos en el proteoma en el hipocampo, particularmente en proteínas que trabajan en sinapsis neuronales. Schuman:"Creemos que, al combinar este ratón con otros modelos de ratón "patológico", Este método se puede utilizar para descubrir las proteínas en determinados tipos de células y cómo cambian los proteomas durante el desarrollo del cerebro. aprendiendo, memoria y enfermedad ".