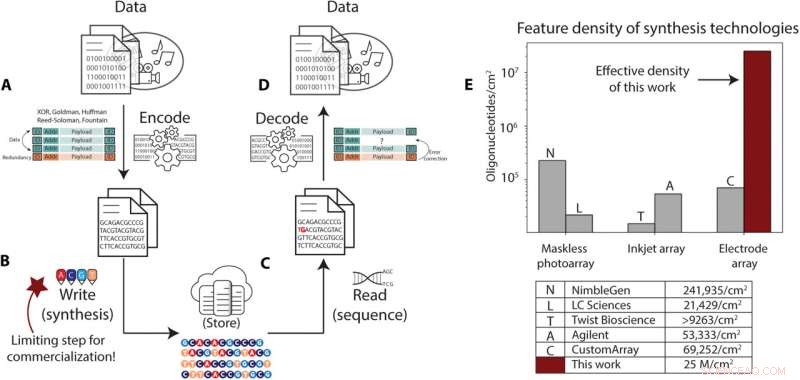
El almacenamiento de datos de ADN requiere un mayor rendimiento de síntesis de lo que es posible con las técnicas actuales. (A a D) Descripción general de la canalización de almacenamiento de datos de ADN. (A) Los datos digitales se codifican a partir de su representación binaria en secuencias de bases de ADN, con un identificador que los correlaciona con un objeto de datos, abordando información que se usa para reordenar los datos al leer e información redundante que se usa para la corrección de errores. (B) Estas secuencias se sintetizan en oligonucleótidos de ADN y se almacenan. (C) En el momento de la recuperación, las moléculas de ADN se seleccionan y copian mediante PCR u otros métodos y se vuelven a secuenciar en representaciones electrónicas de las bases en estas secuencias. (D) El proceso de decodificación toma este ruidoso ya veces incompleto conjunto de lecturas de secuencias, corrige los errores y las secuencias faltantes y decodifica la información para recuperar los datos. (E) Resumen de los procesos de síntesis comerciales y densidades de oligonucleótidos estimadas correspondientes, según lo informado en la literatura o por las propias empresas. La densidad de nuestro método electroquímico está resaltada en rojo oscuro. Crédito:Avances científicos , 10.1126/sciadv.abi6714
Los genetistas pueden almacenar datos en ADN sintético como medio de almacenamiento a largo plazo debido a su densidad, facilidad de copia, longevidad y sostenibilidad. La investigación en el campo había avanzado recientemente con nuevos algoritmos de codificación, automatización, preservación y secuenciación. No obstante, el obstáculo más desafiante en la implementación del almacenamiento de ADN sigue siendo el rendimiento de escritura, que puede limitar la capacidad de almacenamiento de datos. En un nuevo informe, Bichlien H. Nguyen y un equipo de científicos de Microsoft Research y ciencias informáticas e ingeniería de la Universidad de Washington, Seattle, EE. UU., desarrollaron el primer escritor de almacenamiento de ADN a nanoescala. El equipo pretendía escalar la densidad de escritura de ADN a 25 x 10 6 secuencias por centímetro cuadrado, una capacidad de almacenamiento mejorada en comparación con las matrices de síntesis de ADN existentes. Los científicos escribieron y decodificaron con éxito un mensaje en el ADN para establecer un sistema práctico de almacenamiento de datos de ADN. Los resultados ahora se publican en Science Advances .
Archivos de ADN a largo plazo
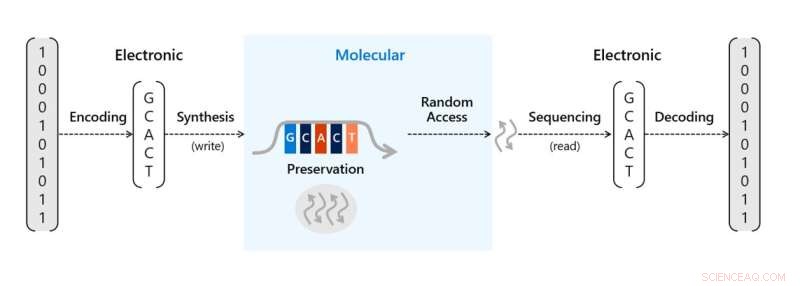
El ritmo actual de generación de datos supera las capacidades de almacenamiento existentes, el ADN es una solución prometedora para este problema con una densidad práctica esperada de más de 60 petabytes por centímetro cúbico. El material es duradero bajo una variedad de condiciones, relevante y fácil de copiar, con la promesa de ser más sostenible o más ecológico que los medios comerciales. Durante el proceso, los datos digitales en forma de secuencias de bits pueden codificarse en secuencias de las cuatro bases naturales del ADN:guanina, adenina, tiamina y citosina, aunque también son posibles bases adicionales. A continuación, el equipo puede escribir las secuencias en forma molecular a través de la síntesis de oligonucleótidos de ADN de novo para crear moléculas específicas basadas en un conjunto de pasos químicos repetitivos. Los oligonucleótidos resultantes pueden conservarse y almacenarse después de la síntesis. Para acceder a los datos, el almacenamiento de ADN puede amplificarse mediante reacciones en cadena de la polimerasa y secuenciarse para devolver las secuencias de bases de ADN al dominio digital, luego las secuencias de bases de ADN pueden decodificarse para recuperar la secuencia original de bits.
Descripción general de la matriz de 650 nm inclinada 2 μm. (A) El análisis de elementos finitos de la generación y difusión de ácido anódico en un electrodo de 650 nm de diámetro con un pozo de 200 nm se representa con una vista transversal a lo largo del plano y =x y (B) vista de arriba hacia abajo en el plano z =0. Los colores azul y amarillo representan regiones con concentraciones de ácido relativamente bajas y altas, respectivamente. (C) Una descripción general de la matriz de síntesis de ADN a nanoescala con imágenes de microscopía electrónica de barrido de la matriz de electrodos de 650 nm y una vista ampliada de un electrodo. (D) Una imagen fluorescente en la que el pozo que rodea a cada ánodo activado está modelado con fluoresceína AAA. El diagrama de dibujos animados muestra qué electrodos en el diseño se activaron. (E) Ilustración de los pocillos modelados con AAA-fluoresceína y AAA-AquaPhluor y (F) superposición de imagen correspondiente de los dos fluoróforos al final del ADN sintetizado en la misma matriz de electrodos de 650 nm. Crédito:Avances científicos , 10.1126/sciadv.abi6714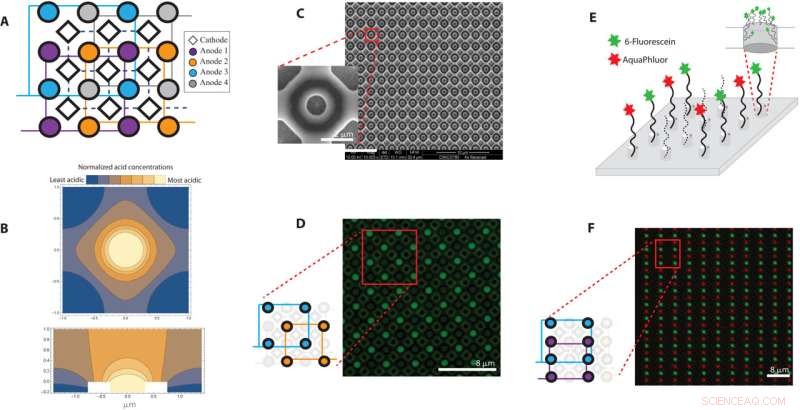
En este estudio, Nguyen et al. produjo una matriz de electrodos que demostró un control específico de electrodos independiente de la síntesis de ADN con tamaños y pasos de electrodos para establecer una densidad de síntesis de 25 millones de oligonucleótidos por cm 2 . Este valor se estima como la densidad de electrodos necesaria para lograr el objetivo mínimo de kilobytes por segundo de almacenamiento de datos en el ADN. El equipo impulsó el control químico-electrónico de última generación y proporcionó pruebas experimentales del ancho de banda de escritura necesario para el almacenamiento de datos de ADN.
El equipo introdujo un controlador molecular de prueba de concepto en forma de un pequeño mecanismo de escritura de almacenamiento de ADN en un chip. El chip podría empaquetar la síntesis de ADN en 3 órdenes de magnitud más que antes para lograr un mayor rendimiento de escritura de ADN. Almacenar información en el ADN a la escala necesaria para uso comercial requería dos procesos cruciales. Primero, el equipo tuvo que traducir los bits digitales (unos y ceros) en hebras de ADN sintético que representaban bits con un software de codificación y un sintetizador de ADN. Luego, deben poder leer y decodificar la información hasta sus bits para recuperar esa información en forma digital nuevamente con un secuenciador de ADN y un software de decodificación.
Desarrollo de matrices electroquímicas para funciones a nanoescala
Durante la síntesis tradicional de cadenas de ADN, los científicos utilizan un método de varios pasos conocido como química de fosforamidita, en el que una cadena de ADN puede crecer secuencialmente mediante la adición de bases de ADN. Cada base de ADN contiene un grupo de bloqueo para evitar múltiples adiciones de bases de ADN a la cadena en crecimiento. Al unirse a una cadena de ADN, se puede administrar ácido en la configuración para escindir el grupo de bloqueo y cebar la cadena de ADN para agregar la siguiente base. Durante la síntesis electroquímica de ADN, cada punto de la matriz contiene un electrodo y cuando se aplica un voltaje, se genera ácido en el electrodo de trabajo (ánodo) para desbloquear las cadenas de ADN en crecimiento, mientras que se genera una base equivalente en el contraelectrodo (cátodo) . El equipo evitó la difusión de ácido en la configuración mediante el diseño de una matriz de electrodos, donde cada electrodo de trabajo alrededor del cual se produjo la formación de ácido durante la síntesis de ADN se hundió en un pozo y se rodeó de cuatro contraelectrodos comunes, es decir, cátodos que impulsaron la formación de base, para confinar el ácido a regiones específicas. Nguyen et al. verificó la efectividad del diseño utilizando análisis de elementos finitos. Durante los experimentos, cuando se presentó en suficiente concentración, el ácido desbloqueó los nucleótidos unidos a la superficie para permitir que se acoplara el siguiente nucleótido. Usando la configuración de chips que contienen puntos característicos para confinar ácidos, desarrollaron matrices electroquímicas con cuatro electrodos individuales para regular la síntesis de ADN. Luego, el equipo realizó experimentos con dos bases marcadas con fluorescencia en verde y rojo. Como prueba de concepto, demostraron la capacidad del dispositivo para escribir datos mediante la síntesis de cuatro hebras de ADN únicas, cada una de 100 bases de longitud con un mensaje codificado, sin errores.
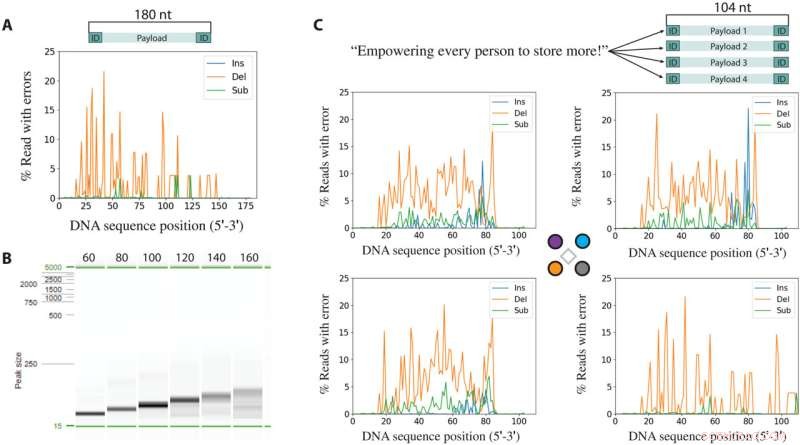
Errores derivados de la síntesis seguida de secuenciación. (A) Inserciones (Ins), deleciones (Del) y sustituciones (Sub) por posición para una secuencia de 180 bases sintetizada y amplificada por PCR. (B) Imagen de electroforesis de los productos de síntesis después de la amplificación por PCR. (C) Mensaje codificado en 64 bytes divididos en cuatro secuencias únicas de 104 bases (arriba). Inserciones, eliminaciones y sustituciones por locus de cada una de las cuatro secuencias en la ejecución de síntesis multiplex. En cada gráfico de análisis de errores, las 20 bases terminales en los extremos 3' y 5' provienen de los cebadores utilizados en la PCR y no son representativas de los errores sintetizados. Crédito:Avances científicos , 10.1126/sciadv.abi6714
Escalado del almacenamiento de datos de ADN con pozos de electrodos a nanoescala. Pequeño mecanismo de escritura de almacenamiento de ADN en un chip. Crédito:Blog de investigación de Microsoft, Science Advances , 10.1126/sciadv.abi6714
Outlook:sintetizar oligonucleótidos cortos en la matriz de electrodos para el almacenamiento de datos
Usando la configuración, Nguyen et al. también demostró la síntesis controlada espacialmente de oligonucleótidos cortos en la matriz de electrodos para evaluar la longitud máxima de ADN que se podría formar. Los científicos crearon una sola secuencia de ADN con 180 nucleótidos y productos de varias longitudes amplificados por PCR a partir de la longitud completa de los oligonucleótidos. A medida que el amplicón se alargaba, los productos de PCR esperados aparecían más débiles y menos definidos, mientras que los amplicones más cortos mostraban bandas más fuertes y mejor definidas, indicativas de mayores errores de síntesis. Basándose en los resultados, los investigadores seleccionaron una longitud de secuencia de 100 bases para facilitar la purificación y proporcionar una demostración práctica del almacenamiento de datos de ADN sin más optimización. De esta manera, el método de prueba de concepto demostrado en este trabajo por Bichlien H. Nguyen y sus colegas allanó el camino para generar secuencias de ADN únicas y a gran escala en paralelo para el almacenamiento de datos. El trabajo superó los informes anteriores sobre secuencias densas de ADN sintético para proporcionar una primera indicación experimental para lograr el ancho de banda de escritura requerido para el almacenamiento de datos en tamaños de características a nanoescala. Los científicos esperan aplicaciones inmediatas de los dispositivos en tecnología de la información y prevén sus aplicaciones prácticas en ciencia de materiales, biología sintética y ensayos de biología molecular a gran escala.
© 2021 Ciencia X Network La síntesis enzimática de ADN ve la luz