
Técnica de nanoendoscopia-AFM 3D. (A) Esquema del método de nanoendoscopia-AFM 3D, donde la nanosonda se introduce repetidamente dentro de la célula en diferentes posiciones en el área deseada. (B) Nanosonda fabricada por FIB utilizada en las mediciones de nanoendoscopia-AFM 3D. (C y D) Curvas F-z típicas obtenidas al penetrar una célula, donde una disminución abrupta de la desviación del voladizo se representa como un pico cuando la nanosonda penetra la membrana celular externa (C), apareciendo como otro pico en caso de que la nanosonda atraviese la membrana nuclear (D). (G) Mapa celular de nanoendoscopia-AFM 3D de todo el volumen celular HeLa (40 × 40 × 6 μm3) encerrado en el cuadrado rojo en (E), donde la membrana celular, el núcleo y las regiones citoplásmicas se pueden distinguir en la sección transversal se muestra en (H). (I) Imagen de nanoendoscopia-AFM 3D de un volumen de células HeLa (10 × 10 × 6 μm3) encerrado en el cuadrado rojo en (F), donde las estructuras granulares internas pueden reconocerse claramente. Crédito:Avances científicos , 10.1126/sciadv.abj4990
La microscopía de fuerza atómica (AFM) ofrece un método para obtener imágenes sin etiquetas de la dinámica biomolecular a nanoescala para resolver cuestiones biológicas que no se pueden abordar a través de otros métodos de bioimagen, como la fluorescencia y la microscopía electrónica de barrido. Dado que tales métodos de imagen solo son posibles para sistemas biológicos extraídos de células o reconstruidos en sustratos sólidos, la nanodinámica dentro de las células vivas sigue siendo en gran medida inaccesible con los métodos de bioimagen existentes. En un nuevo informe publicado ahora en Science Advances , Marcos Penedo y un equipo de investigación en Nanolife Science y biotecnología de la Universidad de Kanazawa en Japón, superaron los límites de la bioimagen mediante el uso de nanoendoscopia-AFM. Durante el proceso, insertaron una sonda con forma de aguja en una célula viva para presentar fibra de actina, mapas tridimensionales (3D) y nanodinámica 2D del andamio interno de la membrana con cambios indetectables en la viabilidad celular. A diferencia de los métodos anteriores de AFM, la nanosonda accedió directamente a los componentes intracelulares objetivo y exploró las capacidades de AFM, incluidas las imágenes de alta resolución, el mapeo nanomecánico y el reconocimiento molecular para expandir el rango observable de estructuras intracelulares en células vivas.
Dinámica intracelular de bioimagen
La dinámica a escala molecular de los componentes intracelulares proporciona información sobre los mecanismos fundamentales de las funciones y enfermedades celulares. Sin embargo, los métodos de obtención de imágenes directas para tales nanodinámicas en células vivas son un desafío. Por ejemplo, mientras que la microscopía electrónica es útil para obtener imágenes de nanoestructuras de células congeladas en el vacío, son incapaces de obtener imágenes de la nanodinámica en células vivas en entornos fisiológicos, excepto como instantáneas estáticas de conformaciones fijas. De manera similar, mientras que la microscopía de fluorescencia a través del etiquetado de fluorescencia proporciona un método poderoso para visualizar la dinámica de las proteínas y los orgánulos en las células vivas, están limitadas por la incapacidad de generar imágenes de manera eficiente a nanoescala. Por lo tanto, existe una fuerte demanda de un método de formación de imágenes intracelulares sin etiquetas en entornos líquidos. La microscopía de fuerza atómica (AFM) es un candidato potencial para el papel con la capacidad de generar imágenes a escala subnanométrica para visualizar la nanodinámica de lípidos, proteínas y ADN sin etiquetas. Sin embargo, tales imágenes no son representativas de los sistemas biológicos como resultado de la extracción de una célula o la reconstrucción en un sustrato sólido in vitro. En este trabajo, por tanto, Penedo et al. propuso un método de imagen basado en AFM conocido como nanoendoscopia-AFM para observar la nanodinámica dentro de las células vivas sin etiquetarlas ni separarlas.
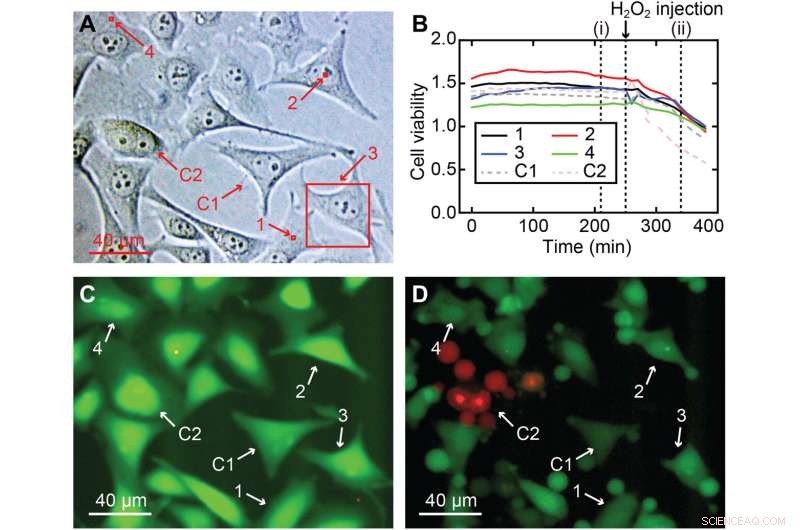
Viabilidad celular de las mediciones de nanoendoscopia-AFM 3D. (A) Diferentes áreas medidas realizadas en un cultivo celular HeLa para la prueba de viabilidad celular, incluidas las regiones del núcleo y la periferia celular:(1) 2 × 2 × 7 μm3, (2) 2 × 2 × 10 μm3, (3) 40 × 40 × 8 μm3 y (4) 2 × 2 × 7 μm3, resaltados en cuadrados rojos; se usaron dos celdas como control, C1 y C2. (B) Proporciones de viabilidad celular a lo largo del tiempo para las cuatro células con imágenes (1 a 4) y para las dos células utilizadas como control (C1 y C2), que muestran que todas las células (con imágenes y control) tenían una intensidad de viabilidad celular plana similar proporción y confirmando que las células no estaban muy dañadas. (C) Ejemplo de una imagen de fluorescencia después de 210 min correspondiente a (i) en (B), donde el color verde fuerte significa una actividad de esterasa normal esperada para una célula viva. Para comprobar la validez del ensayo, se añadió H2O2 después de 260 min al medio para matar las células, lo que provocó una disminución de los índices de viabilidad celular de todas las células, una clara indicación de que las células estaban muriendo. (D) Instantánea de fluorescencia correspondiente al tiempo (ii) en (B), donde los signos de daño son claramente visibles en todas las células, la mayoría de las cuales ya sufrieron encogimiento o apoptosis. Crédito:Avances científicos , 10.1126/sciadv.abj4990
Experimentos de nanoendoscopia-AFM
Durante los experimentos, al igual que una cámara endoscópica, los investigadores insertaron una nanosonda con forma de aguja larga dentro de una célula viva para realizar imágenes AFM en 2D y 3D. El equipo mostró cómo la nanoendoscopia-AFM proporcionó una ventaja única para la obtención de imágenes de células vivas intracelulares sin etiquetas a nanoescala. El método proporciona un poderoso camino para observar fenómenos hasta ahora inexplorados en sistemas biológicos. Penedo et al. introdujo repetidamente la nanosonda dentro de la celda en diferentes posiciones del área deseada a través de mediciones de curva de fuerza versus distancia. Para obtener imágenes de toda la célula, la nanosonda tenía que ser lo suficientemente larga para penetrar completamente en la célula hasta alcanzar el sustrato, y con diámetros inferiores a 200 nm para minimizar el daño celular y facilitar la penetración de la membrana. El equipo usó una punta tetraédrica de silicio comercial como nanosonda, que molieron usando un haz de iones enfocado a las dimensiones preferidas. A continuación, el equipo utilizó las nanosondas dentro de diferentes áreas de una célula HeLa. Adquirieron una imagen de nanoendoscopia-AFM en 3D de una célula completa durante los experimentos e identificaron el núcleo de la célula HeLa del resto de la célula. Otras mediciones también indicaron las estructuras granulares internas. Para minimizar el daño celular durante la penetración, Penedo et al. redujo la fuerza de penetración y la longitud de la muesca tanto como fue posible. También realizaron experimentos de viabilidad celular para confirmar que la nanoendoscopia-AFM 3D no provocaba daños celulares graves al utilizar nanosondas con diámetros inferiores a 200 nm. Usando nanoendoscopia 3D-AFM, facilitaron la obtención de imágenes del citoesqueleto interno en células vivas para observar la organización 3D de las fibras sin soporte. El equipo también fusionó con éxito imágenes intracelulares resultantes de nanoendoscopia-AFM 3D y microscopia confocal.

Combinación de imágenes confocales y nanoendoscopia-AFM 3D. (A) Imagen de fluorescencia confocal donde se ven los filamentos de actina teñidos. (B) La imagen ampliada obtenida en el área indicada por el cuadrado rojo en (A). (C y D) Mapas de nanoendoscopia-AFM 3D de las fibras de actina del citoesqueleto obtenidos en el área resaltada por el cuadrado rojo en (B), donde las posiciones verticales Z de los diferentes filamentos de actina (flechas rojas) y las membranas celulares superior e inferior se resuelven simultáneamente. La imagen semitransparente que se muestra en el cuadrado rojo en (B) corresponde a la proyección 2D de los mapas 3D que se muestran en (C) y (D). Crédito:Avances científicos , 10.1126/sciadv.abj4990
Nanoendoscopia 2D-AFM
La capacidad de insertar una nanosonda larga en una célula muchas veces mientras se mantiene la viabilidad de la célula implicaba la posibilidad de ubicar el vértice de la sonda dentro de una célula viva para realizar mediciones AFM 2D/3D locales sin daños sustanciales. La nanosonda podría insertarse dentro de la célula para medir el lado citoplásmico de la membrana celular a través del modo de modulación de amplitud AFM. Las nanosondas tenían que ser lo suficientemente largas para penetrar completamente en la célula y llegar al fondo, y al mismo tiempo lo suficientemente delgadas para reducir el daño celular. Para lograr esto en la práctica, Penedo et al. desarrolló nanosondas hechas de carbono amorfo utilizando la deposición de un haz de electrones y midió la dependencia de la amplitud con la distancia para determinar la integridad de la célula. Realizaron experimentos de nanoendoscopia-AFM en 2D utilizando una célula de fibroblastos para ilustrar la estructura reticular de la membrana celular interna y observaron la arquitectura celular para estudiar la dinámica interna de las estructuras celulares. El trabajo destacó la posibilidad de utilizar nanoendoscopia-AFM 2D para estudiar la nanodinámica de estructuras internas en células vivas en entornos fisiológicos.

Técnica de nanoendoscopia-AFM 2D. (A) Ilustración del método de nanoendoscopia-AFM 2D, donde la nanosonda se inserta dentro de la célula para medir el lado citoplásmico de la membrana celular mediante el modo de modulación de amplitud AFM. (B) Ejemplo de una nanosonda fabricada con EBD utilizada en nanoendoscopia-AFM 2D, donde la longitud de la aguja debe ser lo suficientemente larga para penetrar completamente la célula y llegar a su parte inferior y lo suficientemente delgada para reducir el daño celular. (C) Fuerza registrada (arriba) y amplitud (abajo) versus curvas de distancia para ubicar con precisión las membranas celulares superior e inferior:La fuerza vertical es nula cuando la nanosonda está lejos, aumentando tan pronto como la nanosonda toca la membrana celular superior; posteriormente, presenta una meseta que corresponde al dominio citoplasmático interno hasta que la curva vuelve a aumentar bruscamente cuando la nanosonda alcanza la membrana celular inferior. El punto de ajuste de amplitud para la regulación de la distancia entre la punta y la muestra debe ser lo suficientemente bajo para garantizar que la punta toque la superficie inferior de la celda. (F) Imágenes consecutivas de nanoendoscopia-AFM 2D de 1 μm × 1 μm realizadas en un fibroblasto BALB/3T3 en la región resaltada por el punto rojo representado en (D), que muestra la estructura reticular de la superficie interna de la membrana celular que forma su andamiaje y también las fluctuaciones de la membrana durante las mediciones. (G) Área ampliada de las imágenes mostradas en (F), trazando una sección entre los puntos A y B (E), donde dos protuberancias separadas por 25 nm se resuelven claramente en la imagen. Crédito:Avances científicos , 10.1126/sciadv.abj4990
Perspectiva
De esta manera, Marcos Penedo y sus colegas mostraron las aplicaciones de la nanoendoscopia-AFM para medir las superficies internas citoplásmicas de las membranas celulares y los andamios asociados para comprender la disposición 3D de los filamentos de actina en su entorno intracelular natural en las células vivas. El equipo trató de minimizar el daño celular mediante el uso de nanosondas ultrafinas en forma de aguja en los experimentos. Los métodos AFM propuestos produjeron mapas 3D de estructuras celulares internas además de proyecciones 2D combinadas con métodos de fluorescencia existentes como la microscopía confocal o de superresolución. El método arrojará luz sobre la maquinaria celular en acción, in vivo, al tiempo que expone los motores moleculares fisiológicos. El método también abrirá nuevas posibilidades para estudiar la nanomecánica intracelular que juega un papel importante en las funciones celulares. El equipo puede utilizar el método para medir las características de rigidez, adhesión y disipación del núcleo para extraer información biológica adecuada para los campos interdisciplinarios de la biología celular y la medicina.
© 2022 Red Ciencia X El nuevo método de imágenes ofrece una visión en vivo de cómo funcionan las células