Crédito:Sociedad Química Estadounidense
Los científicos de todo el mundo se apresuran a encontrar inhibidores del SARS-CoV-2, el nuevo coronavirus detrás de la pandemia COVID-19. Algunos están usando simulaciones por computadora para identificar compuestos prometedores antes de realizar experimentos reales en el laboratorio. Ahora, investigadores que informan en ACS Nano han utilizado modelos informáticos para evaluar cuatro péptidos que imitan el dominio de unión al virus de la proteína humana que permite que el SARS-CoV-2 ingrese a las células.
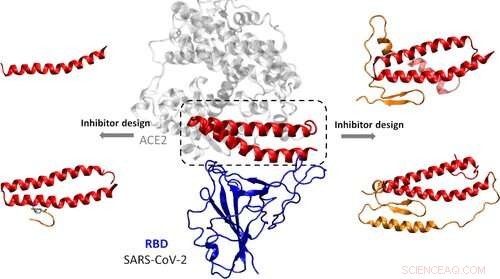
Para infectar células, El SARS-CoV-2 utiliza su proteína de pico para unirse al receptor ACE2, una proteína en la superficie de ciertas células humanas. Esta unión permite que el virus se fusione con la membrana de la célula huésped y gane la entrada. Muchos investigadores han estado tratando de encontrar compuestos que bloqueen regiones clave de la proteína de pico, evitar que el virus infecte las células. Yanxiao Han y Petr Král querían utilizar modelos informáticos para diseñar compuestos que imitaran el objetivo natural de la proteína de pico, ACE2.
Para hacerlo Los investigadores examinaron la estructura cristalina de rayos X publicada recientemente del dominio de unión al receptor del SARS-CoV-2 cuando está unido a ACE2. Identificaron 15 aminoácidos de ACE2 que interactúan directamente con la proteína viral. Luego, los investigadores diseñaron cuatro inhibidores que contienen la mayoría o todos estos aminoácidos, con secuencias adicionales que pensaron que estabilizarían las estructuras. A través de simulaciones por computadora, el equipo estudió cómo los inhibidores podrían adherirse a la proteína de pico en el cuerpo y las energías necesarias para unirse. Uno de los compuestos mostró un ajuste particularmente bueno con la proteína viral. El péptido aún debe probarse en el laboratorio y en pacientes, pero poder reducir los candidatos a fármacos en la computadora podría ayudar a acelerar este proceso, dice el equipo.