
Extracto de la película de lapso de tiempo de detección de miARN en 1 hora usando el ensayo miRacles. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443
Los microARN (miARN) son ARN reguladores cortos no codificantes que pueden reprimir la expresión génica postranscripcionalmente y, por lo tanto, se utilizan cada vez más como biomarcadores de enfermedades. La detección de miARN puede resultar ardua y costosa, ya que requieren amplificación, sondas de marcaje y radiactivas. En un informe reciente publicado en Avances de la ciencia , Arun Richard Chandrasekaran y colaboradores del Instituto y Departamento de Ciencias Biológicas de ARN, en la Universidad de Albany, Universidad Estatal de Nueva York, informó en un solo paso, ensayo de detección de microARN no enzimático utilizando nanointerruptores de ADN conformacionalmente que no responden.
Los científicos llamaron al ensayo 'miRacles, 'para abreviar' bucle condicional activado por micro-ARN de interruptores diseñados '. El ensayo demostró especificidad subatomol y de un solo nucleótido utilizando una lectura de electroforesis en gel de agarosa. En los experimentos, detectaron microARN celulares a partir de extracciones de ARN obtenidos de diferenciar músculos a escala de nanogramos. Los científicos presentaron una configuración experimental rentable para detectar miARN en un marco de tiempo de horas para proporcionar una alternativa convincente a los métodos existentes de reacción en cadena de la polimerasa cuantitativa (qPCR) y transferencia Northern para cuantificar los materiales genéticos reguladores.
Los miARN pueden regular muchos procesos biológicos durante el desarrollo fisiológico normal y la enfermedad al afectar la proliferación celular. diferenciación y apoptosis in vivo. La expresión de miARN se puede cuantificar en tejidos, células y fluidos corporales como biomarcadores estables para eventos celulares y diagnóstico de enfermedades, destacando la importancia de su detección. Sin embargo, La detección de miARN es un desafío debido a la baja abundancia, tamaño pequeño y similitudes en secuencia. Las biomoléculas comprenden aproximadamente el 0,01 por ciento del contenido total de ARN y las copias individuales de ARNm por rango celular pueden variar ampliamente. Adicionalmente, Los miARN de una familia pueden diferir en un solo nucleótido, mientras que los miARN específicos pueden regularse durante la enfermedad y la función celular regular. Como resultado, Las estrategias de detección de miARN requieren una alta especificidad y la capacidad de identificar correctamente algunas moléculas de una muestra abundante con moléculas de ARN predominantes.
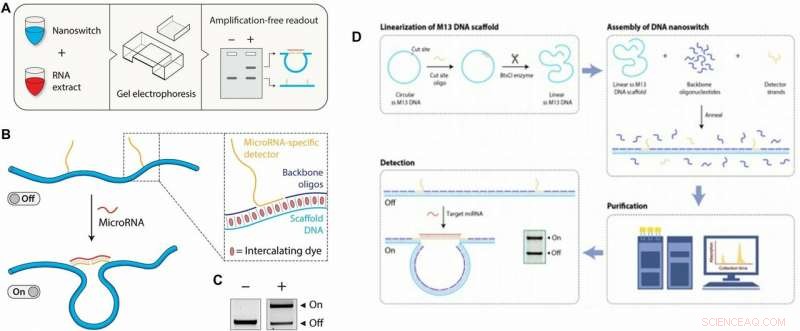
Izquierda:Concepto y flujo de trabajo del ensayo miRacles. (A) Flujo de trabajo del ensayo miRacles:los nanointerruptores de ADN personalizados se mezclan con la muestra de miARN objetivo, incubado y se procesan en un gel de agarosa para su detección. (B) Los nanointerruptores de ADN experimentan un cambio conformacional de un estado lineal "apagado" a un estado "encendido" en bucle cuando se unen a un miARN objetivo. Recuadro:el nanointerruptor está compuesto por un andamio M13 de una sola hebra, oligonucleótidos de la columna vertebral, y extensiones monocatenarias (detectores) complementarias al miARN diana. Los tintes intercalados intrínsecos al proceso de electroforesis proporcionan la señal para visualizar los nanointerruptores. (C) Las dos conformaciones se pueden resolver en un gel de agarosa estándar. Derecha:examen en profundidad del proceso de construcción del nanointerruptor de ADN. (D) Linealización:la preparación de un armazón de M13 monocatenario lineal procede por hibridación de un oligonucleótido para permitir la escisión en el sitio bicatenario por una enzima de restricción (BtsCI en este caso). Ensamblaje:el ensamblaje del nanointerruptor de ADN se realiza mezclando un exceso molar 10x de oligonucleótidos de la cadena principal y hebras detectoras (específicas para el objetivo) al M13 monocatenario y aplicando una rampa de enfriamiento de 90 ° C a 25 ° C a 1 ° C / min. . Purificación:para eliminar el exceso de oligonucleótidos, se utiliza un método de cromatografía líquida. Detección:para utilizar los nanointerruptores, se mezclan con una solución que contiene objetivos de microARN y se procesan en un gel para resolver los estados de encendido y apagado, como se muestra en el recuadro. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443
Los métodos tradicionales para la detección de miARN incluyen transferencia Northern, reacción cuantitativa en cadena de la polimerasa con transcripción inversa (qRT-PCR), secuenciación de próxima generación e hibridación basada en microarrays para separar la señal de miARN del ruido. De los métodos especificados, La transferencia Northern puede identificar directamente miARN nativos, mientras que otros se basan en métodos de etiquetado adicionales o amplificación escalonada, agregando al costo, complejidad y rendimiento de la detección. Por ejemplo, Se pueden utilizar nanoestructuras de ADN innovadoras para la detección de miARN, donde varios grupos de investigación han combinado previamente nanoestructuras con nanopartículas, reacciones en cadena de hibridación y nanohojas de dicalcogenuro de metales de transición para permitir el proceso.
En el presente trabajo, Chandrasekaran y col. desarrolló un dispositivo basado en ADN relativamente simple para resolver un desafío biomédico complejo. En el ensayo de miRacles, Los científicos utilizaron un "reactivo inteligente" compuesto por nanointerruptores de ADN diseñados racionalmente para la detección de miARN nativo simple y rentable sin utilizar equipos especializados en el laboratorio. Los nanointerruptores de ADN se diseñaron originalmente como herramientas para experimentos biofísicos de una sola molécula y luego se reconocieron por su capacidad para detectar y cuantificar interacciones biomoleculares mediante electroforesis en gel. Los esfuerzos de investigación colaborativa anteriores del mismo equipo de investigación se centraron en la detección molecular para cuantificar los niveles de proteínas y detectar secuencias de ADN sintético como prueba de concepto.
El presente trabajo amplió los estudios y conceptos preliminares para producir detección y cuantificación de miARN multiplexados listos para el usuario. Los científicos analizaron nanogramos de extractos de ARN celular en un período de tiempo corto utilizando una configuración experimental construida con suministros de laboratorio comunes. Diseñaron el nanoconmutador de ADN como un dúplex lineal que formaba un bucle en presencia de la molécula de miARN objetivo. Para construir el nanointerruptor, Chandrasekaran y col. utilizó el enfoque de origami de ADN mediante la hibridación de oligonucleótidos cortos complementarios a un andamio de ADN monocatenario.
Diseñaron dos hebras "detectoras" distantes con voladizos complementarios a diferentes segmentos del miARN objetivo. Cuando el miARN se reconoce y se une a la construcción, el interruptor reconfigurado desde el estado lineal "apagado" al estado en bucle "encendido". Cuantificaron los dos estados usando electroforesis en gel de agarosa estándar para detectar la señal que surge del nanointerruptor en bucle. La señal solo fue amplificada por un solo miARN de interés, los resultados se compararon favorablemente con la técnica de transferencia de energía por resonancia de fluorescencia (FRET).

Validación del ensayo miRacles. (A) Especificidad de los nanointerruptores de ADN con detectores diseñados para let-7b. Un desajuste tan bajo como 1 nt entre los detectores y el miARN objetivo elimina la señal. (B) Límite de detección del ensayo. CAROLINA DEL NORTE, control negativo. AU, unidades arbitrarias. (C) Transcurso del tiempo del ensayo para un objetivo de baja concentración. (D) Rango dinámico del ensayo en diferentes tiempos de reacción. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443.
Para validar el concepto, el equipo de investigadores eligió un miARN objetivo let-7b debido a su familia altamente conservada de más de una docena de miARN relacionados que variaban en uno o más nucleótidos. Estos miARN eran adecuados debido a su papel fundamental en las funciones biológicas y la desregulación en múltiples enfermedades humanas. Para eliminar la interferencia y el ruido entre el nanointerruptor y el objetivo, lo que provocó una reducción de la intensidad de la señal en comparación con una combinación perfecta, los científicos rediseñaron racionalmente los nanointerruptores. Para lograr una especificidad perfecta, desestabilizaron la interacción en el lado que contenía el desajuste. Los resultados del estudio ilustraron una alta especificidad del ensayo así desarrollado, proporcionando una respuesta a un desafío clave en la detección de miARN que culminó con una alta relación señal / ruido.
La baja abundancia de miARN también requirió una alta sensibilidad de detección, que los científicos lograron optimizando el protocolo. Luego realizaron experimentos similares para otras dos variantes de miARN (miR-15 ay miR-206), resultando en niveles de detección de subatomol a un solo attomole. Por ejemplo, para una concentración baja de una muestra objetivo (6 pM), la señal aumentó durante 4 horas, con pocos cambios más allá de ese plazo.
Detección de miARN a partir de la diferenciación de células de mioblastos. (A) Esquema que muestra células de mioblastos, recolectados mientras crecen en GM y en los días de diferenciación 1 y 4, procesado para producir fracciones de ARN total y pequeñas. Un marcador temprano de diferenciación miogénica, Myog, y un marcador de diferenciación miogénica tardía, MHC, se midieron mediante (B) transferencia Western y (C) mediante inmunocitoquímica para confirmar la diferenciación. Tanto Myog como MHC fueron regulados positivamente en DM1 y DM4. GAPDH (gliceraldehído-3-fosfato deshidrogenasa) sirvió como control en (B). (D) Chandrasekaran y col. detectaron miR-206 en las muestras diferenciadas con 50 ng de ARN pequeños y (E) con 500 ng de ARN totales. La cuantificación de las intensidades del gel muestra una fuerte regulación progresiva durante la diferenciación, similar en muestras de (F) ARN pequeño y (G) ARN total. A partir de muestras DM4, observamos la detección de tan solo (H) 200 pg de ARN pequeños y (I) 500 pg de ARN totales. Crédito:Science Advances, doi:10.1126 / sciadv.aau9443. 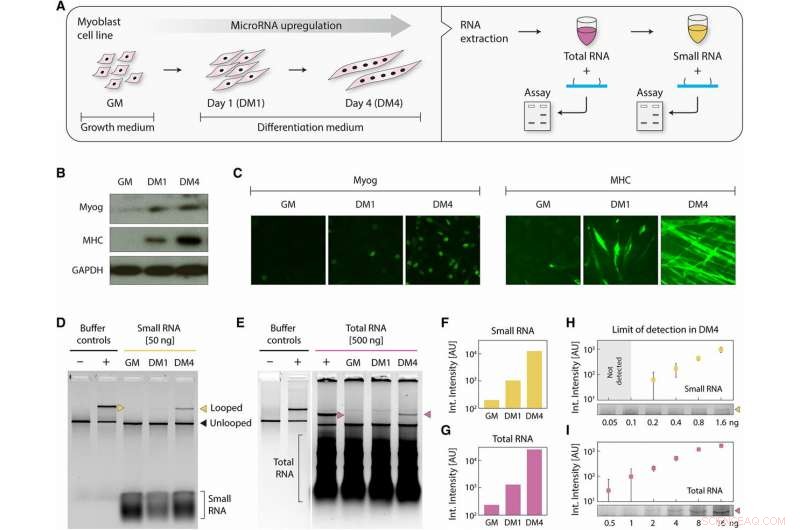
Dado que múltiples ARNm también pueden alterar su expresión durante diferentes etapas celulares o de la enfermedad, el fenómeno requirió capacidades adicionales de detección. Para lograr esto en la configuración experimental, Chandrasekaran y col. utilizó la capacidad de programación de los nanointerruptores y desarrolló un sistema de multiplexación capaz de detectar múltiples miARN de la misma muestra. Colocaron las hebras del detector en las ubicaciones deseadas del andamio de ADN, dando como resultado bucles de diferentes tamaños cuando se unen al miARN objetivo. El tamaño del bucle del nanointerruptor determinó así la migración del gel, resultando en una banda única en el gel para una detección precisa. Para el experimento, los científicos eligieron cuatro miARN presentes en las células musculares; miR-206, miR-125b, miR-24 y miR-133-3p y un miARN de control negativo; miR-39 específico de la especie Caenorhabditis elegans.
Dentro de 50 ng de ARN pequeños, los científicos detectaron los cuatro miARN en varios niveles de expresión, mientras se confirma la no detección con el control negativo. La estrategia de multiplexación permitió a los científicos comparar directamente los niveles de miARN en una sola muestra, sin etiquetado ni amplificación. En total, el trabajo proporcionó un paso adicional en la dirección de expandir el rendimiento del ensayo miRacles. La capacidad también se puede ampliar para acomodar más miARN por conmutador.
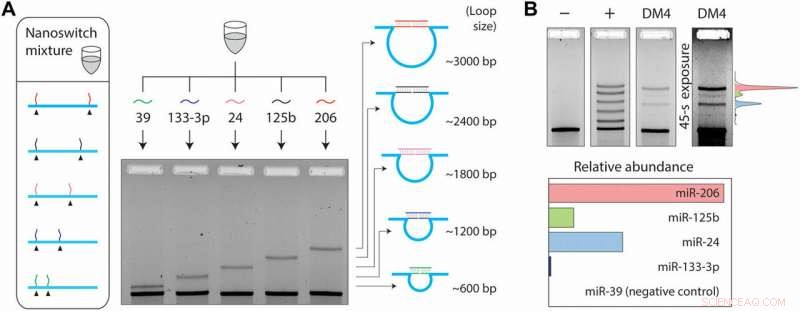
Configuración de multiplexación de cinco canales. (A) La multiplexación permite la detección de diferentes miARN con diferentes tamaños de bucle. (B) Una mezcla de nanointerruptores multiplexados muestra cinco bandas con intensidad similar en un control positivo que consta de los cinco miARN objetivo. En 50 ng de ARN pequeños de DM4, Se detectan cuatro miARN diferentes en varios niveles de expresión, con miR-39 (un miARN específico de C. elegans) no detectado.
De este modo, Chandrasekaran y col. avanzó sustancialmente desde su detección preliminar de prueba de concepto de secuencias de ADN sintético; para establecer, caracterizar y optimizar un ensayo de detección de miARN listo para usar con extractos biológicos. El trabajo demostrado fue un primer ejemplo del uso de nanointerruptores de ADN para detectar miARN a partir de una muestra biológica verdadera. Si bien el rendimiento del ensayo de miARN fue competitivo en comparación con otras técnicas de uso común, la selectividad de 1 nucleótido observada en el presente trabajo fue difícil de lograr con los métodos existentes. La sensibilidad de miRacles también superó la transferencia Northern y los microarrays. El ensayo podría medir miARN sin necesidad de amplificación, con protocolos más simples y sin el error añadido del procesamiento de muestras adicional. El protocolo simplemente mezcló los nanointerruptores con la muestra de líquido para electroforesis en gel, para producir resultados de alta calidad en el laboratorio. El trabajo de investigación es potencialmente transferible de muestras biológicas a clínicas para diagnosticar y monitorear enfermedades.
Más importante, el presente trabajo se alinea con el concepto más amplio de ciencia frugal; una visión prometedora de ciencia rentable que ya ha producido soluciones de bajo costo para técnicas de centrifugación de sangre y purificación de agua en ingeniería biomédica. Chandrasekaran y col. aspirar a seguir contribuyendo a la tendencia emergente de la ciencia, interrumpiendo la relación existente entre costo y rendimiento para proporcionar acceso a gran escala a métodos simples de detección de miARN con reactivos inteligentes de mezcla y lectura.
© 2019 Science X Network