En el ámbito en constante evolución de la microscopía, los últimos años han sido testigos de avances notables tanto en hardware como en algoritmos, impulsando nuestra capacidad de explorar las maravillas infinitesimales de la vida. Sin embargo, el camino hacia la microscopía de iluminación estructurada tridimensional (3DSIM) se ha visto obstaculizado por los desafíos que surgen de la velocidad y la complejidad de la modulación de polarización.
Ingrese al sistema 3DSIM de modulación de alta velocidad "DMD-3DSIM", que combina una pantalla digital con imágenes de súper resolución, lo que permite a los científicos ver las estructuras celulares con un detalle sin precedentes.
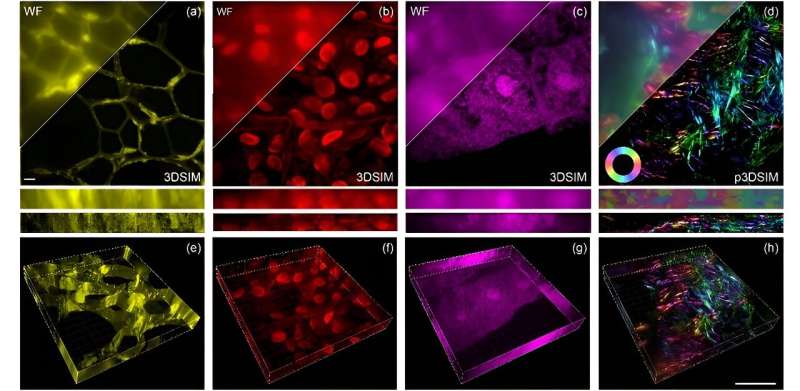
Como se informa en Advanced Photonics Nexus , el equipo del profesor Peng Xi de la Universidad de Pekín desarrolló esta innovadora configuración en torno a un dispositivo de microespejo digital (DMD) y un modulador electroóptico (EOM). Aborda los desafíos de resolución mejorando significativamente la resolución lateral (de lado a lado) y axial (de arriba a abajo), para obtener una resolución espacial 3D que, según se informa, es el doble de la lograda por las técnicas tradicionales de imágenes de campo amplio.
En términos prácticos, esto significa que DMD-3DSIM puede capturar detalles intrincados de estructuras subcelulares, como el complejo de poros nucleares, microtúbulos, filamentos de actina y mitocondrias en células animales. La aplicación del sistema se amplió para estudiar ultraestructuras de células vegetales altamente dispersas, como las paredes celulares de las hojas de adelfa y las estructuras huecas de las hojas de algas negras. Incluso en un corte de riñón de ratón, el sistema reveló un efecto de polarización pronunciado en los filamentos de actina.
Una puerta abierta al descubrimiento
Lo que hace que DMD-3DSIM sea aún más interesante es su compromiso con la ciencia abierta. El equipo de Xi ha hecho que todos los componentes de hardware y mecanismos de control estén disponibles abiertamente en GitHub, fomentando la colaboración y alentando a la comunidad científica a desarrollar esta tecnología.
La técnica DMD-3DSIM no sólo facilita importantes descubrimientos biológicos sino que también sienta las bases para la próxima generación de 3DSIM. En aplicaciones que involucran imágenes de células vivas, los avances en tintes más brillantes y fotoestables, algoritmos de eliminación de ruido y modelos de aprendizaje profundo basados en redes neuronales prometen mejorar la duración de las imágenes, la recuperación de información y la restauración en tiempo real de imágenes 3DSIM a partir de datos ruidosos. Al combinar la apertura de hardware y software, los investigadores esperan allanar el camino para el futuro de las imágenes multidimensionales.
Más información: Yaning Li et al, Microscopía de iluminación estructurada tridimensional con modulación de sincronización de autopolarización de alta velocidad, Advanced Photonics Nexus (2023). DOI:10.1117/1.APN.3.1.016001
Proporcionado por SPIE