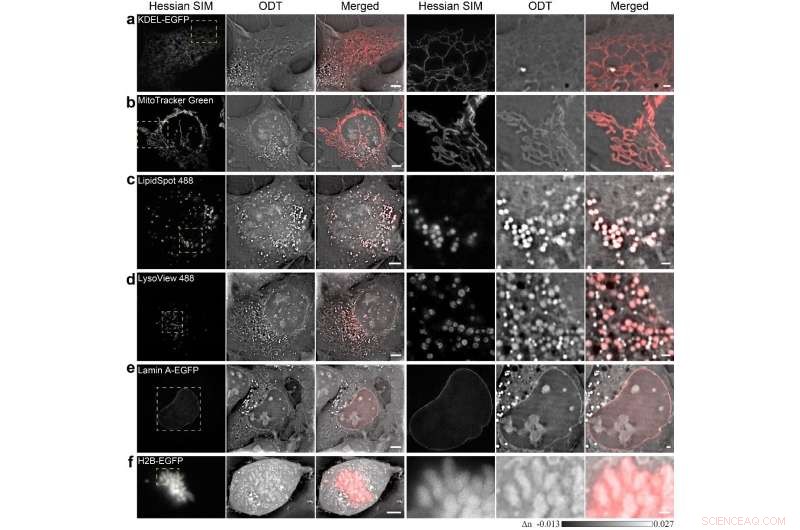
Las regiones encerradas por los cuadros amarillos discontinuos a la izquierda se amplían a la derecha. Barras de escala, 5 μm (izquierda) y 1 μm (derecha). Crédito:por Dashan Dong, Xiaoshuai Huang, Liuju Li, Heng Mao, Yanquan Mo, Guangyi Zhang, Zhe Zhang, Jiayu Shen, Wei Liu, Zeming Wu, Guanghui Liu, Yanmei Liu, Hong Yang, Qihuang Gong, Kebin Shi, Liangyi Chen
La aparición de la microscopía de fluorescencia de superresolución (SR) ha rejuvenecido la búsqueda de nuevas subestructuras celulares e intermedios dinámicos. Sin embargo, limitado por el amplio espectro de emisión de fluoróforos y una fototoxicidad excesiva, La microscopía de fluorescencia SR solo se puede usar para resaltar un puñado de biomoléculas simultáneamente y es incapaz de proporcionar un mapa holístico del entorno celular y el paisaje.
En un nuevo artículo publicado en Ciencias de la luz y aplicaciones , científicos del State Key Laboratory for Mesoscopic Physics and Frontiers Science Center for Nano-optoelectronics, Universidad de Peking, Porcelana, Laboratorio estatal clave de biología de membranas, Laboratorio clave de Beijing de medicina molecular cardiometabólica, Instituto de Medicina Molecular, Universidad de Peking, China y sus colaboradores desarrollaron tomografía computacional de difracción asistida por fluorescencia SR (SR-FACT), que combina la tomografía de difracción óptica (ODT) tridimensional sin etiquetas con microscopía de iluminación estructurada de Hesse de fluorescencia bidimensional. El módulo ODT es capaz de resolver mitocondrias, gotitas de lípidos, la membrana nuclear, cromosomas, el retículo endoplásmico tubular y los lisosomas. El uso de imágenes de células vivas correlacionadas en modo dual durante un período de tiempo prolongado, observaron estructuras subcelulares novedosas llamadas cuerpos de vacuolas oscuras (DB), la mayoría de los cuales se originan en regiones perinucleares densamente pobladas, e interactuar intensamente con orgánulos como las mitocondrias y la membrana nuclear antes de colapsar finalmente en la membrana plasmática. Este trabajo demuestra las capacidades únicas de SR-FACT, lo que sugiere su amplia aplicabilidad en biología celular en general.
SR-FACT visualiza tanto el paisaje celular como la identidad molecular de las células vivas. Se desarrolló un nuevo algoritmo denominado algoritmo de búsqueda iterativa vectorial (VISA) para minimizar los errores de reconstrucción de imágenes tridimensionales en un esquema de exploración tomográfica de alta velocidad y frecuencia de kHz. Como resultado, SR-FACT puede utilizar simultáneamente una velocidad de imagen máxima para capturar la dinámica en células vivas y mantener un flujo de fotones suficiente para una sensibilidad máxima. En el sistema SR-FACT, el módulo ODT logró una resolución lateral de ~ 200 nm a una velocidad de imagen volumétrica de 0,8 Hz (40 × 40 × 20 m 3 ). 2-D-SIM de arpillera, que permite la obtención de imágenes SR a una fracción de la dosis de fotones utilizada por SIM convencional, se utilizó para guiar la interpretación de las estructuras observadas por el módulo ODT. Al realizar imágenes correlacionadas de modo dual en células COS-7, resolvieron seis orgánulos conocidos sin etiquetar:el retículo endoplásmico tubular (RE), mitocondrias, endosomas / lisosomas tardíos (LE / LY), LD, la membrana nuclear y los cromosomas. Todos estos datos destacan la ventaja única de SR-FACT en el estudio del interactoma orgánulo. Es más, también observaron estructuras vacuoladas con pH neutro que contenían principalmente líquido en la luz. Las imágenes SR de células vivas transcurridas durante una hora en combinación con el análisis cuantitativo revelan las rutas de tráfico no convencionales y los roles indispensables de las vacuolas en la organización del interactoma del orgánulo. todo lo cual sugiere que representan orgánulos que antes no se apreciaban.