
Una captura de pantalla del sitio web DIVE. Crédito:Gupta et al.
Los artículos académicos a menudo contienen relatos de nuevos avances y teorías interesantes relacionadas con una variedad de campos. Sin embargo, la mayoría de estos artículos están escritos usando jerga y lenguaje técnico que solo pueden ser entendidos por lectores familiarizados con esa área de estudio en particular.
Por lo tanto, los lectores no expertos suelen ser incapaces de comprender los artículos científicos, a menos que sean seleccionados y hechos más accesibles por terceros que comprendan los conceptos e ideas contenidos en ellos. Teniendo esto en cuenta, un equipo de investigadores del Centro de Computación Avanzada de Texas en la Universidad de Texas en Austin (TACC), La Universidad Estatal de Oregón (OSU) y la Sociedad Estadounidense de Biólogos Vegetales (ASPB) se han propuesto desarrollar una herramienta que pueda extraer automáticamente frases y terminología importantes de los trabajos de investigación para proporcionar definiciones útiles y mejorar su legibilidad.
"Nuestro proyecto está motivado por la necesidad de mejorar la legibilidad de los artículos de revistas, "Weijia Xu, que lideran el equipo de TACC, dijo a TechXplore. "Es un esfuerzo conjunto entre curadores biológicos, editores de revistas e informáticos con el objetivo de desarrollar un servicio web que pueda reconocer y permitir la conservación por parte del autor de la terminología importante utilizada en las publicaciones de las revistas. La terminología y las palabras se adjuntan al final del artículo de la revista para aumentar su accesibilidad para los lectores ".
Xu y sus colegas desarrollaron un marco extensible que se puede utilizar para extraer información de documentos. Luego implementaron este marco dentro de un servicio web llamado DIVE (Extracción de vocabulario de información de dominio), integrándolo con el canal de publicación de revistas de la ASPB. A diferencia de las herramientas existentes para extraer información de dominio, su marco combina varios enfoques, incluida la extracción guiada por ontología, extracción basada en reglas, procesamiento del lenguaje natural (PNL) y técnicas de aprendizaje profundo.

El panorama de la arquitectura del sistema propuesto por los investigadores. Crédito:Gupta et al.
"Los resultados obtenidos por diferentes modelos se almacenan en una base de datos centralizada, "Explicó Xu." También diseñamos un servicio web que permite a los usuarios seleccionar los resultados de la extracción. El servicio web está integrado con la canalización de publicaciones de producción en ASPB ".
Una vez que se envía la versión de vista previa de un artículo de revista y se ingresa a la canalización de la ASPB, el manuscrito se envía automáticamente a DIVE, que lo procesa y produce una URL con la que el autor podrá acceder a los resultados del procesamiento de DIVE. Se solicita al autor del artículo que visite el enlace proporcionado y revise la información extraída antes de poder enviar oficialmente el artículo.
"El autor debe visitar el sitio DIVE para revisar los resultados de la extracción y hacer la aprobación final de la lista de información que se incluirá al final de su artículo, ", Dijo Xu." DIVE también rastrea las correcciones del autor para mejorar las futuras tareas de extracción. En la actualidad, ningún otro editor de revistas ha adoptado un enfoque similar ni lo ha integrado con su canal de publicación ".
Durante sus análisis y al extraer datos clave de documentos, el marco desarrollado por los investigadores utiliza varias técnicas. Esto le permite capturar más información que otros métodos, como ABNER (un reconocedor de entidades con nombre biomédico), que es una herramienta de software de código abierto para la minería de textos de biología molecular que solo puede extraer términos generales (por ejemplo, genes y proteínas). Al contrario de DIVE, ABNER solo se basa en campos aleatorios condicionales (CRF), un método de modelado estadístico que se usa comúnmente en aplicaciones de reconocimiento de patrones y aprendizaje automático.
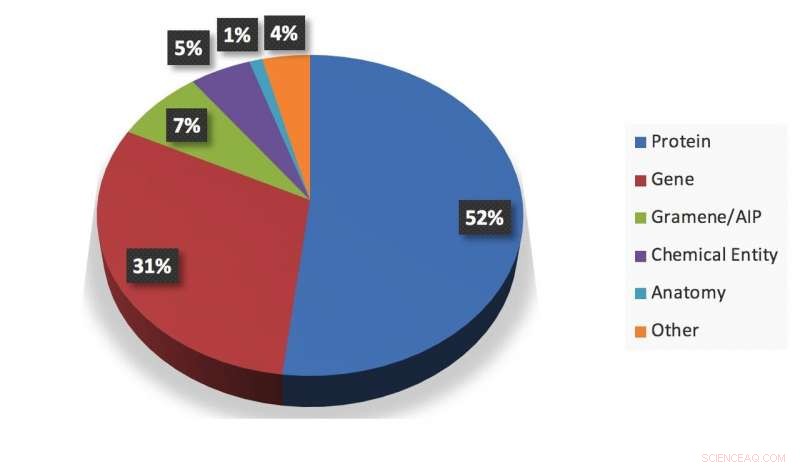
Un resumen visual de una instantánea de la información extraída por el sistema. Crédito:Gupta et al.
"Una de las principales contribuciones de nuestro proyecto es que ayuda a crear conjuntos de datos y modelos que pueden inferir los intereses de investigación de los autores a partir de sus publicaciones. ", Dijo Xu." Nuestro proyecto puede beneficiar a comunidades más amplias de investigadores biológicos. Para los autores, las extracciones y la inclusión de la información clave pueden aumentar la accesibilidad de sus artículos ".
Xu y su colega Amit Gupta evaluaron su marco y compararon su desempeño con el de otras herramientas de extracción de información. incluido ABNER. Sus hallazgos revelaron que el uso de múltiples enfoques, incluido el aprendizaje profundo, DIVE alcanza puntuaciones de mayor precisión que otros modelos previamente entrenados basados únicamente en CRF. Curiosamente, el marco DIVE también se puede actualizar continuamente, ya que se pueden agregar modelos de extracción adicionales en cualquier momento.
La aplicación web DIVE no solo permite a los lectores no expertos comprender mejor los artículos académicos, también puede ayudarles a identificar artículos que se ajusten a sus intereses. Investigadores por otra parte, puede utilizar DIVE para mantenerse informado sobre áreas de investigación particulares, así como conocer nueva terminología y tendencias relacionadas con su campo de interés. Finalmente, la información generada por la aplicación también puede orientar a los curadores de biología en sus decisiones y procesos de recolección de datos.
"Continuamos con nuestro proyecto explorando dos direcciones, "Dijo Xu." Por un lado, Estamos investigando métodos novedosos para incorporar con nuestros modelos de extracción de información para mejorar el rendimiento. Por otra parte, también estamos tratando de expandir nuestro servicio ofreciéndolo a comunidades de usuarios adicionales y editores de revistas ".
© 2019 Science X Network