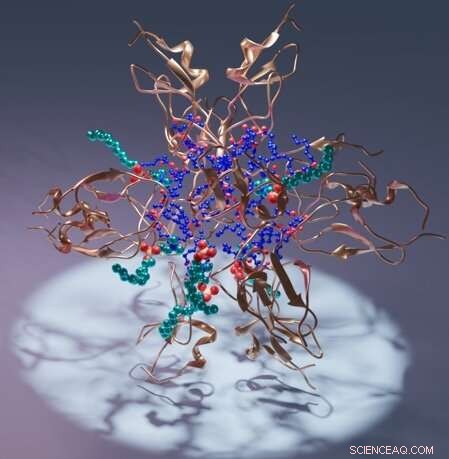
Representación artística de un dominio C1 de proteína quinasa C (cobre), su ligando diacilglicerol (azul) y detergente (cian). Crédito:Sachin Katti.
En un gran avance para el diseño racional de medicamentos, un equipo de Texas A&M AgriLife ha descrito varias estructuras de proteínas de un actor crucial en los procesos celulares. El avance podría traer nuevas ideas para tratamientos de enfermedades como el Alzheimer, el SIDA, el cáncer y otras.
En concreto, el trabajo describe el dominio C1 de la proteína quinasa C, PKC, que ayuda a regular la actividad de la proteína en los organismos. En las estructuras, el dominio C1 envuelve diferentes moléculas de intenso interés terapéutico, proporcionando la primera guía confiable de resolución atómica para diseñar candidatos a fármacos.
Publicado el 16 de mayo en Nature Communications , la investigación fue dirigida por Tatyana Igumenova, Ph.D., profesora asociada en el Departamento de Bioquímica y Biofísica de la Facultad de Agricultura y Ciencias de la Vida de Texas A&M. El autor principal del proyecto es Sachin Katti, Ph.D., becario postdoctoral que trabaja con Igumenova.
El estudio involucró una colaboración con Inna Krieger, Ph.D., profesora asistente de investigación, y James Sacchettini, Ph.D., profesor, ambos en el Departamento de Bioquímica y Biofísica.
Una de las estructuras proteicas más buscadas
Una célula sana responde a las señales químicas de formas precisas e intrincadas. Recibir entradas químicas del entorno de la célula y enviarlas a los sistemas de control central dentro del núcleo celular es tarea de proteínas especializadas como la PKC.
La actividad incorrecta de PKC aparece en muchas enfermedades humanas. Como resultado, hay mucho interés en encontrar formas de ajustar la actividad de la PKC con fármacos. El diseño de dichos medicamentos ofrecerá nuevos enfoques para tratar la enfermedad de Alzheimer, el SIDA, el cáncer y más.
"La proteína quinasa C es una de las proteínas más intensamente estudiadas en biología celular y farmacología", dijo Igumenova. "Un obstáculo importante ha sido la falta de información estructural precisa para guiar los esfuerzos de diseño de fármacos".
Una complicación para el diseño de fármacos es que la familia PKC tiene 11 miembros. Diferentes miembros de la familia PKC pueden tener efectos fisiológicos opuestos, por lo que un candidato a fármaco exitoso debe ser selectivo sobre a qué PKC se dirige.
Para hacer eso, los candidatos a fármacos deben encajar en una PKC objetivo como una llave para una cerradura. Pero determinar la estructura 3D de un "interruptor de encendido" de PKC, el dominio C1, unido a los activadores de PKC no ha sido fácil.
Las estructuras de proteínas generalmente se resuelven mediante cristalografía de rayos X. La técnica implica el uso de rayos X para determinar la posición de los átomos en un cristal. Para este método, los investigadores deben crear condiciones en las que cristalice la proteína de interés. Sin embargo, los intensos esfuerzos en muchos laboratorios de investigación durante las últimas tres décadas no lograron producir cristales de dominios C1 unidos a ligandos relevantes. Debido a esta falta de progreso, varios investigadores declararon que la tarea era imposible, dijo Igumenova.

Los cristales de un dominio de proteína quinasa C se formaron espontáneamente en el tubo de muestra de RMN de Katti. Credit:Sachin Katti.
Solving a 30-year problem
Accepting the problem as challenging, Katti and Igumenova decided instead to study the molecules in solution using nuclear magnetic resonance, NMR, spectroscopy. This involved finding the right components to mimic cell membranes, where the C1 domain would encounter ligands.
"Then, one fine day, Sachin discovered crystals forming in an old NMR tube," Igumenova said. "I give all the credit to Sachin, who basically said, 'I'm going to go and test them and see if they are actually the protein.' And he was right. It gave us confidence that crystallization is possible."
In turn, Katti gives credence to the insights obtained from NMR, and a bit of luck.
"I think that's the beauty of doing research where you have to use multiple approaches," he said. "You never know when one approach is going to be useful for doing something with other approaches."
Insights from NMR and X-ray crystallography
The new protein structures, along with the team's NMR results, have already yielded interesting information. One long-standing mystery has been how C1 domains can accommodate ligands that have very different chemical structures, Igumenova said.
"Our previous NMR work indicated that the loops of the C1 domain that bind ligands are very dynamic," Igumenova said. "This C1 domain is like a PAC-man. It binds the membrane, and then it searches for a ligand. Once it finds the ligand, it latches on."
In addition, the structure shows that the ligand-binding groove has a "water-loving," or hydrophilic, surface at the bottom, and "water-repelling," or hydrophobic, surface at the top.
"If you think about a lipid molecule, the head group is hydrophilic and the tail is hydrophobic," Igumenova said. "So, when C1 domains bind lipid ligands, the patterns match."
The team's results include the structure of a C1 domain bound to its natural ligand, diacylglycerol. In addition, the team describes several other structures of C1 that include different compounds of pharmacological interest.
The work also provides a method for testing different drug candidates, Katti said.
"If you want to study fish, you want to study them in water," Katti said. "Now we know how to create a membrane-like environment where these very hydrophobic compounds can be tested for C1 binding."
Next, Katti and Igumenova plan to explore C1 domains from other PKC family members.
"It's important for us to focus on C1 domains because they have inherent differences that can be exploited to achieve selectivity," Igumenova said. "What we are finding now is that not all C1 domains are created equal." Los hallazgos revelan un nuevo mecanismo de activación de ALK