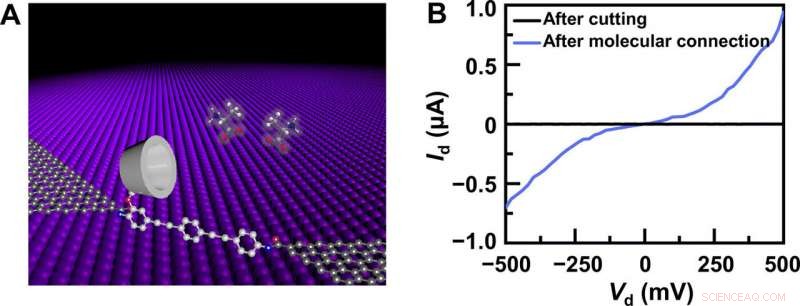
Estructura del dispositivo y caracterización eléctrica. (A) Representación esquemática de un GMG-SMJ basado en PM-β-CD. La máquina molecular que presenta un PM-β-CD se conectó covalentemente con contactos puntuales de grafeno a través de enlaces amida. (B) Curvas I-V de GMG-SMJ después del corte con plasma de oxígeno y después de una conexión molecular adicional. Crédito: Avances de la ciencia , doi:10.1126 / sciadv.abe4365
La química analítica tiene como objetivo discriminar de manera eficiente entre dos aminoácidos. En un nuevo informe en Avances de la ciencia , Zihao Liu y un equipo de investigación en Química, Física, y ciencia de materiales en China y Japón, utilizó un método eléctrico de una sola molécula basado en nanocircuitos moleculares para formar uniones estables de grafeno-molécula-grafeno de una sola molécula. El equipo desarrolló estas uniones moleculares mediante la unión covalente de una máquina molecular. Luego utilizaron el pH para variar el tipo y la carga de los aminoácidos para encontrar distintas fluctuaciones de corriente multimodales que se originan en diversas interacciones entre el huésped y el huésped en relación con los cálculos teóricos. Los datos de conductancia produjeron tiempos de nivelación característicos y velocidades de transferencia para cada aminoácido para permitir mediciones in situ precisas y en tiempo real. Los científicos probaron cuatro aminoácidos y sus enantiómeros (moléculas quirales que son imágenes especulares entre sí) para mostrar la capacidad de distinguir entre ellos en unos pocos microsegundos para proporcionar un método simple y preciso para la identificación de aminoácidos, así como proteína de una sola molécula. secuenciación.
Bloques de construcción de proteínas
Los aminoácidos forman componentes básicos de proteínas, moléculas con funciones fisiológicas especiales y elementos estructurales clave en productos farmacéuticos con aplicaciones en las ciencias de la vida. Los investigadores pueden reconocer enantiómeros de aminoácidos para proporcionar información importante relativa al reconocimiento quiral y las funciones fisiológicas en sistemas biológicos. Detectando la estructura, La pureza enantiomérica y el comportamiento dinámico de los aminoácidos pueden promover la producción de nuevas técnicas para la secuenciación de proteínas y las investigaciones farmacéuticas. La detección e identificación de aminoácidos con diferentes estructuras y quiralidad son de importancia crítica en la proteómica, productos farmacéuticos y nanobiotecnología. Sin embargo, Las máquinas moleculares existentes solo funcionan comparando los parámetros termodinámicos del conjunto, que es insuficiente. Por tanto, es importante desarrollar una técnica molecular general para revolucionar los métodos existentes y reconocer las moléculas diana con alta precisión. En este trabajo, Liu y col. demostraron una técnica de molécula única para identificar directamente diferentes aminoácidos proteinogénicos y sus enantiómeros a través de mediciones dinámicas precisas de interacciones huésped-huésped basadas en una técnica de molécula única conocida como uniones de molécula única de grafeno-molécula-grafeno abreviadas como GMG-SMJ para covalentemente incorporan sistemas moleculares individuales que se comportan como el canal conductor en un nanocircuito eléctrico.
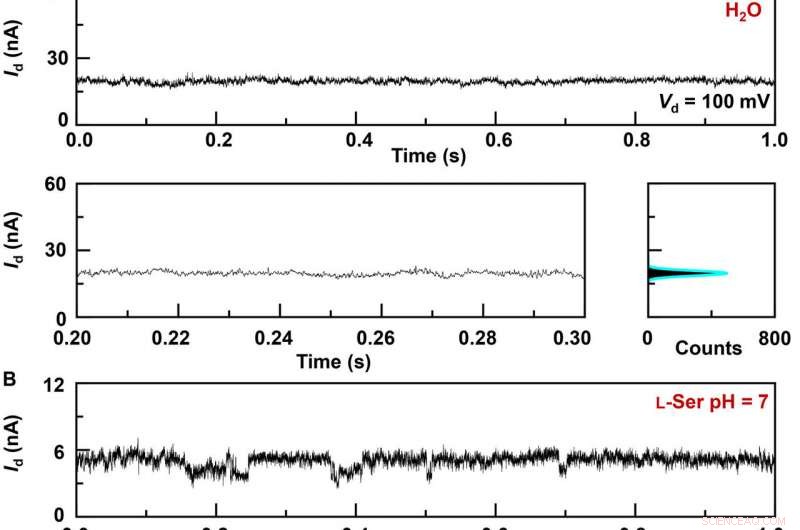
Grabaciones actuales en tiempo real de la dinámica de interacción anfitrión-huésped en GMG-SMJ. Curvas I-t de diferentes GMG-SMJ (arriba), las partes agrandadas (abajo a la izquierda), y sus correspondientes histogramas (abajo a la derecha):(A) Un dispositivo de trabajo en agua pura, (B) el mismo dispositivo en una solución acuosa de L-Ser 100 nM, y (C) otro dispositivo en una solución acuosa de L-Ala 100 nM. Los insertos en los histogramas son las partes agrandadas de los picos pequeños. pH =7, T =298 K, y el voltaje de polarización =0,1 V. Crédito:Science Advances, doi:10.1126 / sciadv.abe4365
El nuevo enfoque proporcionó una plataforma robusta para formar componentes electrónicos de una sola molécula para crear dispositivos optoelectrónicos moleculares. Para desarrollar los constructos, Liu y col. intercalado covalentemente una máquina molecular entre un par de contactos puntuales de grafeno con nanopartículas para la detección de aminoácidos y el reconocimiento de quiralidad. Los resultados abrirán nuevas rutas para desarrollar la nanotecnología de secuenciación precisa de proteínas de una sola molécula para aplicaciones prácticas.
Desarrollo de dispositivos, caracterización eléctrica y mediciones en tiempo real
El equipo de investigación sintetizó grafeno de capa única mediante deposición de vapor químico sobre láminas de cobre y lo transfirió a obleas de dióxido de silicio / silicio y modeló los electrodos metálicos mediante fotolitografía. Para desarrollar los electrodos de contacto puntuales de grafeno con nanopartículas, utilizaron un método litográfico de líneas discontinuas. Luego construyeron GMG-SMJ (uniones de una sola molécula de grafeno-molécula-grafeno) conectando covalentemente una sola máquina molecular con electrodos de grafeno a través de enlaces amida. El equipo midió las curvas de corriente-voltaje de los dispositivos en una variedad de etapas para identificar la formación de GMG-SMJ. El transporte de carga a través de la unión resultó de conexiones de una sola molécula. Luego realizaron caracterizaciones eléctricas dependientes del tiempo para monitorear la conductancia de las uniones moleculares simples (SMJ) permetiladas-β-ciclodextrina (PM-β-CD) en tiempo real. Primero midieron las construcciones en agua y luego en diversas soluciones de diferentes aminoácidos. Las trayectorias representativas en tiempo actual revelaron distribuciones de picos múltiples para diferentes aminoácidos en solución, como L-serina y L-alanina. Los múltiples estados distintos de cada aminoácido solo se originaron a partir del proceso de asociación / disociación entre el centro funcional PM-β-CD y los aminoácidos circundantes.
Análisis computacionales de SMJ de L-Ala @ PM-β-CD. (A) Diagrama esquemático de los orbitales moleculares de frontera calculados de la máquina molecular. (B a D) Configuraciones moleculares típicas durante la asociación con aminoácidos con carga diferente:(B) catión, (C) zwiterión, y (D) anión. (E) Espectros de transmisión correspondientes a un voltaje de polarización cero. El rojo, amarillo, y las curvas verdes muestran la transmisión cuando el anión, zwitterion, y formas catiónicas estaban en la cavidad, respectivamente. La curva azul muestra la cavidad vacía. Los espectros completos se proporcionan en la fig. S4. (F a H) Representación esquemática de aminoácidos en catión, zwitterion, y formas aniónicas. Crédito:Science Advances, doi:10.1126 / sciadv.abe4365 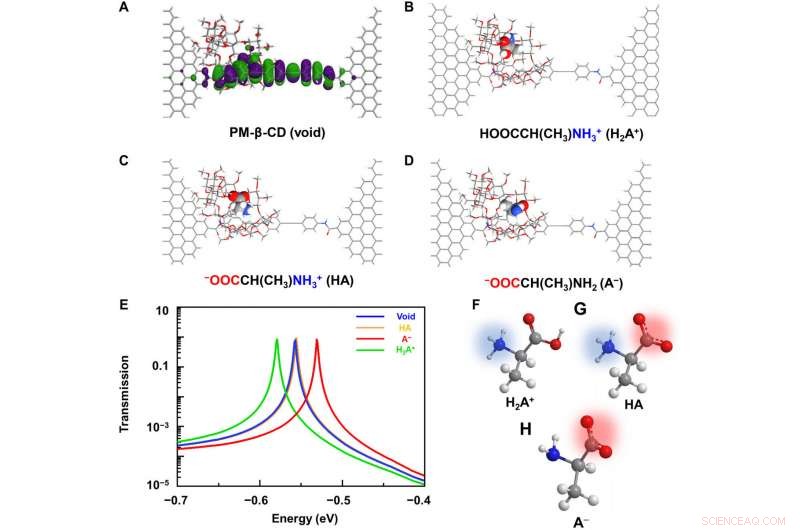
To better understand the correlation between host-guest interactions in the L-alanine based PM-β-CD single molecule junctions, Liu y col. calculated the transmission spectra of the PM-β-CD host with differently charged L-Alanine guests. Para lograr esto, they used a nonequilibrium Green's function technique based on density functional theory as implied in the Atomistix Toolkit package. They noted the conductance contribution from the perturbed highest occupied molecular orbital (p-HOMO) to be dominant at low bias voltages as reflected with transmission spectra. The transmission spectra of the configurations were significantly different near the Fermi level of electrodes to afford different conductance stages. The researchers then analyzed the transitions between each level during amino acid recognition. They used L-alanine as an example to observe reversible transitions from the dissociation to form cations, zwitterions, and anion forms. The dissociation level dominated the device conductance, and the team collected the information using a four-state model, which described the transitions to understand the capacity to detect different structures of amino acids when interacting with PM-β-CD. The amino acids had at least two association processes for the carboxylic group and the other amino group. The greater the number of states recorded, the more accurate the recognition of results were.
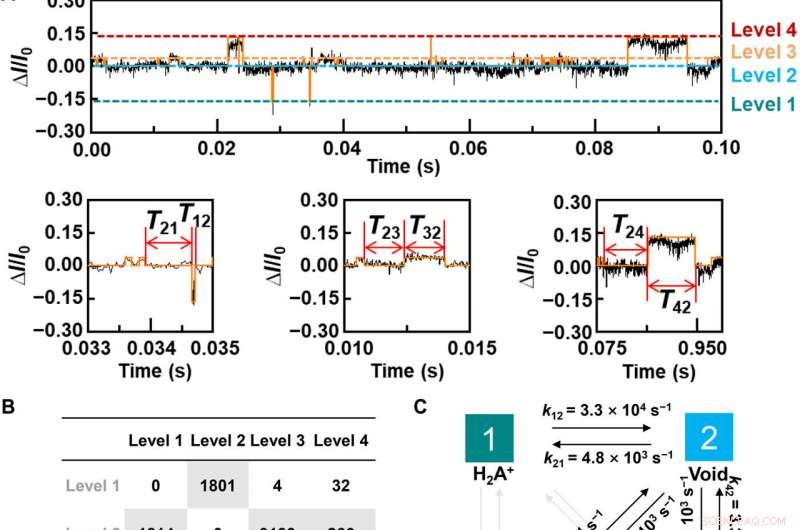
Dynamic analysis for L-Alanine detection. (A) Plot of ΔI/I0 as a function of time during the host-guest interaction. The red curve is the idealized fit by using a QuB software. pH =7, T =298 K, and the bias voltage =0.1 V. (B) Transition statistics between each state. The horizontal columns mean the initial state. (C) Kinetic model for the L-Ala recognition process. (D to I), Plots of time intervals of (D) level 1 to level 2, (E) level 2 to level 1, (F) level 2 to level 3, (G) level 3 to level 2, (H) level 2 to level 4, and (I) level 4 to level 2 at 298 K. Credit:Science Advances, doi:10.1126/sciadv.abe4365
Enantiomer recognition and outlook
Due to the similarity between enantiomers, it was more challenging to differentiate the structures compared to species identification. The associated energy differences were small and close to computational errors. To overcome these complexities, the team established a distinct "fingerprint database" for each amino acid to compare the conductance and kinetic data of different enantiomers. The team then achieved the current change and relaxation time data and compared them with the fingerprint database. Due to this universality, the method can also recognize chiral functional drug molecules to establish a broad approach for biomolecular detection at the single molecule-level.
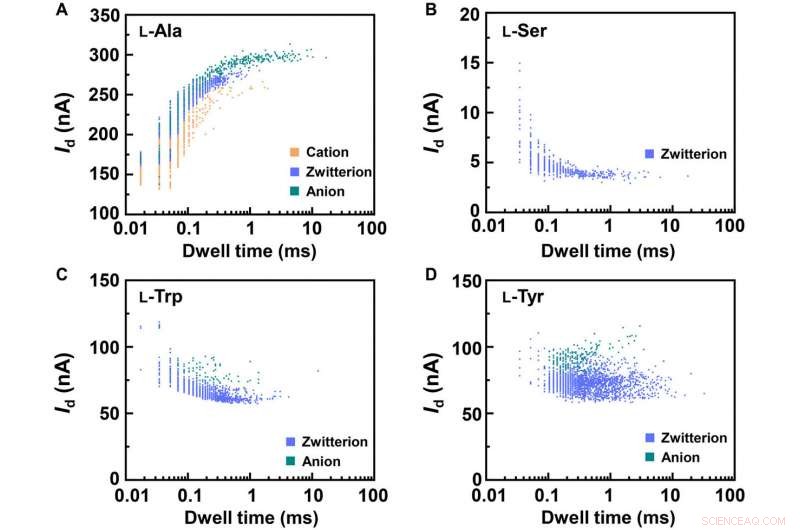
Statistical analyses of the translocation events for different amino acids. Translocation events (current as a function of the dwell time) were analyzed for (A) L-Ala, (B) L-Ser, (C) L-Trp, and (D) L-Tyr, respectivamente. The two-dimensional (2D) contour plots are composed of all the events in 10-s recording at pH =7, showing the capability of detecting the different structures of amino acids when interacting with PM-β-CD. Crédito:Science Advances, doi:10.1126/sciadv.abe4365
De este modo, Zihao Liu and colleagues presented a practical single-molecule approach for real-time electrical recognition of amino acids with different structures and chirality within microseconds. The host-guest dynamic processes could be observed for diverse amino acids at the level of the anion, zwitterion and cation shuttling. Using the permethylated-β-cyclodextrin (PM-β-CD) complex, the team recognized differently charged states of amino acids based on current fluctuation range and thermodynamic/kinetic parameters. The technique can revolutionize existing methods to accurately sequence single-molecule gene/protein sequencing toward universal applications. The method can also offer a universal tool to recognize many important molecules in environmental or biological systems to understand the basis of life at the molecular level.
© 2021 Science X Network