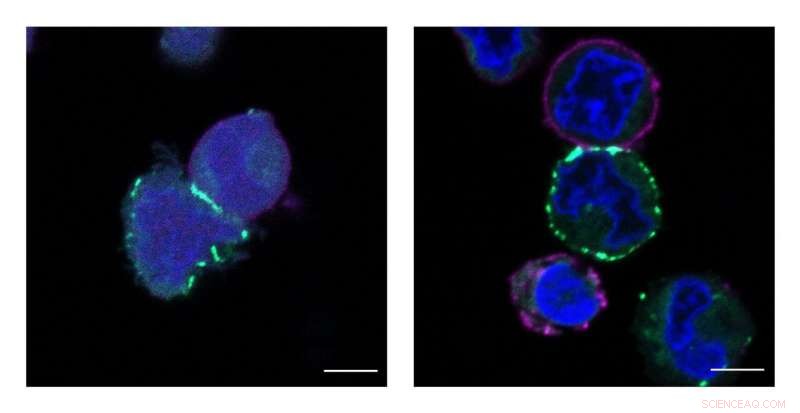
MacMillan Group de Princeton desarrolló una nueva tecnología llamada μMap que identifica a los vecinos más cercanos de las proteínas en la superficie celular. Estas imágenes muestran células etiquetadas, o resaltado en verde, por el catalizador μMap. Crédito:Tamara Reyes-Robles / Merck Exploratory Science Center
Cuando los socios corporativos de la Princeton Catalysis Initiative se sentaron hace dos años con David MacMillan, le presentaron un desafío biológico en el corazón de los posibles medicamentos contra el cáncer y otras terapias:¿qué proteínas en la superficie de una célula se tocan entre sí?
Lo que querían era análogo a un reflector que brillaba en una cueva oscura:algo que iluminara con una luz metafórica una proteína y sus vecinos más cercanos en la membrana celular. Grande, moléculas complejas, las proteínas son la materia de la vida, el mismo punto de apoyo en el que gira todo sobre nosotros:la forma en que pensamos, la forma en que crecemos, las enfermedades que contraemos. Las proteínas pueden determinar esto enviando mensajes a sus vecinos. Pero mientras que los científicos podían saber previamente quién estaba dentro de la cueva, no sabían quién estaba junto a quién, y, por lo tanto, carecían de conocimientos importantes sobre estas comunicaciones esenciales de proteína a proteína.
El Grupo MacMillan del Departamento de Química anunció en la edición actual de Ciencias que han desarrollado ese reflector.
La tecnología de vanguardia, denominado μMap por el equipo de investigadores de Princeton y científicos de Merck, utiliza un fotocatalizador, una molécula que, cuando se activa por la luz, estimula una reacción química para identificar relaciones espaciales en las superficies celulares. El catalizador genera un marcador que marca las proteínas y sus vecinos moleculares, lo que a su vez permite el mapeo preciso de su microambiente.
La tecnología podría afectar la proteómica, genómica y neurociencia, por nombrar algunos de los campos más obvios. Pero las aplicaciones de la biología fundamental son tan amplias que MacMillan, quien es el Profesor Distinguido de Química de la Universidad James S. McDonnell de Princeton, tiene hambre de poner la tecnología "en manos de todos" para ver lo que los científicos de otros campos pueden hacer.
"Para las tecnologías que tenemos ahora, el problema no es si puedes etiquetar cosas, ", dijo." El problema es que puedes etiquetar miles de cosas y, por lo tanto, no puedes saber qué hay allí y qué está justo al lado. Eso resulta ser realmente realmente importante porque las moléculas, proteínas o enzimas que se envían señales entre sí suelen estar al lado de la otra. Bien, el estado de la técnica no te dice lo que está cerca ".
Entonces se les ocurrió un enfoque radicalmente nuevo.
"Hicimos algunos experimentos críticos e inmediatamente pudimos demostrar que estábamos etiquetando cosas a una distancia realmente corta, ", Dijo MacMillan." Ahora sabemos exactamente lo que hay en el vecindario. Y eso nunca se había hecho antes. Para biología, será como encender el interruptor de la luz y ver todo de repente ".
Los científicos del Centro de Ciencias Exploratorias de Merck (MESC), Rob Oslund y Olugbeminiyi Fadeyi, coautores de artículos que tienen su sede en Cambridge, Massachusetts, dijo que la tecnología podría inspirar grandes desarrollos nuevos en biología. "Dado el importante papel de comprender las interacciones de las proteínas dentro de los microambientes celulares, "Oslund dijo, "esta tecnología tiene el potencial de ser una herramienta revolucionaria para los laboratorios de ciencias biológicas tanto académicos como industriales de todo el mundo".
El μMap, micro-mapa pronunciado, identifica vecinos dentro de un radio de 1 a 10 nanómetros alrededor de una proteína en particular. (Para referencia, un cabello humano tiene alrededor de 100, 000 nanómetros de ancho.) La resolución en este nivel identifica las 10 o 15 moléculas más cercanas.
Jacob Geri, becario de investigación postdoctoral en el Merck Center for Catalysis de la Universidad de Princeton y co-primer autor de la Ciencias artículo con el estudiante de posgrado James Oakley y la científica del MESC Tamara Reyes-Robles, dicho μMap hace esto usando luz azul para impulsar una reacción catalítica.
Funciona de la siguiente manera:el catalizador; en este caso, un compuesto de metal orgánico:se une selectivamente a cualquiera de unos 40, 000 proteínas en la superficie de una célula, donde actúa como una especie de antena. Luz azul, que tiene una energía fotónica muy alta, sirve como disparador. Cuando brilló en la celda, esta luz azul es captada por la antena, que convierte su energía fotónica en energía química. Esa energía latente no se enfría; no se difunde; no vaga sin rumbo fijo por la membrana celular pintando todo lo que encuentra. Simplemente se sienta.
Basado en un artículo publicado hace unos 40 años, Al grupo de MacMillan se le ocurrió la idea de emplear el uso de una molécula orgánica llamada diazirina que es particularmente receptiva a esta energía latente. Cuando una diazirina se mueve muy cerca del catalizador, dentro de 0,1 nanómetros, la energía química se transfiere a la diazirina. La diazirina, a su vez, reacciona de forma tan violenta que libera un subproducto y se convierte en lo que se conoce como carbeno. una especie "enojada" que se adhiere a las proteínas vecinas.
"El catalizador transfiere tanta energía que la molécula se desgarra para exponer un átomo de carbono increíblemente inestable, que luego se adherirá a todo lo que pueda, "explicó Geri.
El catalizador puede realizar esta reacción química muchas veces, por lo que el proceso se repite para todas las moléculas localizadas, proteínas y enzimas. Debido a que los carbenos tienen una vida tan corta (solo un par de nanosegundos), su reacción proporciona una vívida instantánea en tiempo real de todas las moléculas contiguas. Después, los investigadores pueden armar un mapa preciso del microambiente, la misma tecnología que los científicos estaban buscando.
"Gran parte del mecanismo de la enfermedad tiene lugar a través de la forma en que estas células se comunican entre sí, y solo pueden hablar si se tocan, " said Geri. "That's why the surface of the cell is so important. If they touch, they can communicate."
He added:"We can now figure out what's making that communication happen or what's making that communication change. It's really been an amazing experience, working on this."
MacMillan's group chose two categories of human cells to investigate. One was a class of proteins that had known interactions, selected as a kind of control group to prove that their interactions could be captured by μMap. The second group was "more interesting, " said Geri. It centered on proteins called PD-L1 and PD-1, which are associated with the body's immune system and its response to cancer cells.
Normalmente, sick cells like cancer cells would present as molecular interlopers that need to be cleared by the immune system. But cancer cells are deceptive, said MacMillan. They send out a "don't kill me" signal through a cloaking mechanism involving the PD-L1 and PD-1 axis. Since cancer therapies are successful partly based on their capacity to block that signal, scientists want to know more about how it is transmitted. Mapping the precise neighborhood is an essential early step. When researchers put the μMap catalyst on PD-L1 and PD-1, the molecules in their micro-environment are tagged. Protein-protein interactions that had previously been hypothesized could now be directly observed. And several correlations were detected that had never been conceived of.
"Ahora, we don't do the cancer biology, " said MacMillan. "But we've invented this tool that can give you a lot of information about these cancer cells. We think that by using this information, you can start to target those proteins as a way to also remove interfering signals. And if you can remove those signals, you make your immune system better at going after these cancer cells."
Soon after MacMillan arrived at Princeton, he began driving research in harnessing blue LED light to perform previously impossible feats of chemistry. Merck became involved in 2006, with a seed donation toward MacMillan's research. The company has since donated additional monies, and in 2019 they announced a 10-year funding commitment towards the Princeton Catalysis Initiative, which fosters interdisciplinary collaborations to accelerate the discovery of new research areas.
"Our collaboration created a novel cellular chemistry approach leveraging photoredox catalysis to activate diazirines, an important class of organic molecules, in a temporal-controlled manner, " said Merck's Fadeyi. "Because of the routine use of diazirines within chemical biology and biology, this method will be in high demand not only for protein labeling, but for identifying the binding targets of other biomolecules to elucidate their functional roles."
He added:"The collaboration was successful due to the close interactions between Merck scientists and Dave's lab."
MacMillan likewise lauded the discovery as proof of the value of collaboration across academic and industry lines, like those envisioned when the Princeton Catalysis Initiative first came into being in 2018.
"As chemists, we don't know any good questions in biology—zero, ", dijo." Entonces, you're taking these people who know everything about biology, and they have this problem that they're trying to solve. And it's ultimately a great problem for a chemistry group. Al mismo tiempo, it's not a problem that a chemistry group would ever think about because they don't know biology. You have these two different areas and you put them together and you start to realize there all these great things you can do.
"This is what I love about the social science of science, " he added. "It is absolutely a beautiful example of how it took a village to solve a problem."