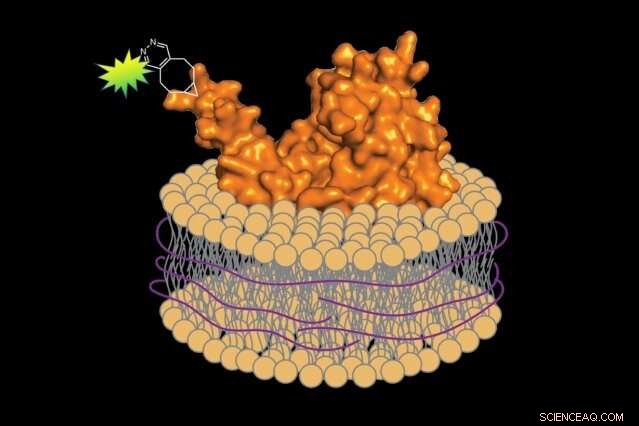
Usando un método sin detergente, Los biólogos pueden preparar proteínas marcadas con fluorescencia junto con un pequeño segmento de la membrana celular asociada. preservando el ambiente nativo de la proteína. Crédito:Jean-Marie Swiecicki
Todas las células tienen una membrana lipídica que rodea sus componentes internos, formando una barrera protectora para controlar lo que entra y lo que queda fuera. Las proteínas incrustadas en estas membranas son esenciales para la vida; ayudan a facilitar el transporte de nutrientes, conversión y almacenamiento de energía, y comunicación celular. También son importantes en las enfermedades humanas, y representan alrededor del 60 por ciento de los objetivos farmacológicos aprobados. Para estudiar estas proteínas de membrana fuera de la complejidad de la célula, los investigadores deben usar detergente para quitar la membrana y extraerlos. Sin embargo, Determinar el mejor detergente para cada proteína puede implicar un extenso ensayo y error. Y, la eliminación de una proteína de su entorno natural corre el riesgo de desestabilizar la estructura plegada y alterar la función.
En un estudio publicado el 9 de diciembre en Biología química celular , Los científicos del MIT idearon una forma rápida y generalizable de extraer, purificar, y etiquetar las proteínas de la membrana para obtener imágenes sin ningún tipo de detergente, llevando una parte de la membrana circundante para proteger la proteína y simular su entorno natural. Su enfoque combina técnicas químicas y bioquímicas bien establecidas de una manera nueva, aislar eficientemente la proteína para que pueda etiquetarse con fluorescencia y examinarse bajo un microscopio.
"Siempre bromeo diciendo que no es muy realista estudiar las proteínas en el jabón, "dice la autora principal Barbara Imperiali, profesor de biología y química. "Hemos creado un flujo de trabajo que permite obtener imágenes de las proteínas de membrana mientras se mantienen sus identidades e interacciones nativas. Con suerte, ahora menos personas evitarán estudiar las proteínas de membrana, dada su importancia en muchos procesos fisiológicos ".
Como miembro del laboratorio Imperiali, El ex postdoctorado y autor principal Jean-Marie Swiecicki investigó las proteínas de la membrana del patógeno Campylobacter jejuni transmitido por los alimentos. En este estudio, Swiecicki se centró en PglC y PglA, dos proteínas de membrana que juegan un papel en permitir que las bacterias infecten las células humanas. Sus experimentos requirieron etiquetar PglC y PglA con etiquetas fluorescentes para rastrearlos. Sin embargo, no estaba satisfecho con los métodos existentes para hacerlo.
En algunos casos, las etiquetas fluorescentes que deben incorporarse a la proteína para visualizarla son demasiado grandes para colocarlas en posiciones definidas. En otros casos, estas etiquetas no brillan lo suficiente, o interferir con la estructura y función de la proteína.
Para evitar tales problemas, Swiecicki decidió utilizar un método conocido como "mutagénesis de aminoácidos no naturales". Los aminoácidos son las unidades que componen la proteína, y la mutagénesis de aminoácidos no naturales implica la adición de un nuevo aminoácido que contiene un grupo químico diseñado dentro de la secuencia de la proteína. Este grupo químico se puede etiquetar con una etiqueta que brilla intensamente.
Swiecicki insertó el código genético de las proteínas de la membrana de C. jejuni en una bacteria diferente, Escherichia coli. Dentro de E. coli, podría incorporar el aminoácido no natural, que podría modificarse químicamente para agregar la etiqueta fluorescente.
Cuando llegó el momento de eliminar las proteínas de la membrana, Sustituyó el detergente por una sustancia diferente:un polímero de estireno-ácido maleico (SMA). A diferencia del detergente, SMA envuelve la proteína extraída y un pequeño segmento de la membrana asociada en una capa protectora, preservando su entorno nativo. Imperiali explica, "Es como una bufanda que protege tu cuello del frío".
Swiecicki pudo luego monitorear las proteínas brillantes bajo un microscopio para verificar que su técnica fuera lo suficientemente selectiva como para aislar proteínas de membrana individuales. Todo el proceso, él dice, toma solo unos días, y generalmente es mucho más rápido y confiable que los métodos de extracción a base de detergente, lo que puede llevar meses y requiere la experiencia de bioquímicos altamente capacitados para optimizar.
"No diría que es una fórmula mágica que va a funcionar para cada proteína, ", dice." Pero es una herramienta muy eficiente que podría facilitar el estudio de muchos tipos diferentes de proteínas de membrana ". él dice, incluso puede ayudar a facilitar las pruebas de detección de drogas de alto rendimiento.
"Como alguien que trabaja con complejos de proteínas de membrana, Puedo dar fe de la gran necesidad de mejores métodos para estudiarlos, "dice Suzanne Walker, profesor de microbiología en la Facultad de Medicina de Harvard que no participó en el estudio. Espera extender el enfoque descrito en el documento a los complejos de proteínas que investiga en su propio laboratorio. "Aprecié la gran cantidad de detalles incluidos en el texto sobre cómo aplicar la estrategia con éxito, " ella agrega.
Los próximos pasos serán probar la técnica en proteínas de mamíferos, y aislar múltiples proteínas a la vez en la capa de SMA para observar sus interacciones. Y, por supuesto, cada nueva técnica merece un nombre. "Todavía estamos trabajando en un acrónimo pegadizo, "Imperiali dice." ¿Alguna idea? "
Esta historia se vuelve a publicar por cortesía de MIT News (web.mit.edu/newsoffice/), un sitio popular que cubre noticias sobre la investigación del MIT, innovación y docencia.