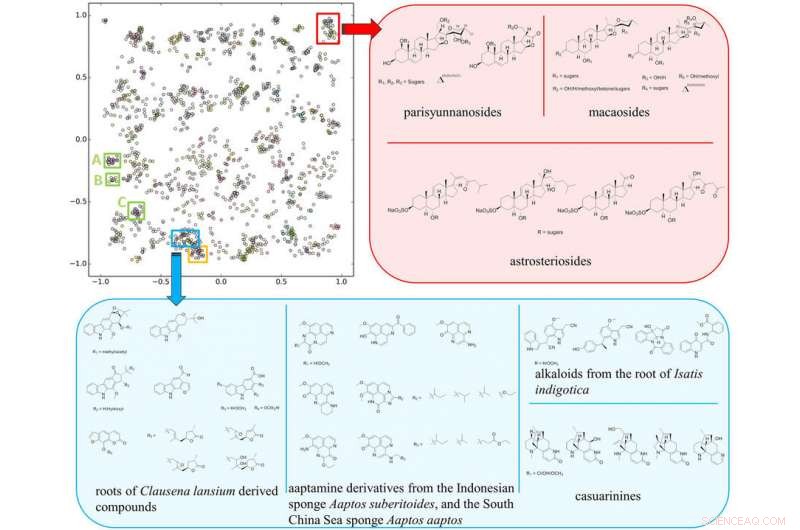
El mapa de clústeres SMART basado en el resultado de entrenamiento de 2, 054 espectros HSQC sobre 83, 000 iteraciones, con recuadros insertados que representan diferentes clases compuestas discutidas en el texto. Crédito:Universidad de California - San Diego
Un equipo interdisciplinario de investigadores de la Universidad de California en San Diego ha desarrollado un método para identificar las estructuras moleculares de productos naturales que es significativamente más rápido y más preciso que los métodos existentes. El método funciona como el reconocimiento facial de estructuras moleculares:utiliza un dato espectral único para cada molécula y luego lo ejecuta a través de una red neuronal de aprendizaje profundo para colocar la molécula desconocida en un grupo de moléculas con estructuras similares.
El nuevo sistema se llama "INTELIGENTE, "que significa tecnología de reconocimiento preciso de moléculas pequeñas, y tiene el potencial de acelerar diez veces el proceso de identificación de la estructura molecular. Este desarrollo podría representar un cambio de paradigma en el análisis químico, campos farmacéuticos y de descubrimiento de medicamentos, ya que el 70 por ciento de todos los medicamentos aprobados por la Administración de Alimentos y Medicamentos (FDA) se basan en productos naturales como microorganismos del suelo, plantas terrestres y, cada vez más, formas de vida marina como las algas.
"La estructura de una molécula es la información habilitadora, "dijo Bill Gerwick, profesor de oceanografía y ciencias farmacéuticas en la Institución de Oceanografía Scripps de UC San Diego. "Tienes que tener la estructura para cualquier aprobación de la FDA. Si quieres tener propiedad intelectual, tienes que patentar esa estructura. Si quieres hacer análogos de esa molécula, necesitas saber cuál es la molécula inicial. Es una información fundamental ".
Chen Zhang, un doctorado en nanoingeniería estudiante de UC San Diego colaborando con Gerwick y el primer autor del artículo publicado en Informes científicos de la naturaleza , dijo que determinar la estructura de una molécula puede ser un cuello de botella en el proceso de investigación de productos naturales, los expertos tardan meses e incluso años en determinar con precisión la estructura correcta y completa. Si bien cada molécula y su línea de tiempo de identificación son diferentes, el enfoque SMART brinda a los investigadores una pista temprana sobre a qué familia pertenece una nueva molécula, reduciendo drásticamente el tiempo necesario para caracterizar un nuevo producto natural.
"La forma en que pudimos acelerar el proceso es esencialmente mediante el uso de software de reconocimiento facial para observar la información clave que obtenemos sobre las moléculas, Gerwick dijo. La pieza clave de información que utiliza el equipo se llama resonancia magnética nuclear de coherencia cuántica singular heteronuclear, o RMN HSQC, espectro. Produce un mapa topológico de puntos que revela qué protones de la molécula están unidos directamente a qué átomos de carbono, una disposición única para cada molécula.
Zhang y Gerwick se unieron a Gary Cottrell, profesor de ciencias de la computación e ingeniería en la Escuela de Ingeniería Jacobs de UC San Diego, para desarrollar un sistema de aprendizaje profundo entrenado con miles de espectros HSQC extraídos de investigaciones anteriores. Esta red neuronal convolucional toma una imagen 2-D del espectro de RMN HSQC de una molécula desconocida y la mapea en un espacio de 10 dimensiones agrupadas cerca de moléculas similares, facilitando a los investigadores dilucidar la estructura de una molécula desconocida.
"Chen adoptó este enfoque para obtener espectros de RMN de más de 4, 000 compuestos de la literatura al recortar literalmente las imágenes de los PDF de los artículos, "Dijo Cottrell." ¡Fue un esfuerzo increíble! Aún así, normalmente, estos datos no son suficientes para entrenar una red profunda, pero usamos una tecnología llamada red siamesa, en el que entrenas sobre pares de imágenes. Esto amplifica su conjunto de entrenamiento aproximadamente al cuadrado del número de compuestos en una familia, y es lo que hizo factible este proyecto ".
Esta colaboración es la primera vez que Gerwick ha sido mentor de un estudiante de ingeniería, y el intercambio de ideas resultó fructífero.
"Ha sido una interacción maravillosa. UC San Diego tiene algo realmente mágico al respecto, y esa es la profundidad de la colaboración que se produce entre los departamentos:es fenomenal, ", Dijo Gerwick." Cuando intentas tomar cuidadosamente de otra disciplina algo que tal vez sea un lugar común en esa disciplina y lo aplicas de una manera nueva y única en nuestra disciplina, es una oportunidad para tener realmente este tipo de cambio de paradigma. Y creo que esta tecnología con algún avance, podría ser un cambio de paradigma real en la forma en que hacemos todo tipo de química y análisis químico ".