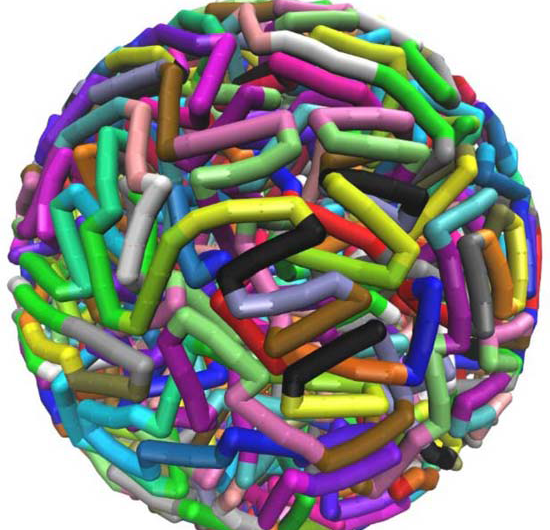
Específico de secuencia, inducido por torsión, configuraciones elásticas retorcidas, generado por simulaciones de dinámica molecular en supercomputadoras en el Centro de Computación Avanzada de Texas, ayudar a explicar cuánto tiempo caben las hebras de ADN en espacios pequeños. Crédito:Christopher G. Myers, B. Montgomery Pettitt, Rama médica de la Universidad de Texas
Un misterio biológico se encuentra en el centro de cada una de nuestras células, a saber:cómo se puede enrollar un metro de ADN en el espacio de una micra (o una millonésima parte de un metro) dentro de cada núcleo de nuestro cuerpo.
Los núcleos de las células humanas ni siquiera son el lugar biológico más poblado que conocemos. Algunos bactiófagos, virus que infectan y se replican dentro de una bacteria, tienen ADN aún más concentrado.
"¿Cómo entra ahí?" B. Montgomery (Monte) Pettitt, bioquímico y profesor de la rama médica de la Universidad de Texas, pregunta. "Es un polímero cargado. ¿Cómo supera la repulsión en su densidad cristalina líquida? ¿Cuánto orden y desorden se permite? y ¿cómo juega esto un papel en los ácidos nucleicos? "
Usando las supercomputadoras Stampede y Lonestar5 en el Texas Advanced Computing Center (TACC) de la Universidad de Texas en Austin, Pettitt investiga cómo el ADN de los fagos se pliega en espacios hiperconfinados.
Escribiendo en la edición de junio de 2017 de la Revista de química computacional , explicó cómo el ADN puede superar tanto la repulsión electrostática como su rigidez natural.
¿La clave para hacerlo? Torceduras.
La introducción de giros o curvas pronunciadas en configuraciones de ADN empaquetado dentro de una envoltura esférica reduce significativamente las energías y presiones generales de la molécula. según Pettitt.
Él y sus colaboradores utilizaron un modelo que deforma y retuerce el ADN cada 24 pares de bases, que está cerca de la longitud promedio que se predice a partir de la secuencia de ADN del fago. La introducción de tales defectos persistentes no solo reduce la energía de flexión total del ADN confinado, pero también reduce el componente electrostático de la energía y la presión.
"Demostramos que un amplio conjunto de configuraciones de polímeros es consistente con los datos estructurales, "él y su colaborador Christopher Myers, también de la Rama Médica de la Universidad de Texas, escribió.
Información como esta no se puede obtener estrictamente en el laboratorio. Requieren supercomputadoras que sirvan como microscopios moleculares, trazar el movimiento de átomos y enlaces atómicos en escalas de tiempo y longitud que no son factibles de estudiar con experimentos físicos solamente.
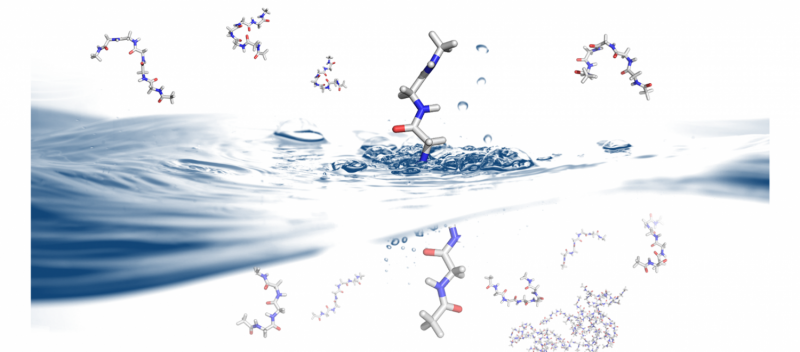
Cómo y por qué las proteínas se pliegan es un problema que tiene implicaciones para el diseño y la terapéutica de las proteínas. B. Montgomery Pettitt y su grupo de investigación en la Rama Médica de la Universidad de Texas utilizan las supercomputadoras Stampede y Lonestar5 en el Centro de Computación Avanzada de Texas para explorar la dinámica del plegamiento de proteínas en solución. Crédito:Christopher G. Myers, B. Montgomery Pettitt, Rama médica de la Universidad de Texas
"En el campo de la biología molecular, hay una maravillosa interacción entre la teoría, experimento y simulación, ", Dijo Pettitt." Tomamos los parámetros de los experimentos y vemos si están de acuerdo con las simulaciones y las teorías. Este se convierte en el método científico de cómo ahora avanzamos nuestras hipótesis ".
Problemas como los que le interesan a Pettitt no se pueden resolver en una computadora de escritorio o en un grupo de campus típico, pero requieren cientos de procesadores de computadora trabajando en paralelo para imitar los movimientos diminutos y las fuerzas físicas de las moléculas en una célula.
Pettitt puede acceder a las supercomputadoras de TACC en parte gracias a un programa único conocido como Revista de química computacional iniciativa, que hace que los recursos informáticos de TACC, experiencia y capacitación disponibles para investigadores dentro de las 14 instituciones de University of Texas Systems.
"Investigación computacional, como el del Dr. Pettitt, que busca unir nuestra comprensión de lo físico, químico, y finalmente fenómenos biológicos, implica tantos cálculos que solo es realmente accesible en grandes supercomputadoras como los sistemas Stampede o Lonestar5 de TACC, "dijo Brian Beck, investigador de ciencias de la vida en TACC.
"Tener recursos de supercomputación TACC disponibles es fundamental para este estilo de investigación, Pettitt dijo.
ENCONTRAR EL ORDEN DE LAS PROTEÍNAS ALTERADAS
Otro fenómeno que siempre ha interesado a Pettitt es el comportamiento de las proteínas intrínsecamente desordenadas (IDP) y los dominios intrínsecamente desordenados. donde partes de una proteína tienen una forma desordenada.
A diferencia de los cristales o del ADN altamente empaquetado de los virus, que tienen distintos, formas rígidas, Los desplazados internos "se convierten en un lío pegajoso, "según Pettitt. Y, sin embargo, son fundamentales para todas las formas de vida.
Se cree que en eucariotas (organismos cuyas células tienen subestructuras complejas como núcleos), aproximadamente el 30 por ciento de las proteínas tienen un dominio intrínsecamente desordenado. Más del 60 por ciento de las proteínas involucradas en la señalización celular (procesos moleculares que toman señales desde el exterior de la célula o a través de las células que le dicen a la célula qué comportamientos activar y desactivar en respuesta) tienen dominios desordenados. Similar, El 80 por ciento de las proteínas de señalización relacionadas con el cáncer tienen regiones IDP, lo que las convierte en moléculas importantes para comprender.
Entre los desplazados internos que Pettitt y su grupo están estudiando se encuentran los factores de transcripción nuclear. Estas moléculas controlan la expresión de genes y tienen un dominio de señalización rico en aminoácidos flexibles, glicina.

Las imágenes de arriba muestran las distribuciones de densidad promedio sobre 21 configuraciones de ADN, cada una simulada por 100 nanosegundos de dinámica molecular después de la minimización usando a) configuraciones completamente elásticas yb) retorcidas, para comparación con c) Mapa de densidad Cryo-EM a partir de reconstrucciones de fagos asimétricos de P22 con la densidad de la cápside eliminada gráficamente. Crédito:Christopher G. Myers, B. Montgomery Pettitt, Rama médica de la Universidad de Texas
El plegamiento del dominio de señalización del factor de transcripción nuclear no se debe a los enlaces de hidrógeno ni a los efectos hidrófobos, como la mayoría de las moléculas de proteínas, según Pettitt. Bastante, cuando las moléculas más largas encuentran demasiadas glicinas en un espacio, van más allá de su solubilidad y comienzan a asociarse entre sí de formas inusuales.
"Es como agregar demasiada azúcar al té, "Pettitt explica." No se volverá más dulce. El azúcar debe desprenderse de la solución y encontrar un compañero, precipitándose en un bulto ".
Escribiendo en Ciencia de las Proteínas en 2015, describió simulaciones moleculares realizadas en Stampede que ayudaron a explicar cómo y por qué los desplazados internos colapsan en estructuras parecidas a glóbulos.
Las simulaciones calcularon las fuerzas de las interacciones dipolo-dipolo carbonilo (CO):atracciones entre el extremo positivo de una molécula polar y el extremo negativo de otra molécula polar. Determinó que estas interacciones son más importantes en el colapso y la agregación de cadenas largas de glicina que en la formación de enlaces H.
"Dado que la columna vertebral es una característica de todas las proteínas, Las interacciones de CO también pueden desempeñar un papel en proteínas de secuencia no trivial donde la estructura finalmente está determinada por el empaquetamiento interior y los efectos estabilizadores de los enlaces de H y las interacciones de CO-CO. " Él concluyó.
La investigación fue posible gracias a una asignación de tiempo de cómputo en Stampede a través del Extreme Science and Engineering Discovery Environment (XSEDE), que cuenta con el apoyo de la National Science Foundation.
Pettitt, un campeón de la supercomputación desde hace mucho tiempo, no solo usa los recursos de TACC él mismo. Anima a otros eruditos, incluidos sus colegas del Centro Sealy de Biología Estructural y Biofísica Molecular, utilizar supercomputadoras también.
"La informática avanzada es importante para el análisis y el refinamiento de datos de los experimentos, Microscopía electrónica y de rayos X, e informática, ", dice." Todos estos problemas tienen problemas de procesamiento de big data que se pueden abordar mediante la informática avanzada ".
Cuando se trata de descubrir los misterios de la biología en las escalas más pequeñas, nada supera a una supercomputadora gigante.