El género Musa, que abarca aproximadamente 70 especies herbáceas, se encuentra predominantemente en las regiones tropicales y subtropicales de Asia y Oceanía. Este género es conocido por ser uno de los cultivos alimentarios más importantes a nivel mundial y por ser una planta ornamental popular en los mercados.
A pesar de las importantes contribuciones realizadas por las tecnologías de secuenciación de tercera generación en la producción de genomas de alta calidad, sigue existiendo una escasez de recursos genómicos para los cultivares de banano, sus parientes silvestres y las especies ornamentales dentro de Musa, lo que obstaculizó la mejora tanto de los cultivos como de los ornamentales. variedades.
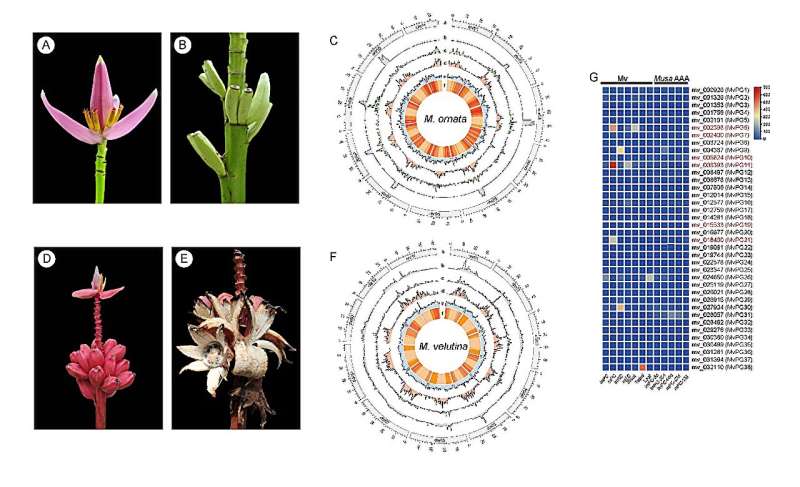
Musa ornata W. Roxburgh (Mo) y Musa velutina H. Wendl. &Drude (Mv) pertenecen a la sección Musa de la familia Musaceae. Están estrechamente relacionados con Musa acuminata y son originarios de Bangladesh, Myanmar y el noreste de la India. Estas especies no sólo se cultivan ampliamente como plantas ornamentales populares en las regiones tropicales, sino que también contribuyen a la dieta local a través de sus frutos.
Por lo tanto, Mo y Mv son candidatos deseables para la secuenciación del genoma de alta calidad para mejorar el futuro mejoramiento molecular. En este estudio, los conjuntos de genomas a nivel cromosómico de Mo y Mv se generaron utilizando lecturas largas de Oxford Nanopore y lecturas de Hi-C. Los genomas de Mo y Mv se ensamblaron en 11 pseudocromosomas con tamaños de genoma de 427,85 Mb y 478,10 Mb, respectivamente.
Las secuencias repetitivas comprendieron el 46,70% y el 50,91% del total de los genomas de Mo y Mv, respectivamente. Las evaluaciones de calidad del genoma confirmaron la contigüidad (LAI:13,68 y 16,81), la precisión (tasas de mapeo de lecturas de Illumina:95,55 % y 94,29 %) y la integridad (BUSCO:98,08 % y 98,51 %) de los dos genomas.
Según las predicciones genéticas, se anotaron un total de 39.177 y 31.256 genes codificadores de proteínas de alta confianza para Mo y Mv, respectivamente. En comparación con Musa acuminata y Mv, se observaron varias inversiones y translocaciones grandes en chr04 de Mo. Los análisis de enriquecimiento de genes expresados diferencialmente (DEG) y de ontología genética (GO) indicaron que los genes regulados positivamente en los pericarpios maduros de Mv se asociaron principalmente con el metabolismo del sacárido. procesos, particularmente en la pared celular y la región extracelular.
Además, se identificaron varios genes de poligalacturonasa (PG) que mostraron niveles de expresión más altos en los pericarpios maduros de Mv en comparación con otros tejidos, lo que puede ser responsable de la dehiscencia del pericarpio. Además, este estudio también identificó genes asociados con la vía de biosíntesis de antocianinas.
En conjunto, los conjuntos de genomas a nivel cromosómico de Mo y Mv brindan información valiosa sobre el mecanismo de la dehiscencia del pericarpio y la biosíntesis de antocianinas en el banano, lo que contribuirá significativamente a futuros esfuerzos de mejoramiento genético y molecular.
El artículo "Los conjuntos de genomas a nivel cromosómico de Musa ornata y Musa velutina proporcionan información sobre la dehiscencia del pericarpio y la biosíntesis de antocianinas en el banano" se ha publicado en Horticulture Research. .
Más información: Tian-Wen Xiao et al, Los conjuntos de genomas a nivel cromosómico de Musa ornata y M. velutina proporcionan información sobre la dehiscencia del pericarpio y la biosíntesis de antocianinas en el plátano, Investigación en horticultura (2024). DOI:10.1093/hora/uhae079
Información de la revista: Investigación en horticultura
Proporcionado por la Universidad Agrícola de NanJing